Учебный сайт
Бредихина Данилы
- VII
- Восстановление окружения молекулы белка в кристалле
3HDD
Анализ внутрикристаллических контактов
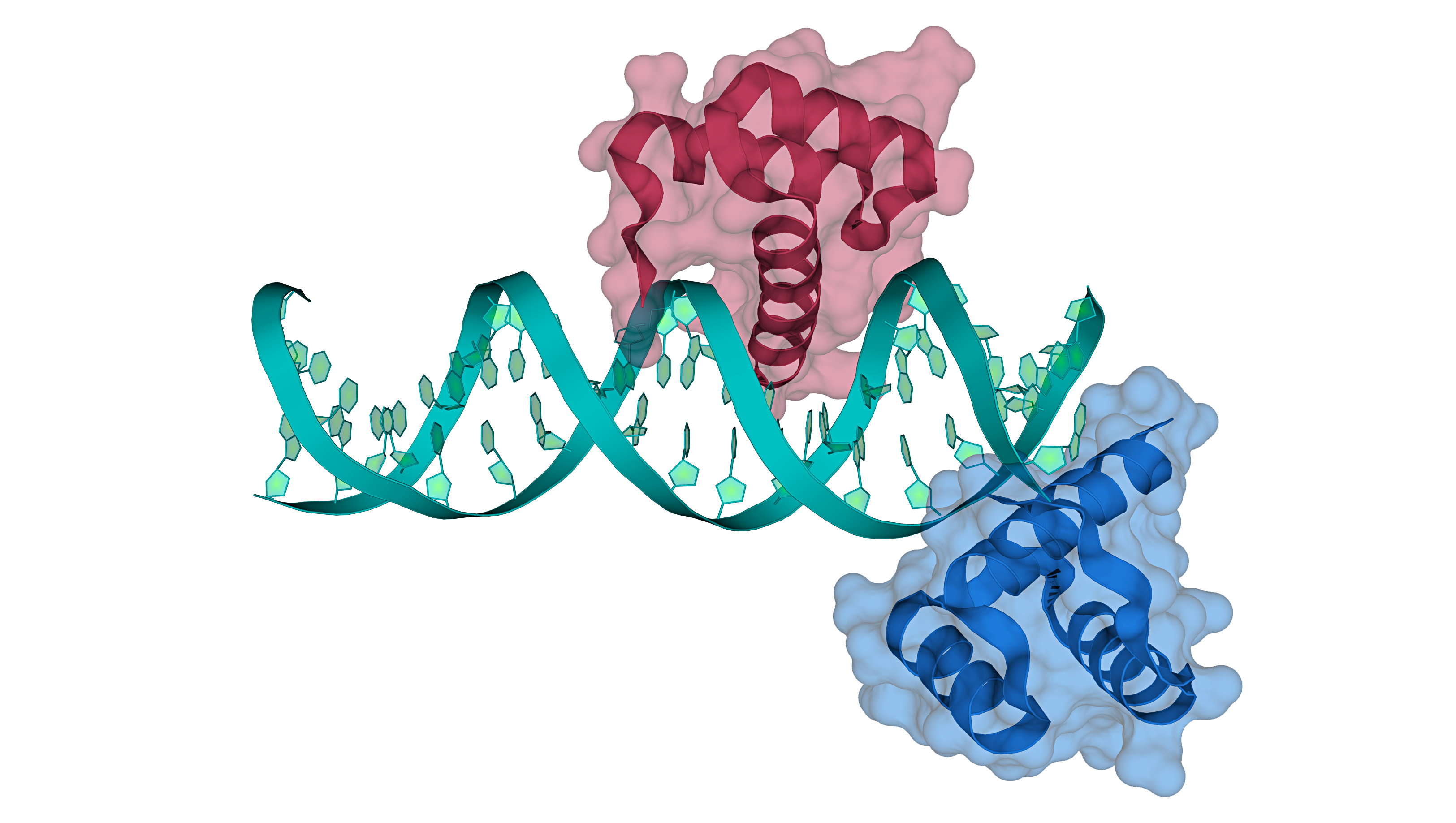
Модель 3HDD представляет структуру комплекса гомеодомена engrailed Drosophila melanogaster и ДНК с разрешением 2.20 Å. (Гомеодомен – укладка ДНК- и РНК-связывающих белков, распространённая среди факторов транскрипции, коим и является продукт гена engrailed.) С помощью PyMol было построено изображение структуры, на котором белковые цепи A и B отмечены малиновым и синим цветами, соответственно, а ДНК – бирюзовым:
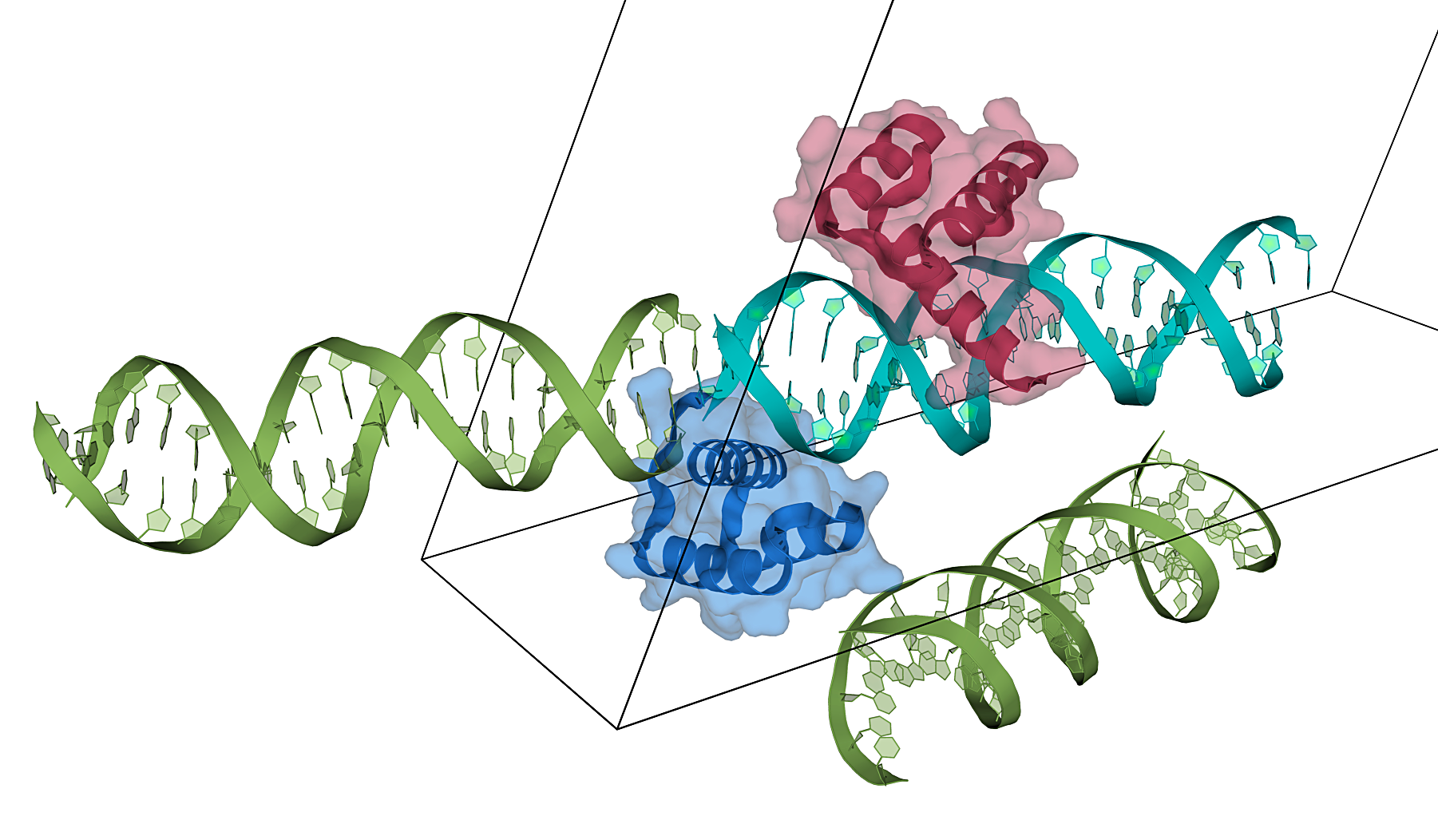
Для построения соседних асимметрических единиц в PyMol применялась команда symexp. Эта команда позволяет получить изображение молекулы ДНК из соседней ячейки, которая контактирует с цепью B белка. На изображении ниже можно увидеть границу ячейки, а также ДНК из соседних ячеек (они обозначены зелёным цветом).
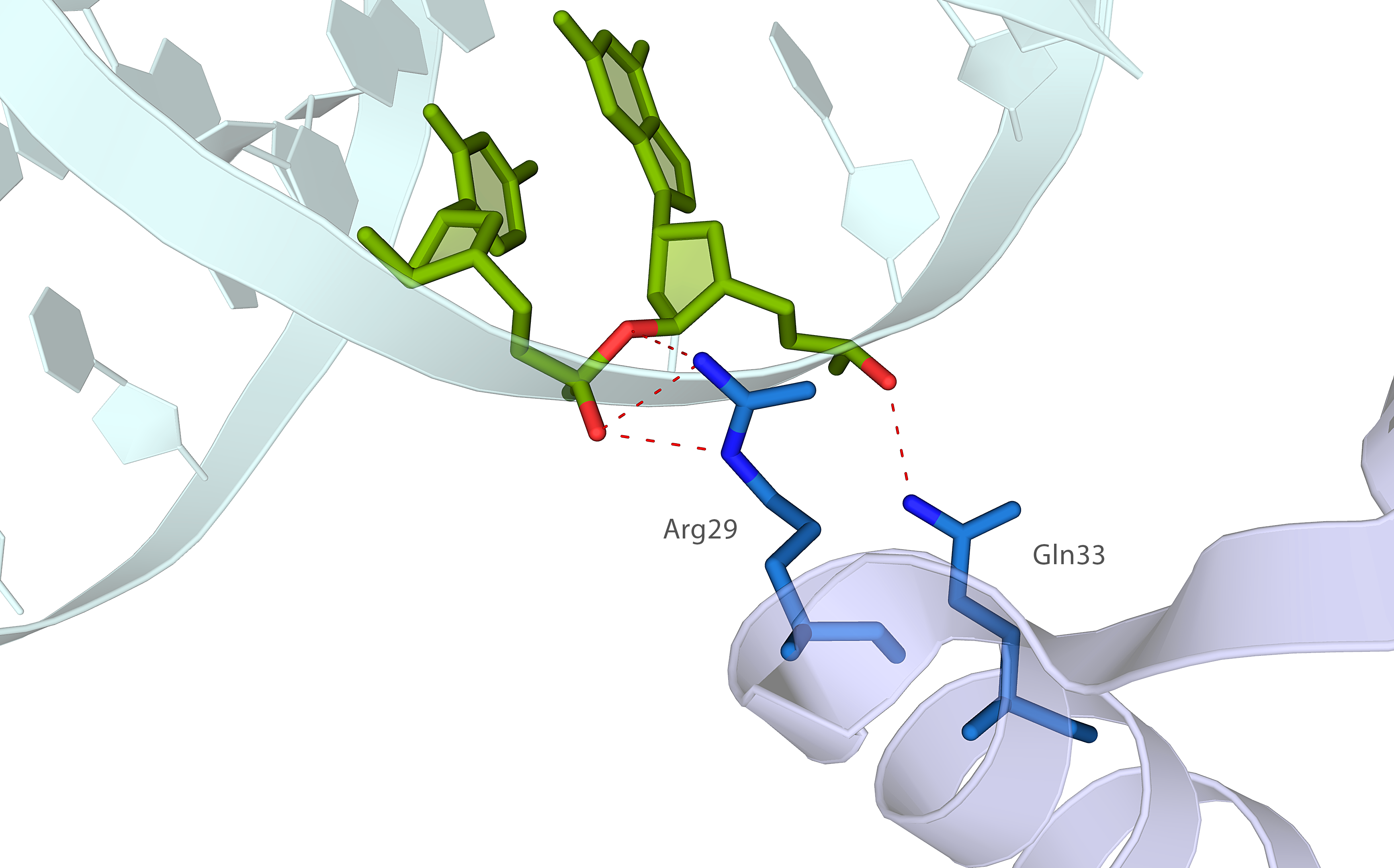
Для отображения водородных связей между ДНК соседней ячейки и цепью B белка в PyMol были использованы, в частности, команды:
symexp symd, dna, chain B, 4, 1 # создать объекты с префиксом symd # для объекта dna на расстоянии 3 Å от цепи B, # присваивая каждому новому объекту уникальный идентификатор (segi = 1) select Bres, byres (chain B within 3.5 of symd*) select DNAres, byres (symd* within 3.5 of chainB) distance hbonds, Bres, DNAres, 3.5, 0, 2
На изображениях ниже показаны цепь B белка, молекулы ДНК соседних ячеек, а также водородные связи между ними (фрагменты белка и ДНК, расположенные на расстоянии не более 3,5 Å, показаны синим и зелёным цветами, соответственно; водородные связи, предсказанные PyMol, обозначены красным):
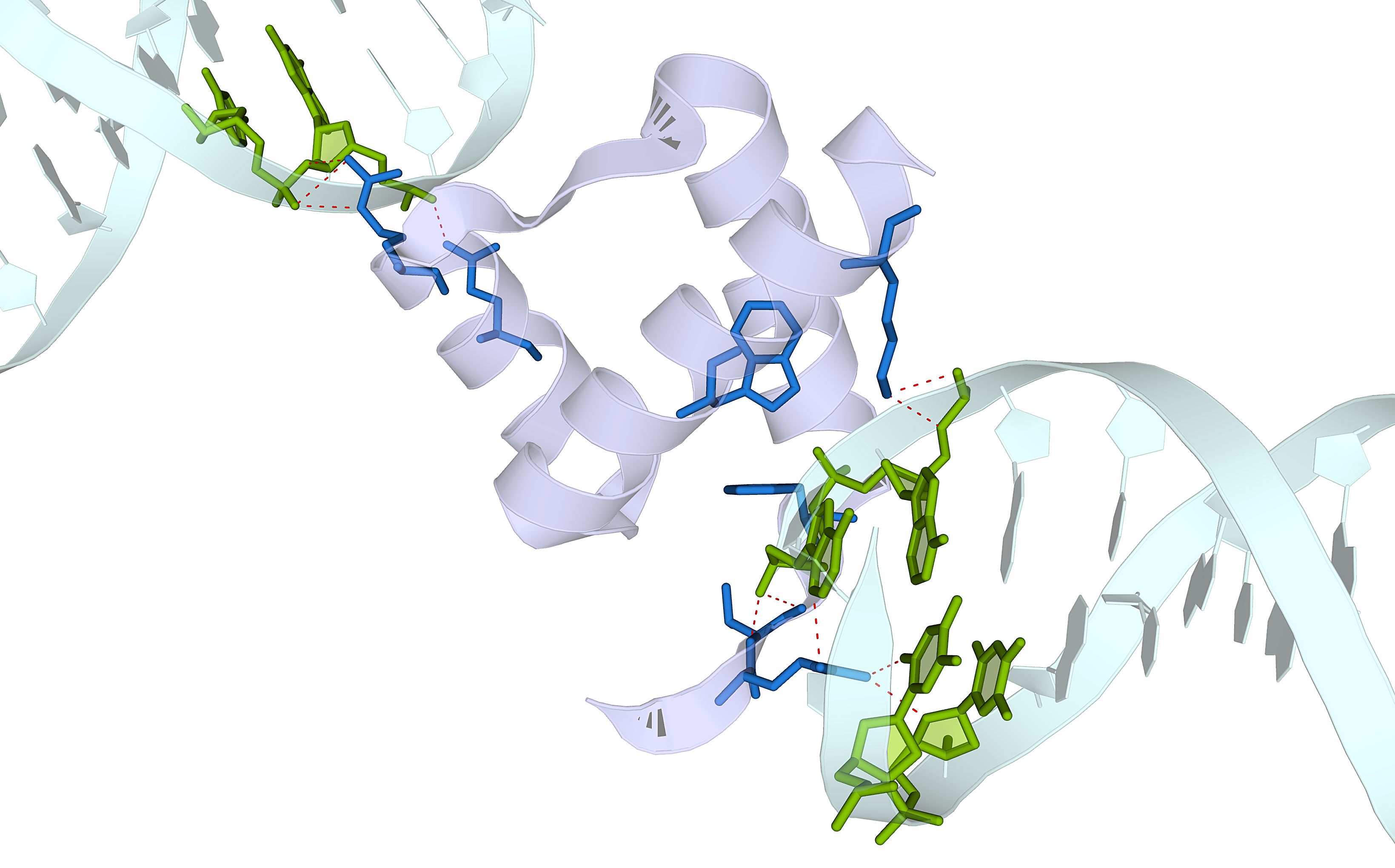
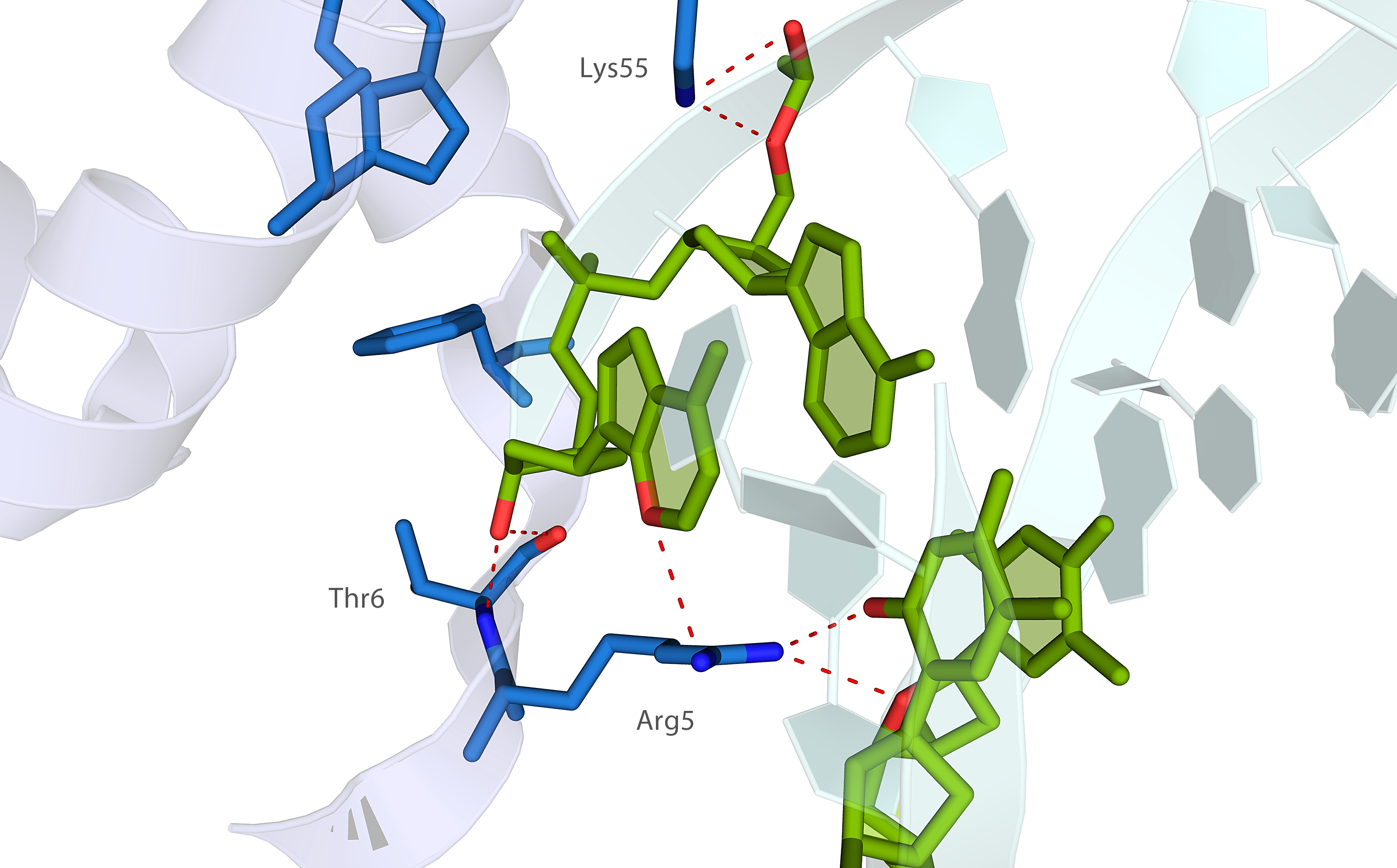
Цепь B белка взаимодействует как с цепью ДНК в «своей» ячейке, так и с цепями ДНК соседнех ячеек. Это показано на изображениях ниже:
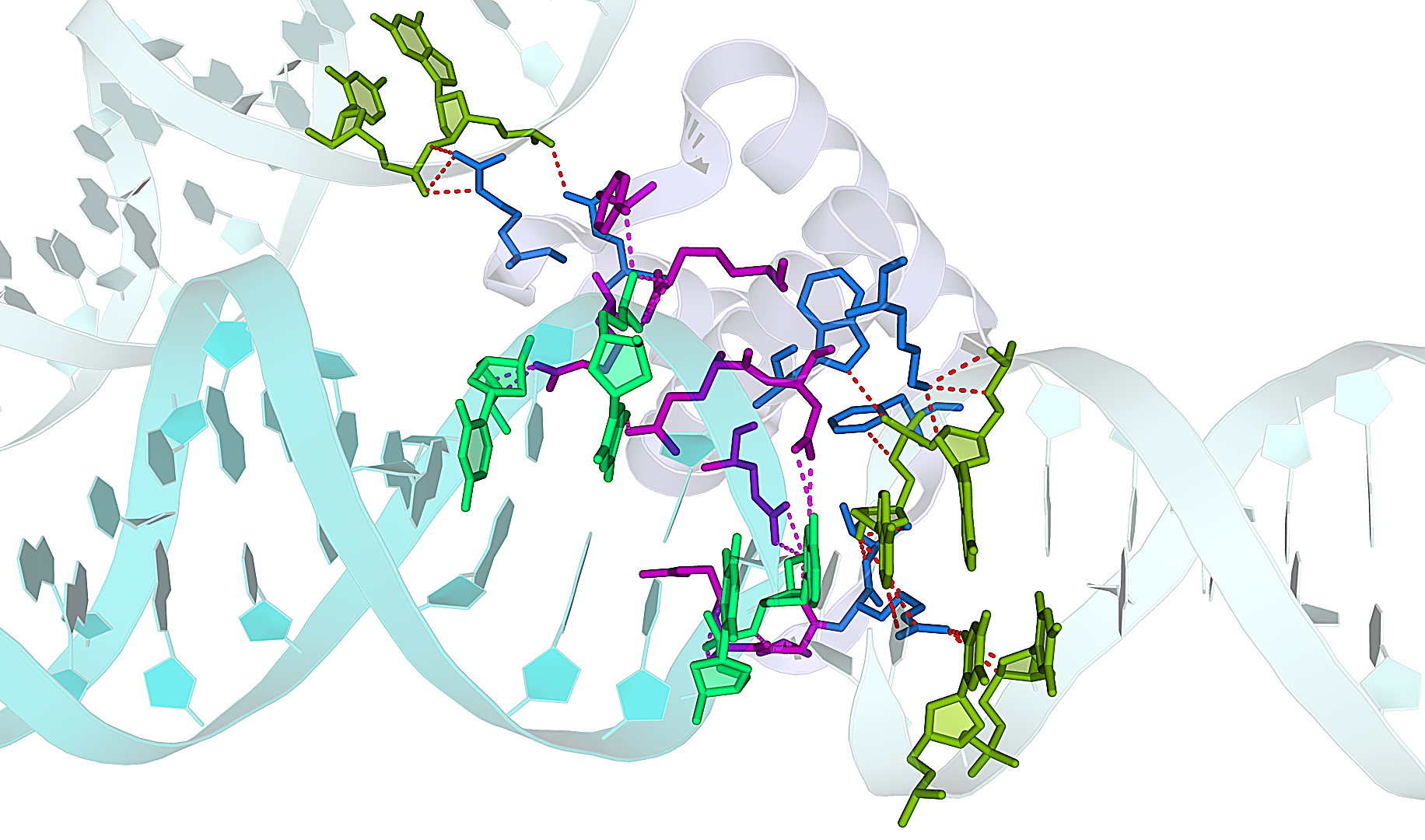
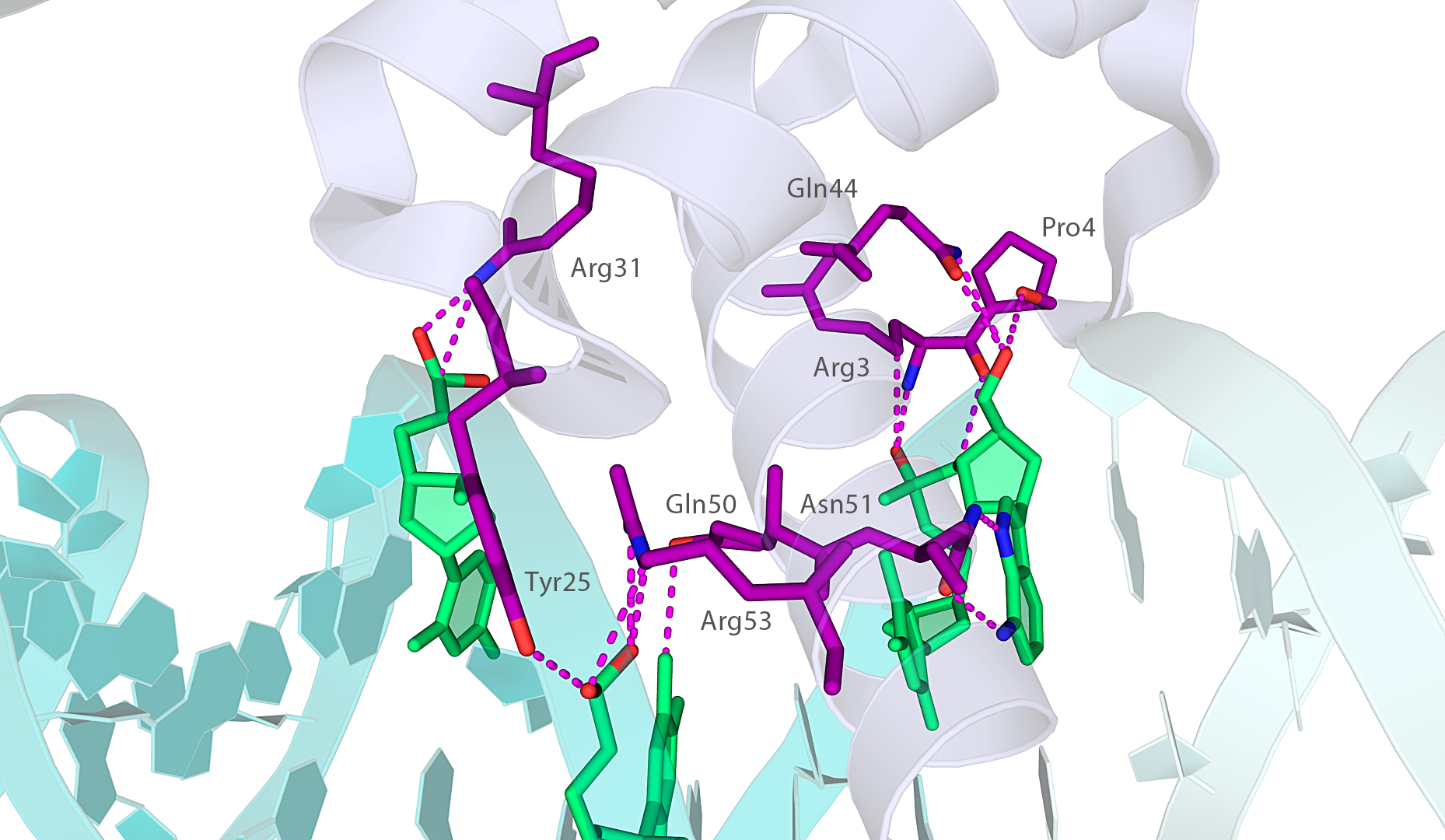
Фиолетовым и синим показаны остатки цепи B белка, расположенные на расстоянии не более 3,5 Å от молекул ДНК «своей» и соседних ячеек, соответственно. Аналогично, фрагменты ДНК показаны бирюзовым и зелёным цветами для данной и соседней ячеек, соответственно.
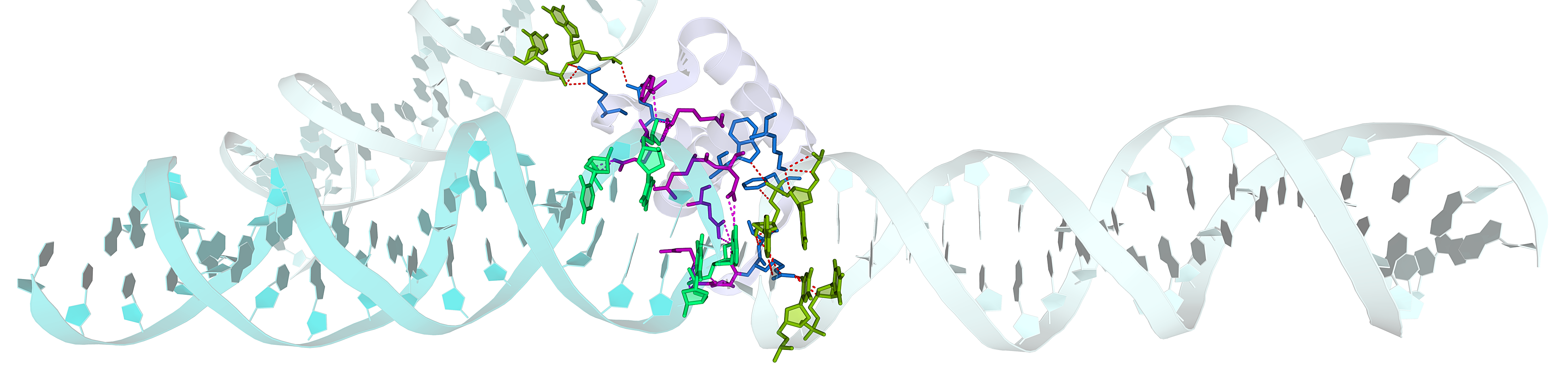
Построение соседних ячеек кристалла
С помощью команды symexp в PyMol было восстановлено изображение ДНК-белкового комплекса в соседних ячейках кристалла. Периодичность элементов хорошо прослеживается на следующем изображении:

Выводы
Как видно на примере структуры 3HDD, одной асимметрической единицы не всегда достаточно для анализа структуры. Именно визуализация молекулы ДНК соседней ячейки кристалла и выявление контактов белка с ДНК позволили объяснить странное, на первый взгляд, положение цепи B белка, находящейся «на краю» двойной спирали ДНК асимметрической единицы.
1UWZ
Построение соседних ячеек кристалла
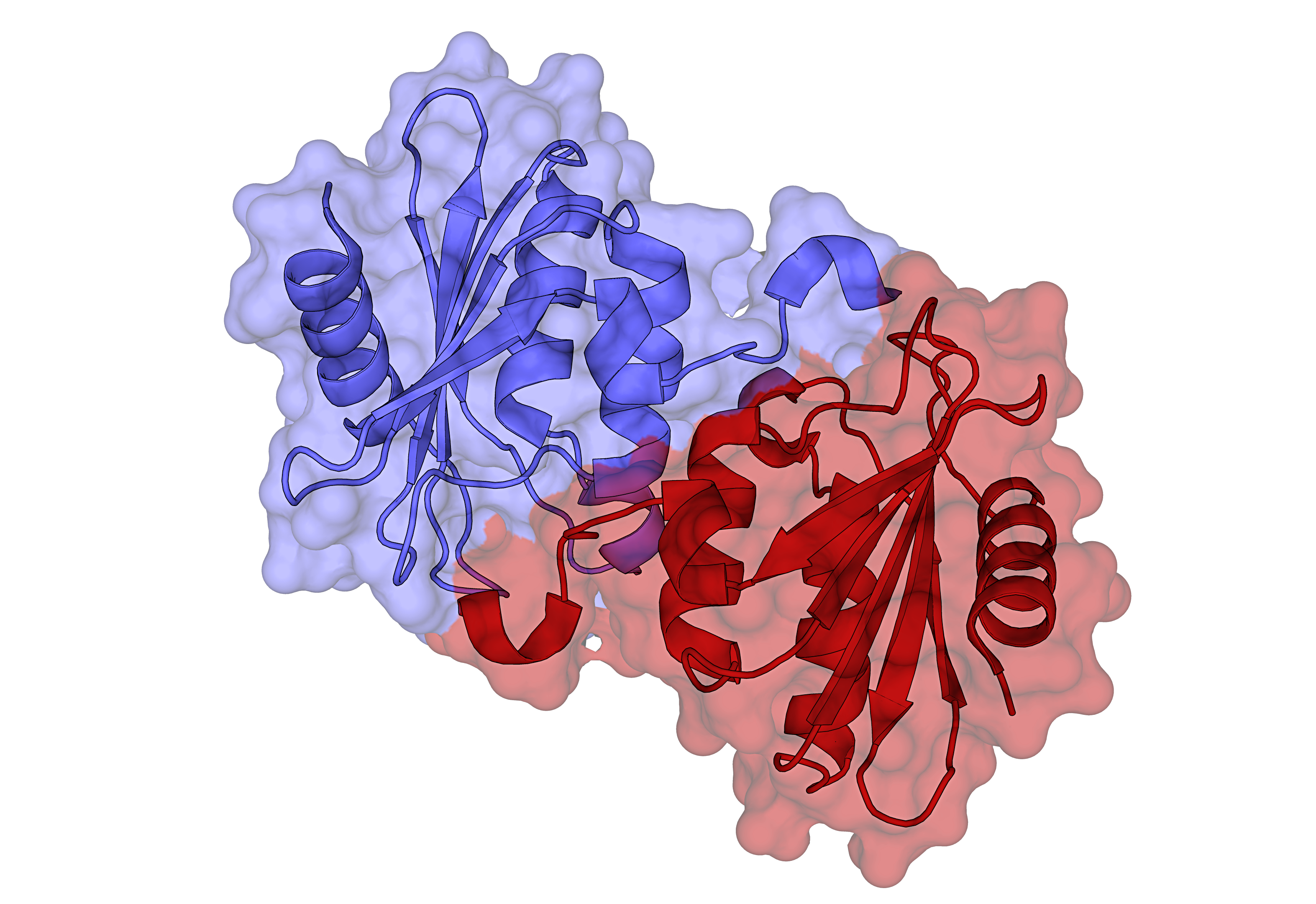
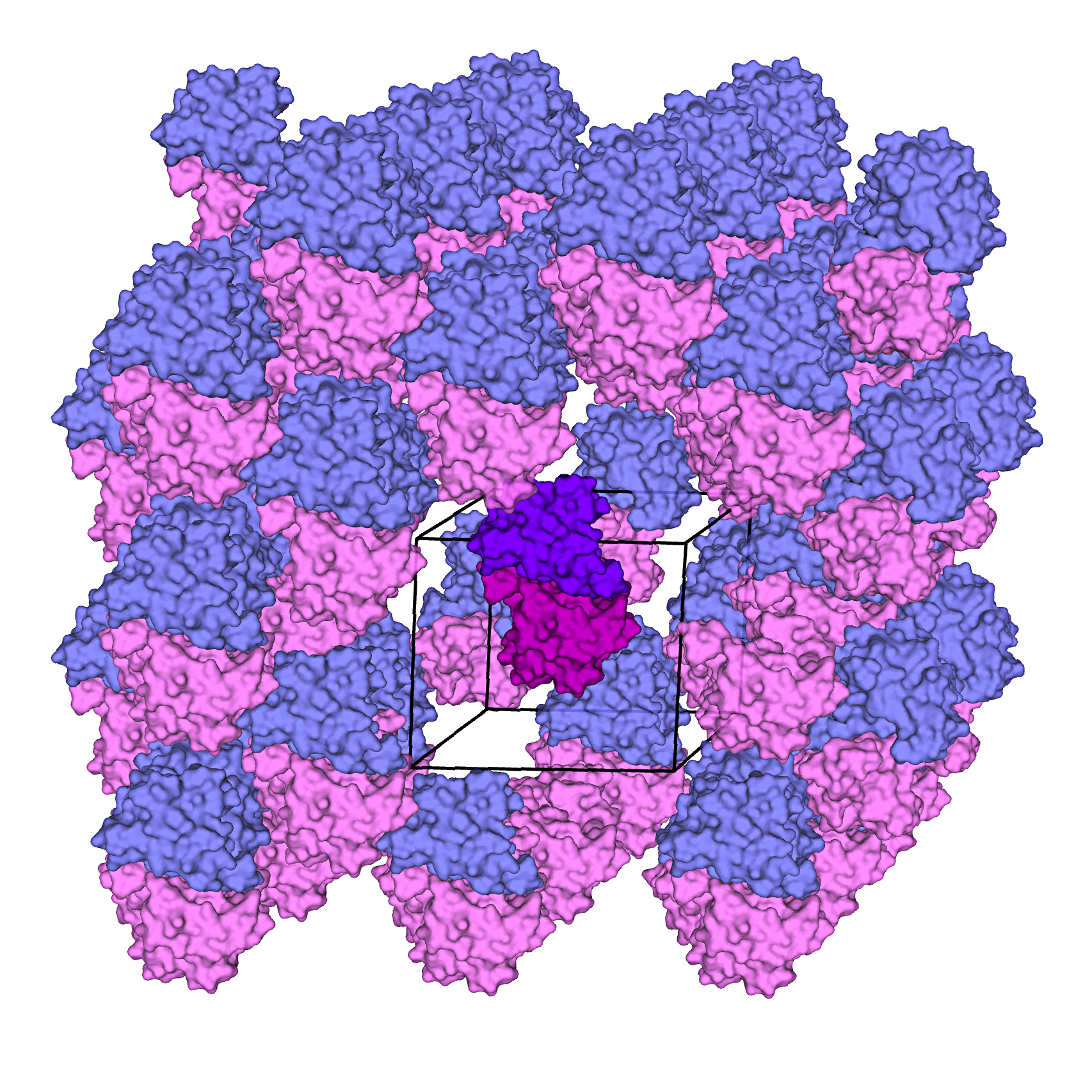
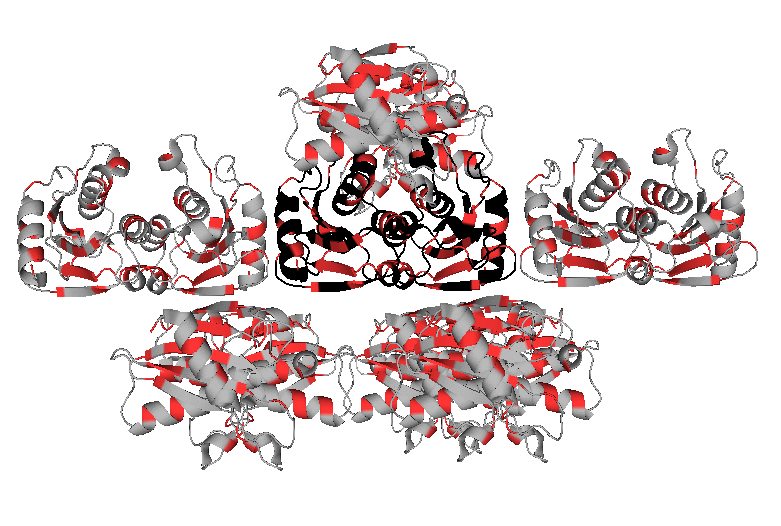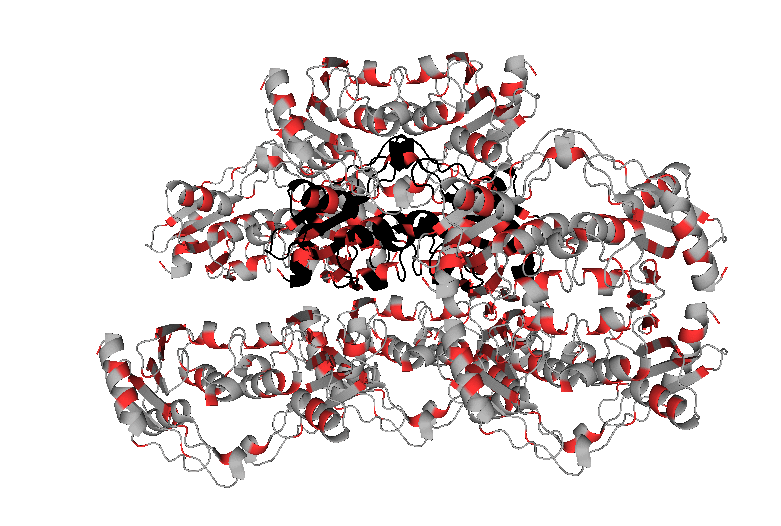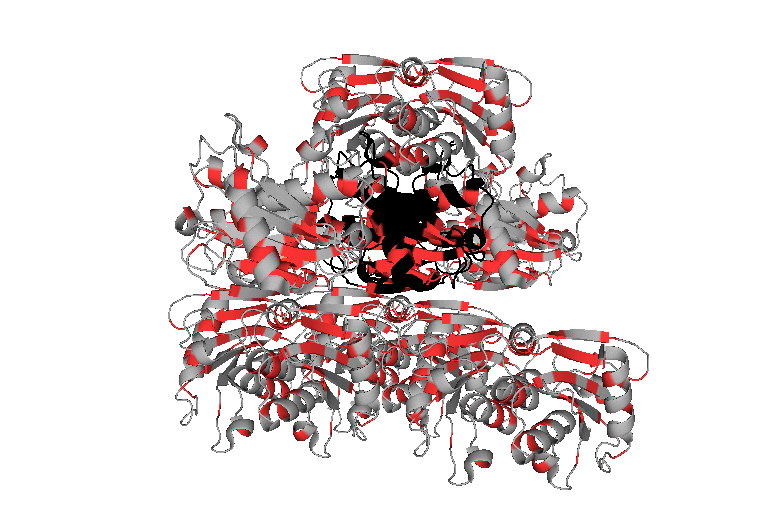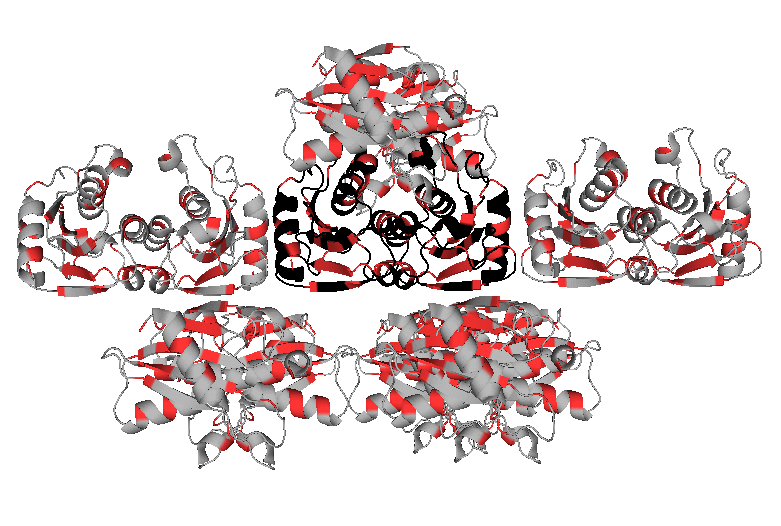
Модель 1UWZ описывает структуру белка цитидиндезаминазы Bacillus subtilis. Асимметрическая единица состоит из цепей A и B белка; активная форма белка представляет собой гомодимер или гомотетрамер. Изображения структуры белка и соседних ячеек кристалла представлены ниже.
Оттенки синего и красного (или фиолетового) цветов обозначают цепи A и B белка, соответственно. При визуализации молекул соседних ячеек кристалла белки на расстоянии 30 Å от исходной молекулы были скрыты в целях большей наглядности изображения.
Кристаллографические характеристики
В поле CRYST1 записи PDB указаны кристаллографические характеристики. Для модели 1UWZ эта строка имеет следующий вид:
CRYST1 73.743 66.418 55.388 90.00 115.60 90.00 C 1 2 1 8
Первые три числа – значения длин направляющих векторов кристалла, следующие три – углы между направляющими векторам. Число молекул в ячейке равно 8. Название кристаллографической группы C 1 2 1 означает, что элементарная ячейка 1UWZ – моноклинной сингонии (α,γ = 90˚, β ≠ 90˚), с примитивной решёткой Бравэ. Для этой пространственной группы заданы следующие операторы симметрии:
X, Y, Z
-X, Y, -Z # поворот на 180˚ в плоскости XZ
X+1/2, Y+1/2, Z # смещение
-X+1/2, Y+1/2, -Z # поворот со смещением
Анализ внутрикристаллических контактов
Чтобы проследить водородные связи и полярные взаимодействия между белками соседних ячеек, были использованы, в частности, следующие команды в PyMol:
symexp symp1, 1uwz, 1uwz, 3, 1 select this, byres (1uwz within 3.5 of symp1*) select that, byres (symp1* within 3.5 of 1uwz) distance hbonds, this, that, 3.5, 2
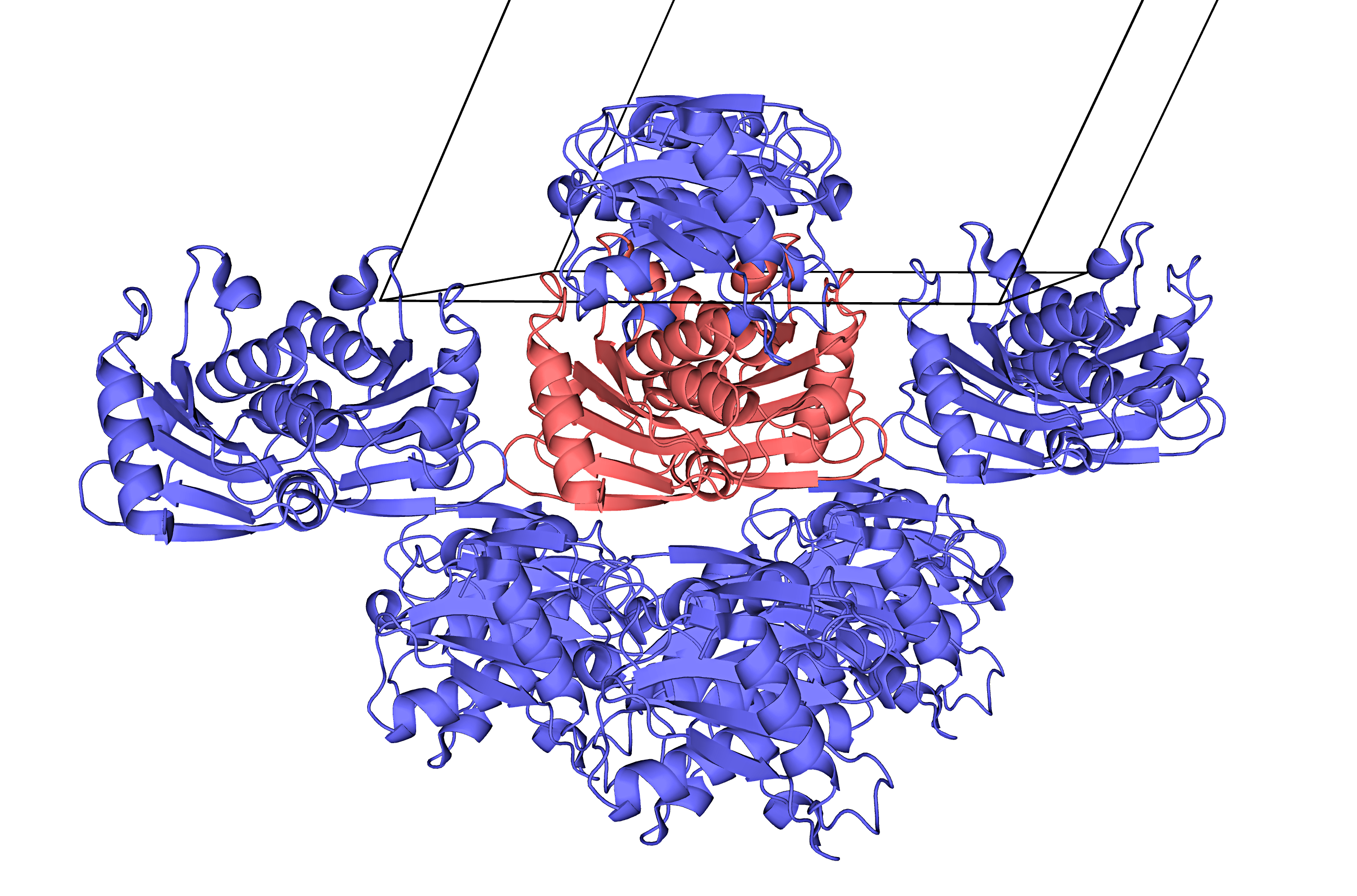
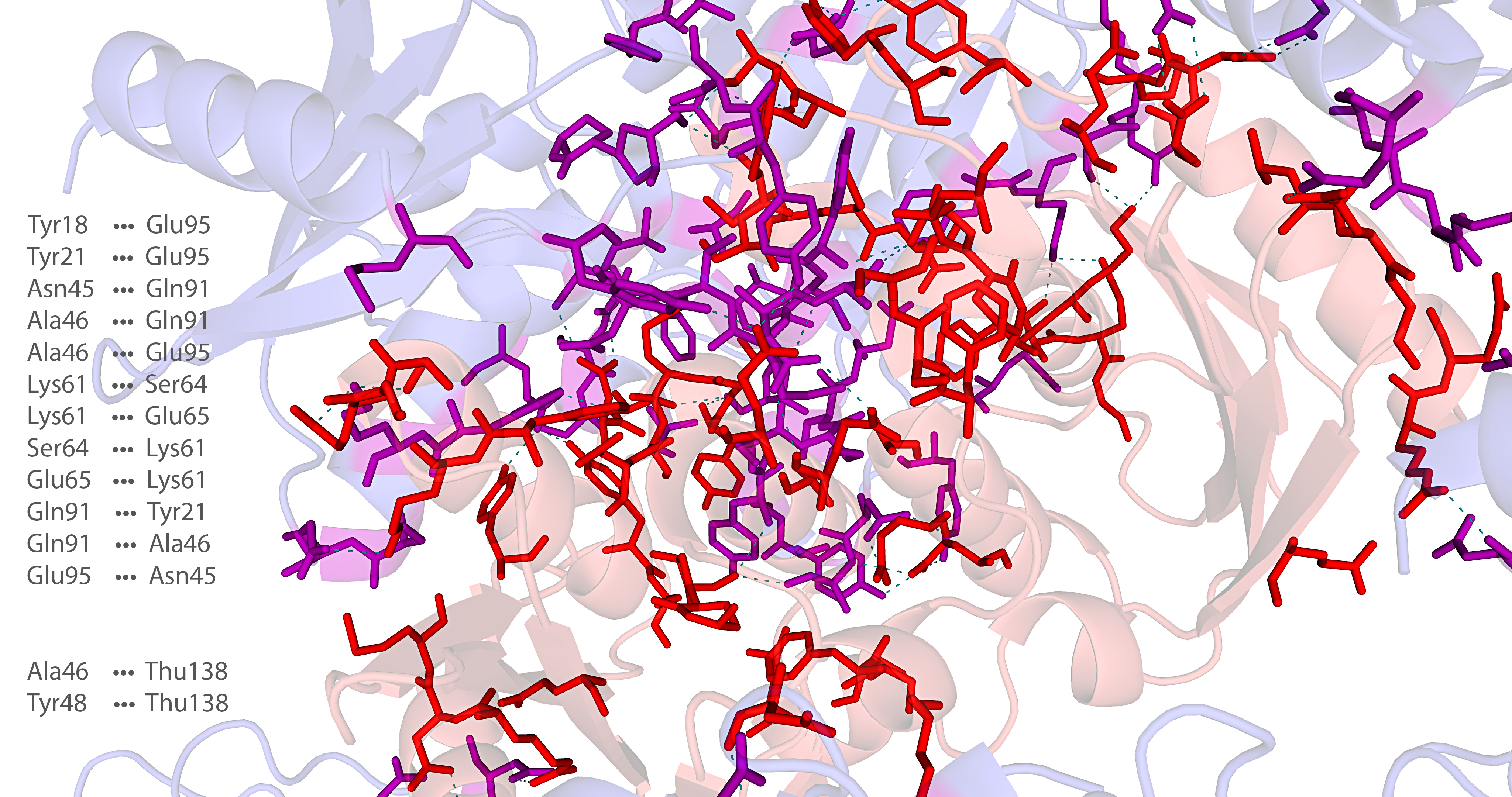
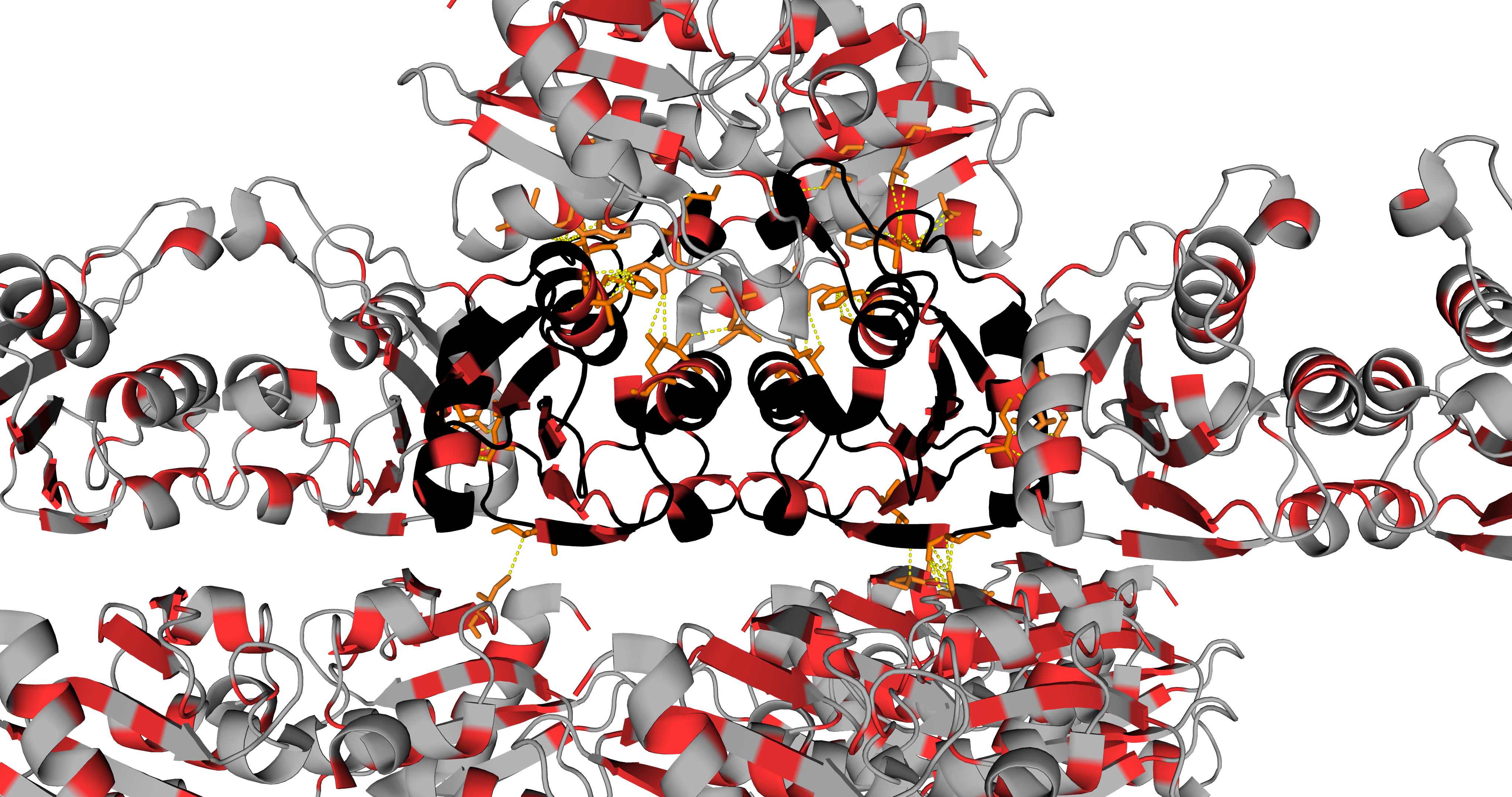
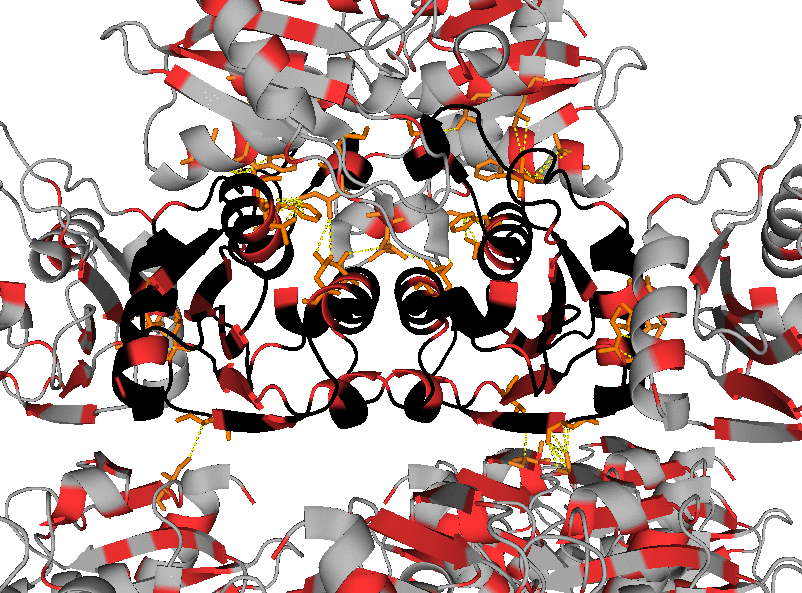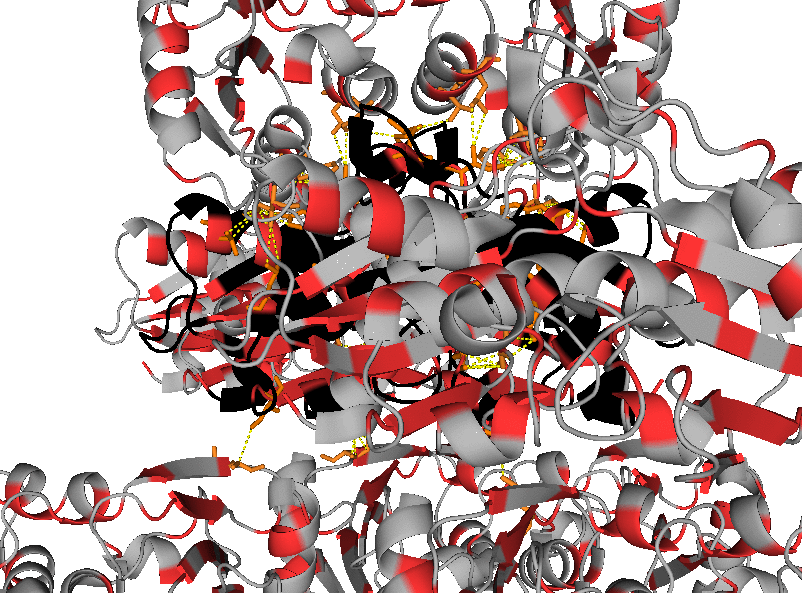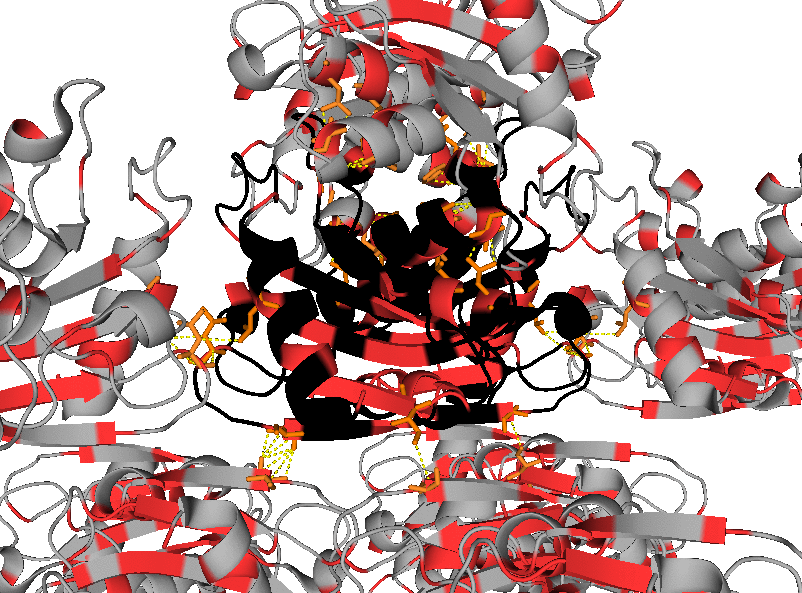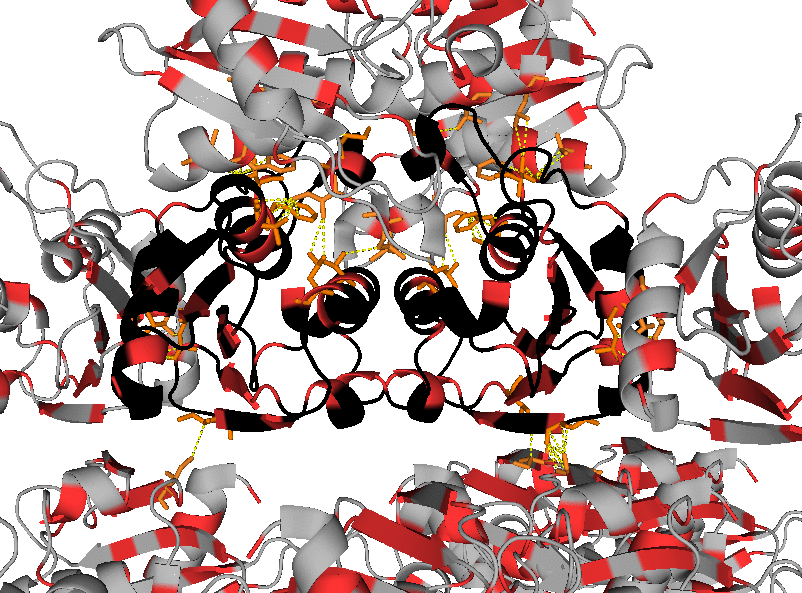
Как можно видеть на изображениях ниже, молекула белка в кристалле имеет множество контактов с соседствующими молекулами. Так как молекулы белка компактно расположены в кристалле, рассмотрение взаимодействий на изображениях приведено для отдельных фрагментов структуры.
Красный оттенок на изображениях ниже соответствует исходной молекуле белка в файле, синий – её соседям. Остатки молекул, удалённые на расстояние менее 3,5 Å, отмечены цветами, отличными от цветов цепей белка, которым принадлежат эти остатки. Взаимодействия показаны зелёной пунктирной линией.
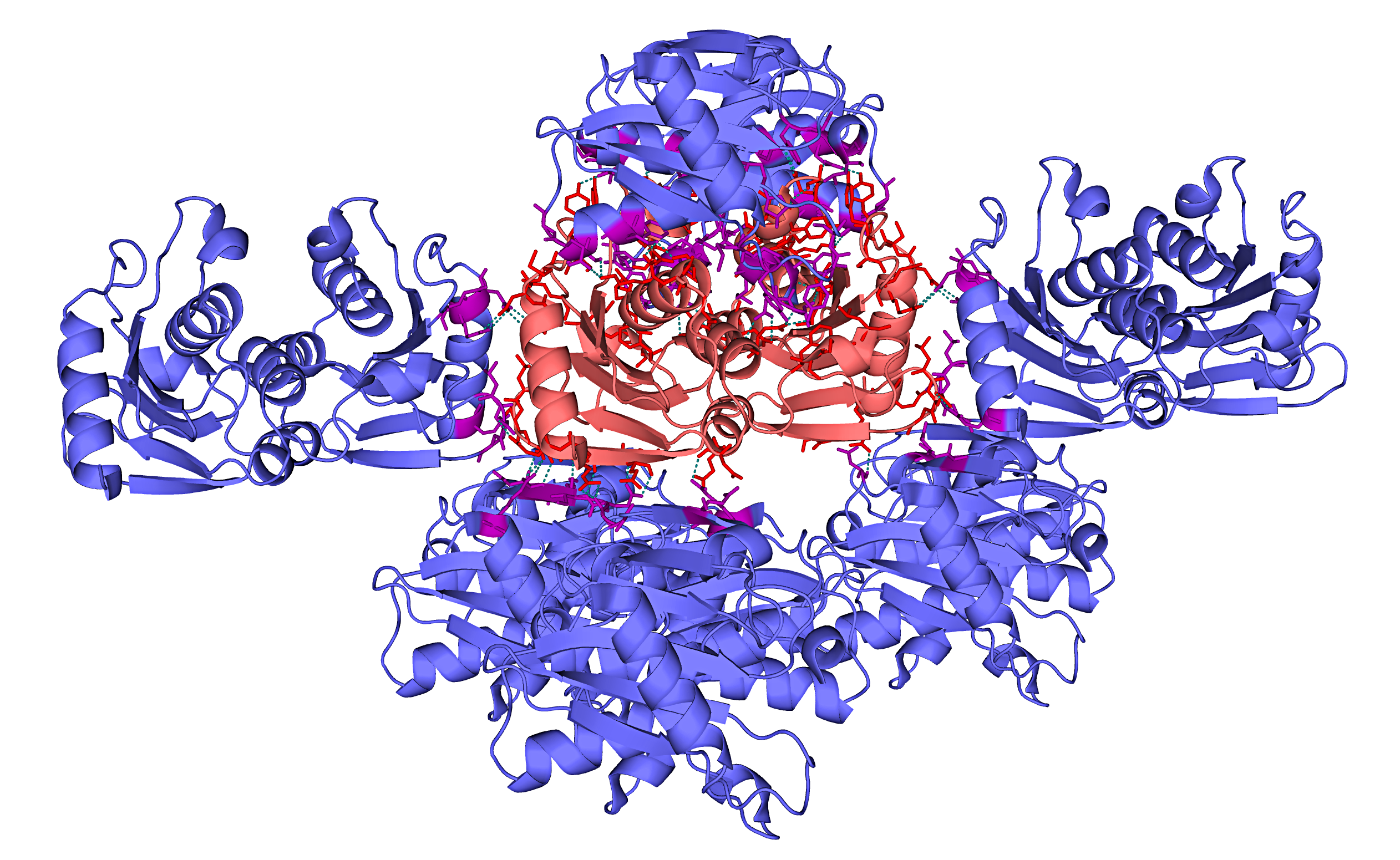
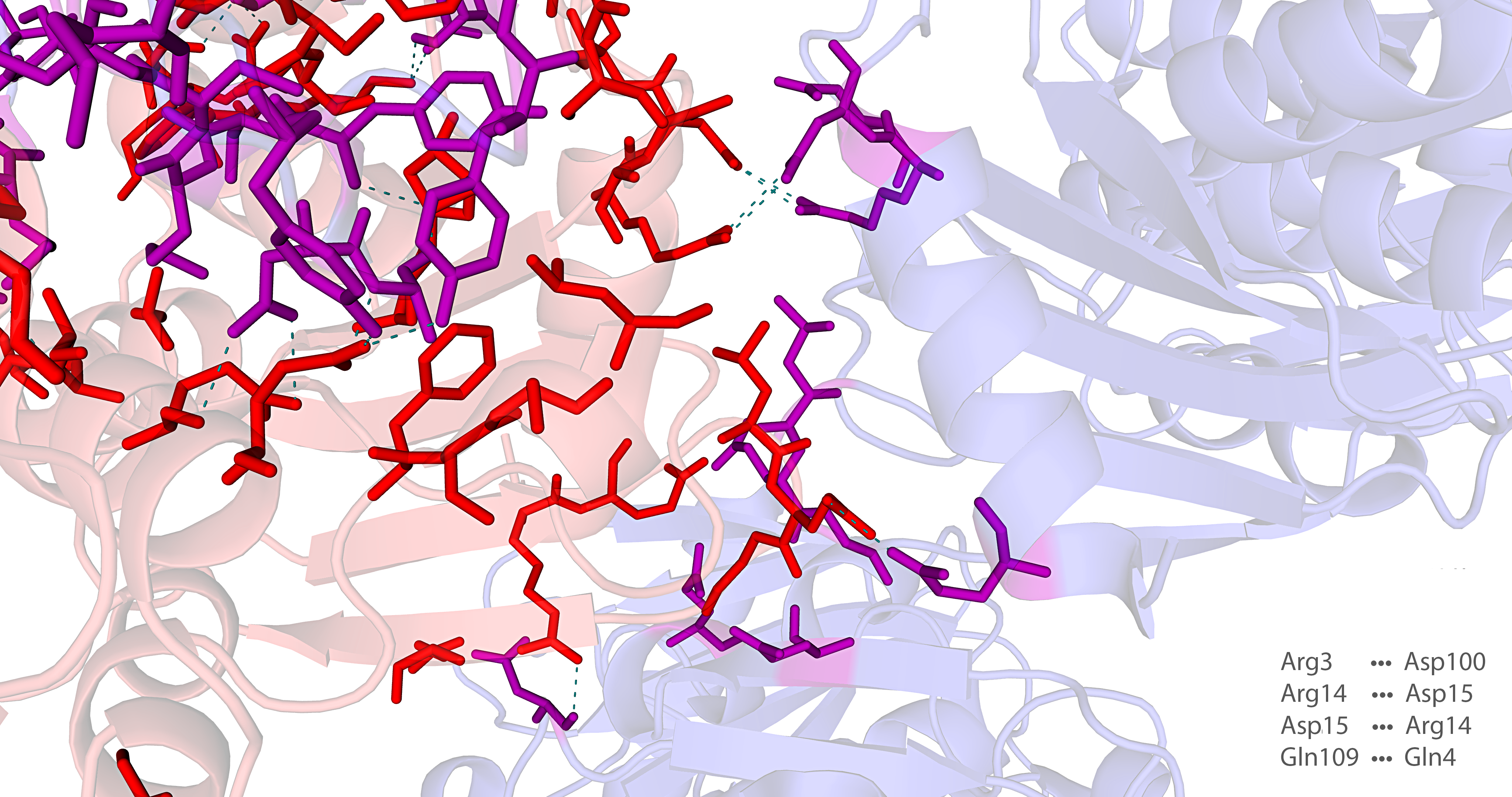

Следует отметить, что при рассмотрении взаимодействий ярко проявляется симметрия структуры. Например, присутствуют водородные связи Arg3 ••• Asp100 и Asp100 ••• Arg3, Arg14 ••• Asp15 и Asp15 ••• Arg14.
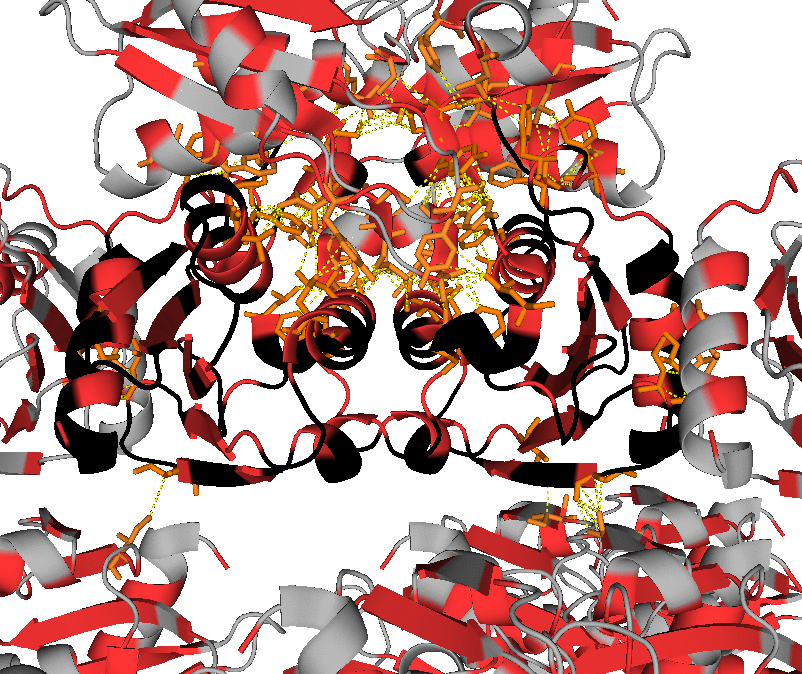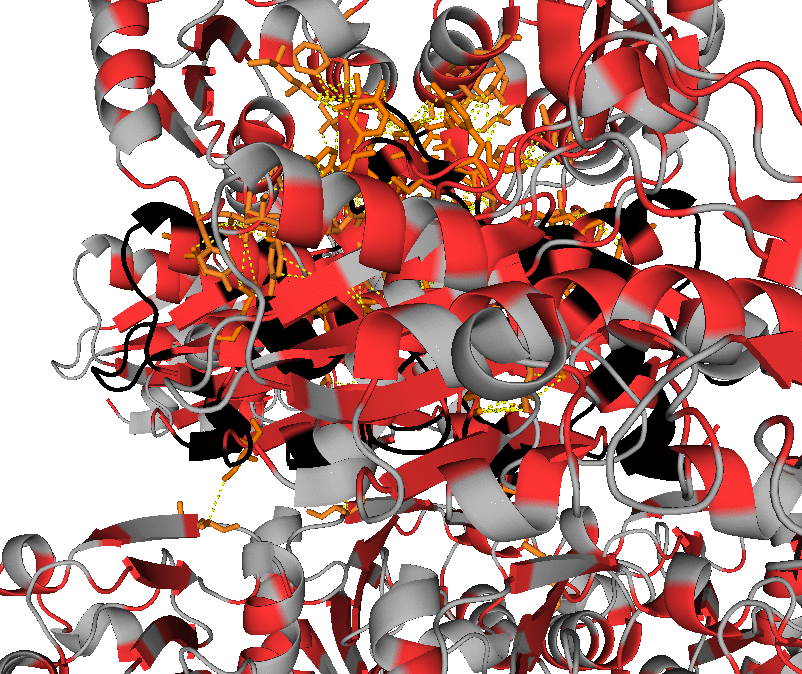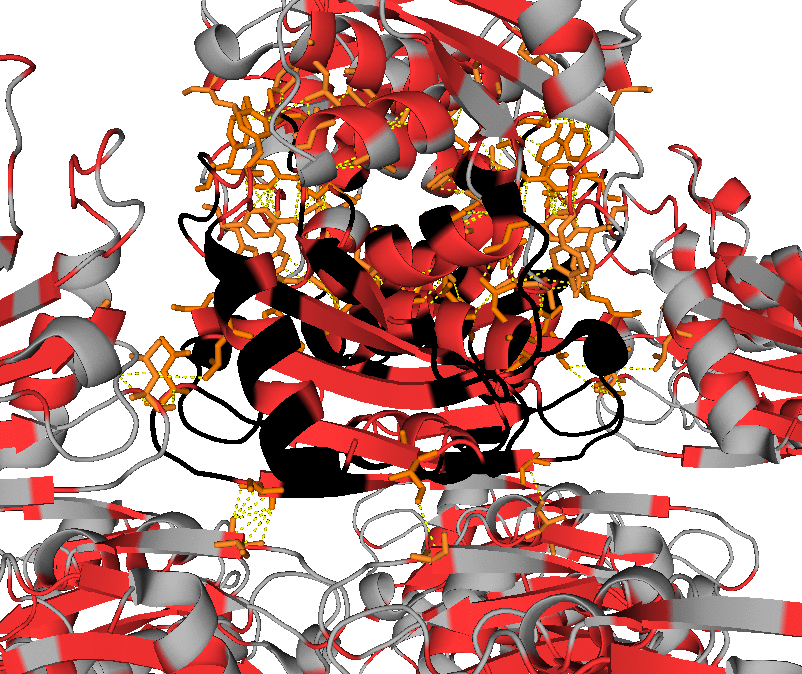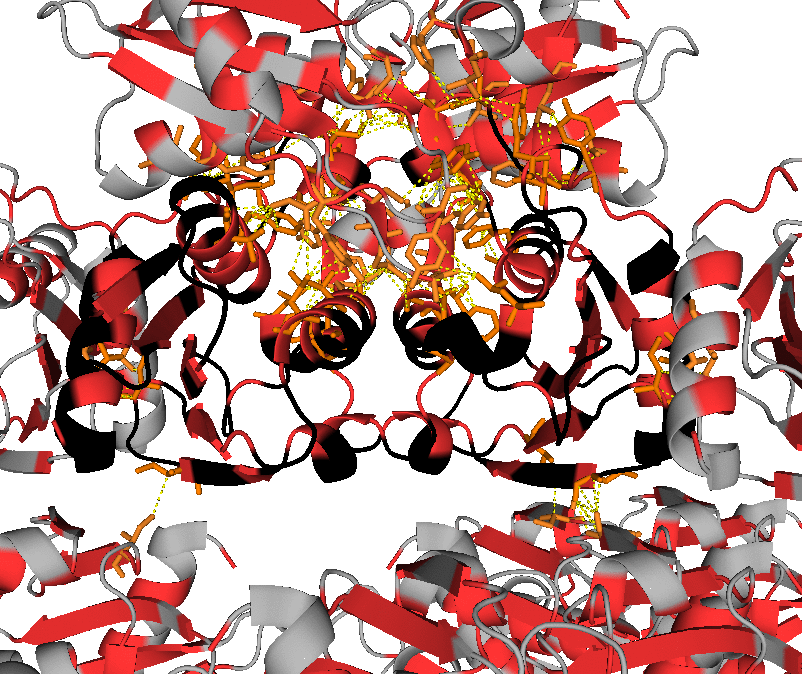
В файле модели структуры 1UWZ можно найти молекулы тетрагидродезоксиуридина (Thu) – ингибитора цитидиндезаминаз. Взаимодействия с ним белка были обнаружены при визуализации полярных контактов и водородных связей (см. фрагмент 3 ↑ ).
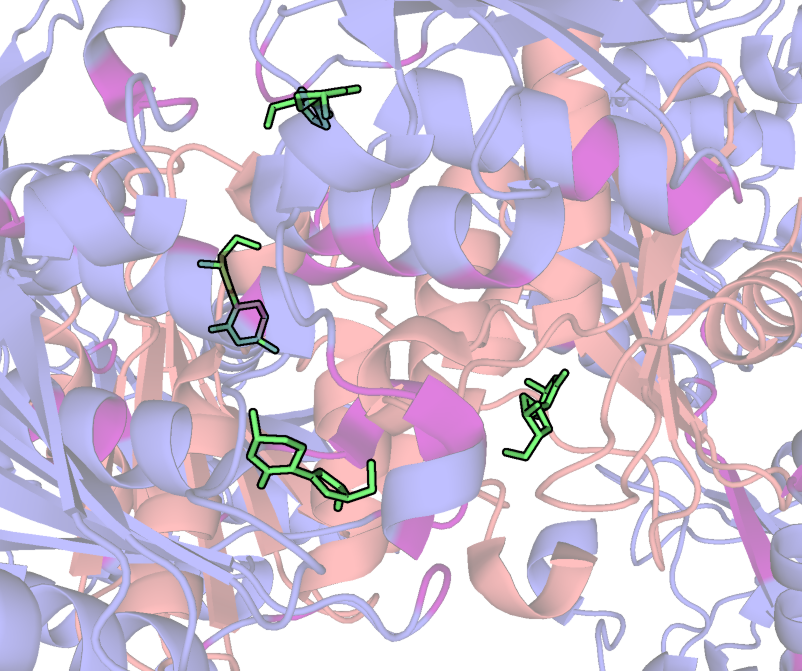
Необходимо также уделить внимание гидрофобным взаимодействиям. Для их визуализации в PyMol были использованы, в частности, команды:
select hyph, resn leu+ile+val+phe+trp+met symexp symp1, 1uwz, 1uwz, 3, 1 distance hyphint, byres(hyph and 1uwz), byres(hyph and symp1*), 4 select hyph_res1, byres((hyph and 1uwz) within 4 of (hyph and symp1*)) select hyph_res2, byres((hyph and symp1*) within 4 of (hyph and 1uwz)) show sticks, hyph_res*
Были получены изображения, демонстрирующие наличие гидрофобных взаимодействий между соседствующими молекулами белка в кристалле (гидрофобные аминокислоты в структуре отмечены оранжевым или красным цветами). На их основании можно сделать следующее предположение: хотя интермолекулярные гидрофобные контакты присутствуют, интрамолекулярные гидрофобные взаимодействия более выражены. Также можно предположить, что гидрофобные взаимодействия играют важную роль при олигомеризации (на основании большого количества потенциальных гидрофобных контактов между димерами).
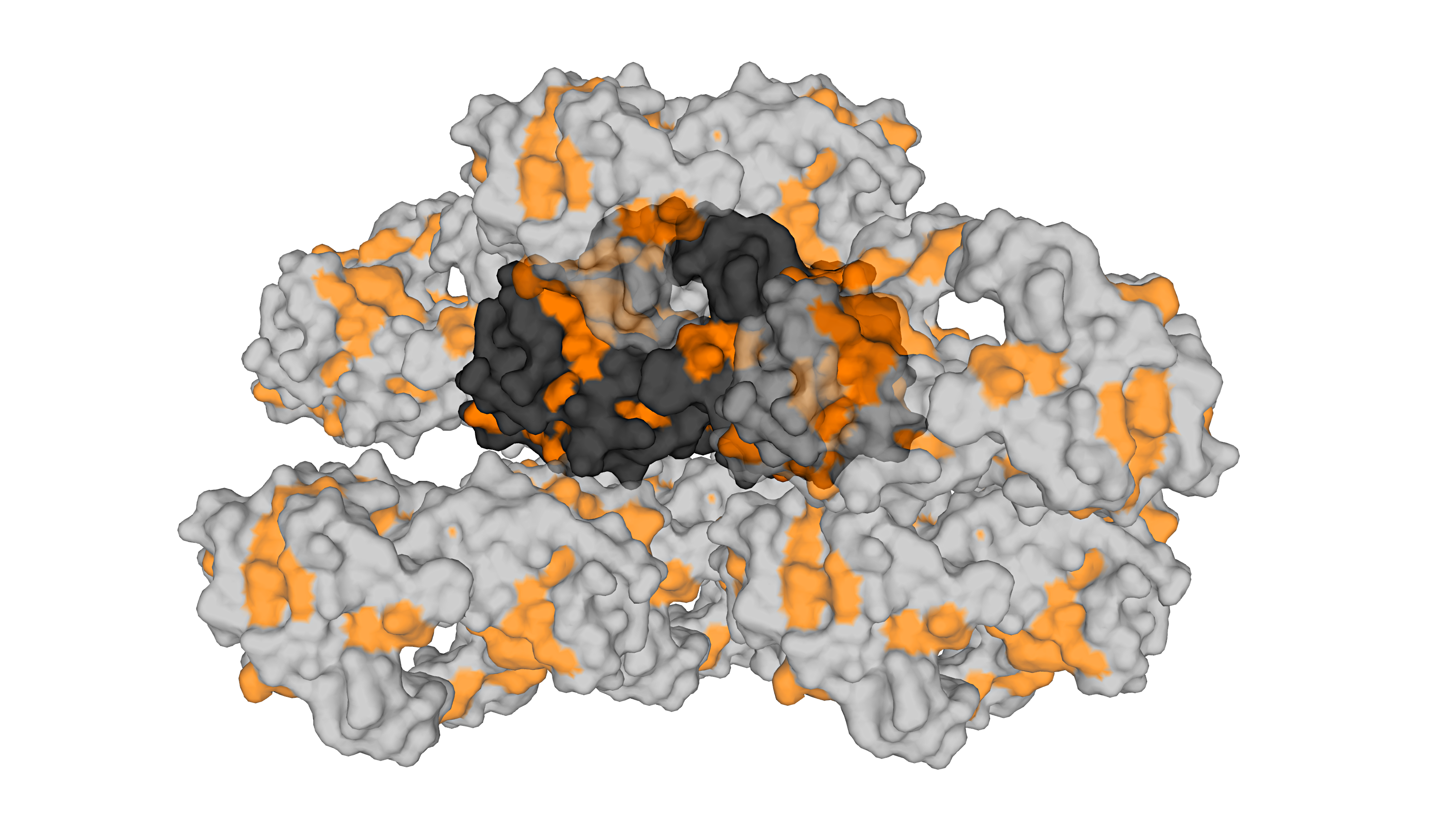
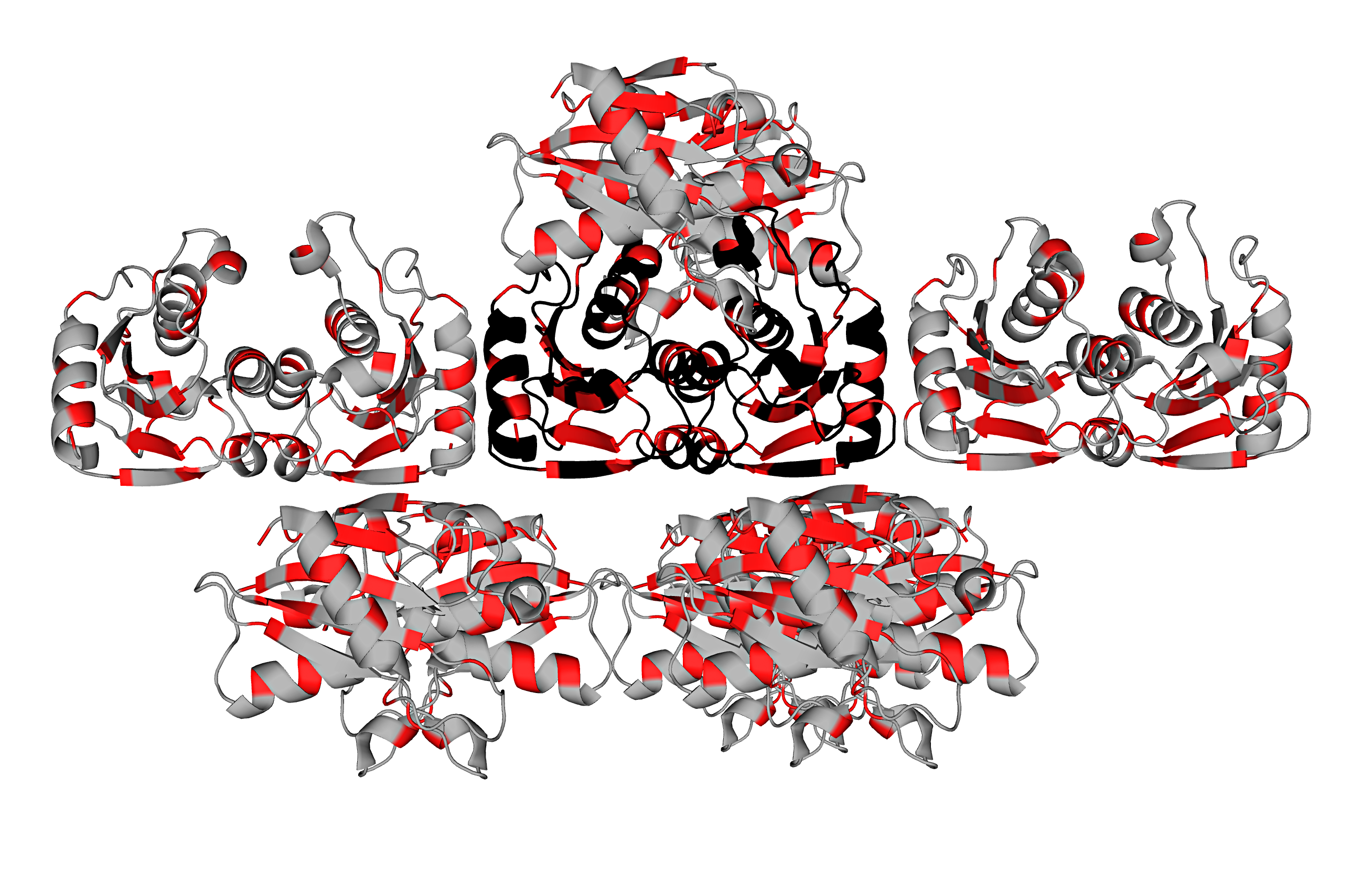
Выводы
Анализ взаимодействий белка с соседствующими в кристалле молекулами позволяет получить более полное представление о структуре. Рассмотрение и визуализация этих контактов для структуры 1UWZ позволили продемонстрировать свойство симметрии в кристалле, а также получить представление о взаимодействии (в частности, о полярных и водородных связях, гидрофобных контактах) молекул соседних ячеек. В случае структуры 1UWZ анализ таких взаимодействий позволяет также получить информацию о взаимодействии субъединиц олигомерного белка.