Учебный сайт
Бредихина Данилы
- VII
- ЯМР-спектроскопия. Биологические единицы
Запись PDB, полученная методом ЯМР
На сайте PDB из структур, решённых методом NMR в 2014 году, была выбрана структура 2MM9. Запись 2MM9 содержит 20 моделей. Структура представляет собой мономер; длина белковой цепи составляет 93 остатка.

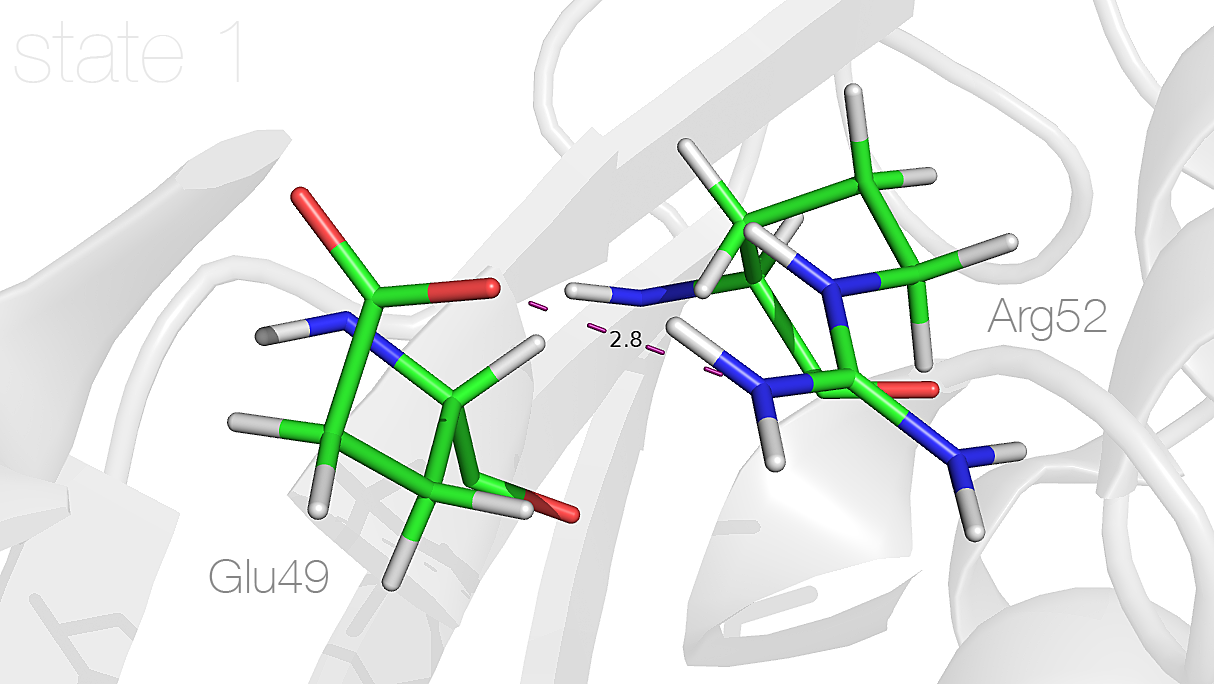
В первой модели была найдена водородная связь между боковыми цепями Arg52 и Glu49 (NH2···OE1) с помощью, в частности, следующих команд в PyMol:
# Поиск водородных связей атомов NH1 или NH2 аргинина # и OE1 или OE2 глутамата select R, name NH* within 3.5 of (resn GLU and name OE*) select E, (resn GLU and name OE*) within 3.5 of name NH* distance bond, R, E, 3.5
Если рассмотреть эту пару остатков в модели 6, их взаимное расположение значительно отличается от такового в первой модели; при этом расстояние между соответствующими атомами становится больше, и уже нельзя сделать заключение об образовании водородной связи:
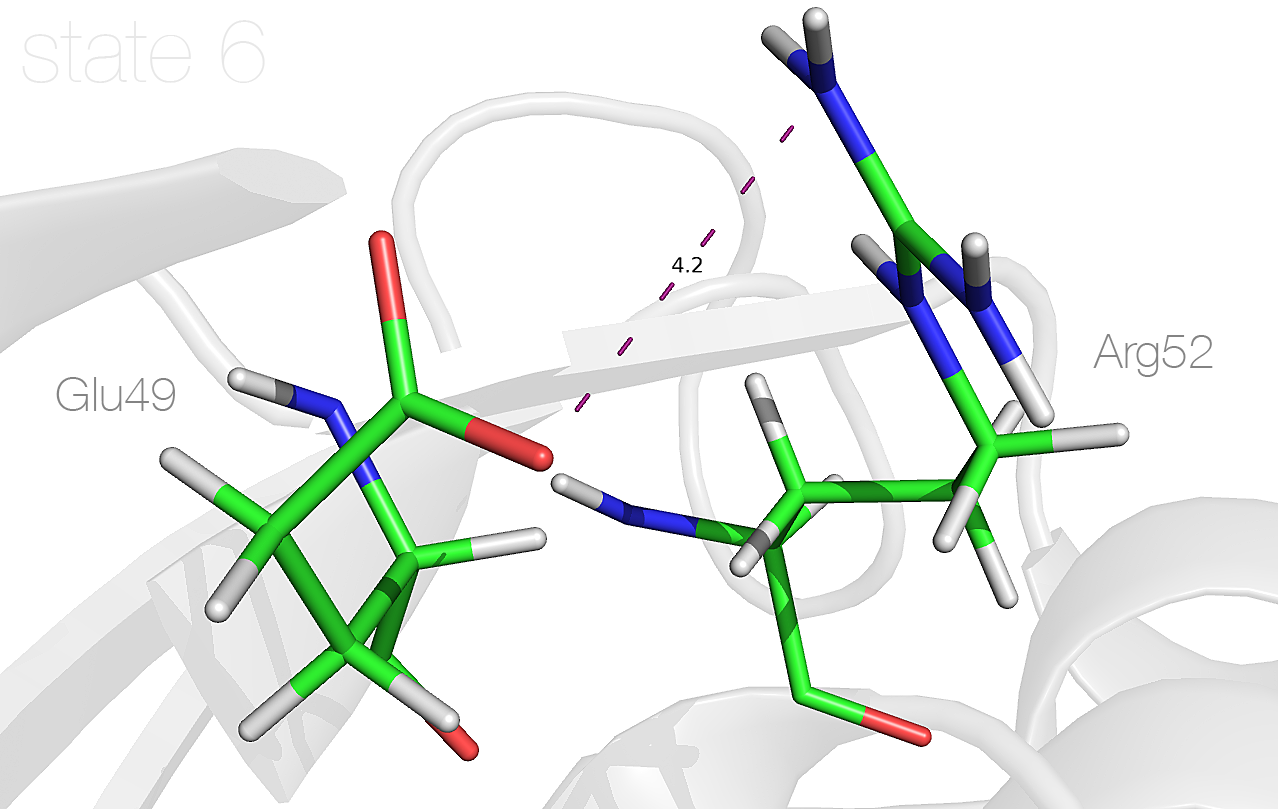
Наблюдаемое различие в пространственном расположении остатков является следствием неоднозначного восстановления укладки белка в эксперименте ЯМР.
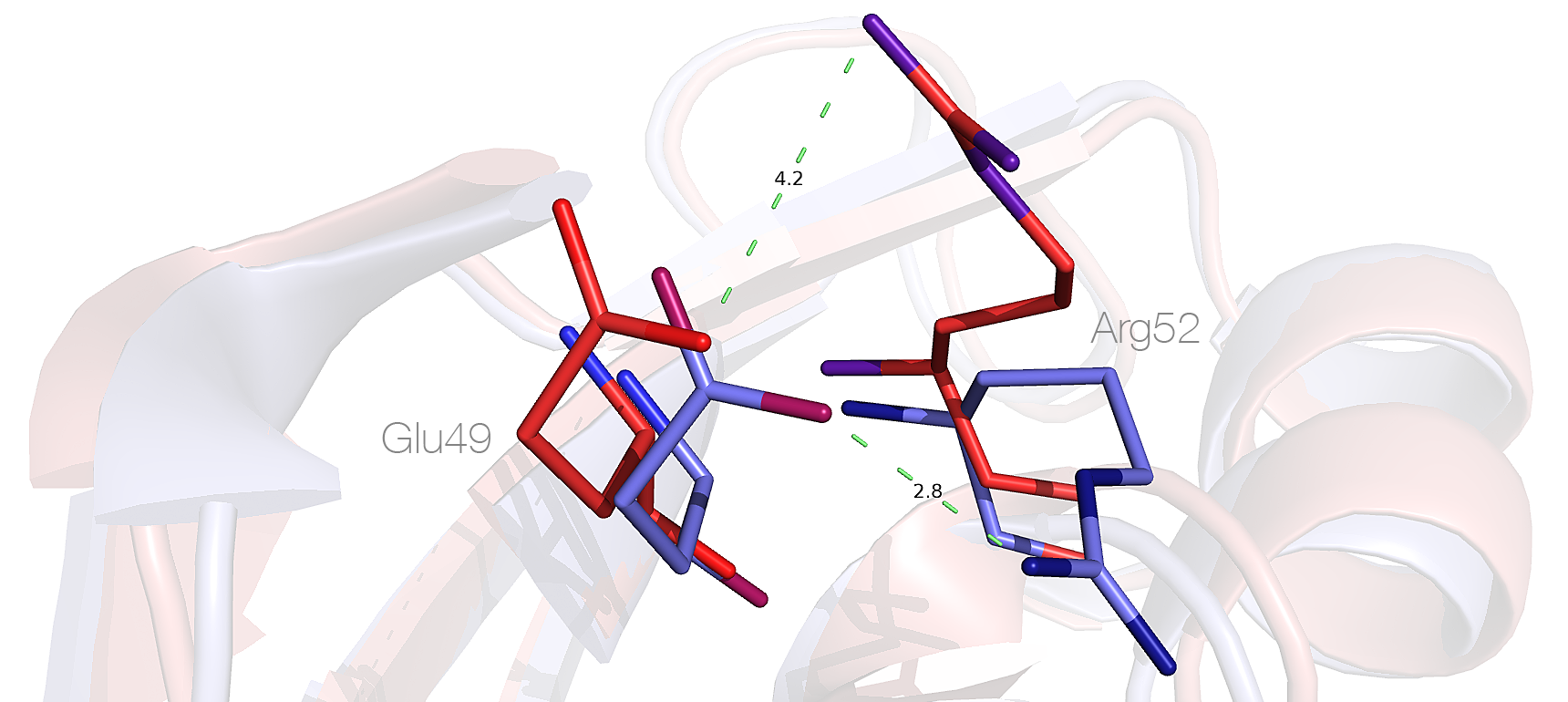
На изображении ниже показаны остатки Arg52 и Glu49 в первой (синие цвета) и шестой (красные цвета) моделях, а также расстояние между одинаковой парой атомов.
Биологические единицы
Структура 4CPE стрептавидина была разрешена в 2014 году (рентгеноструктурный анализ, разрешение 1.06 Å). Асимметрическая единица структуры включает две цепи (A и B), в то время как биологическая единица – четыре цепи. На изображении ниже разные копии асимметрической единицы выделены разными цветами:
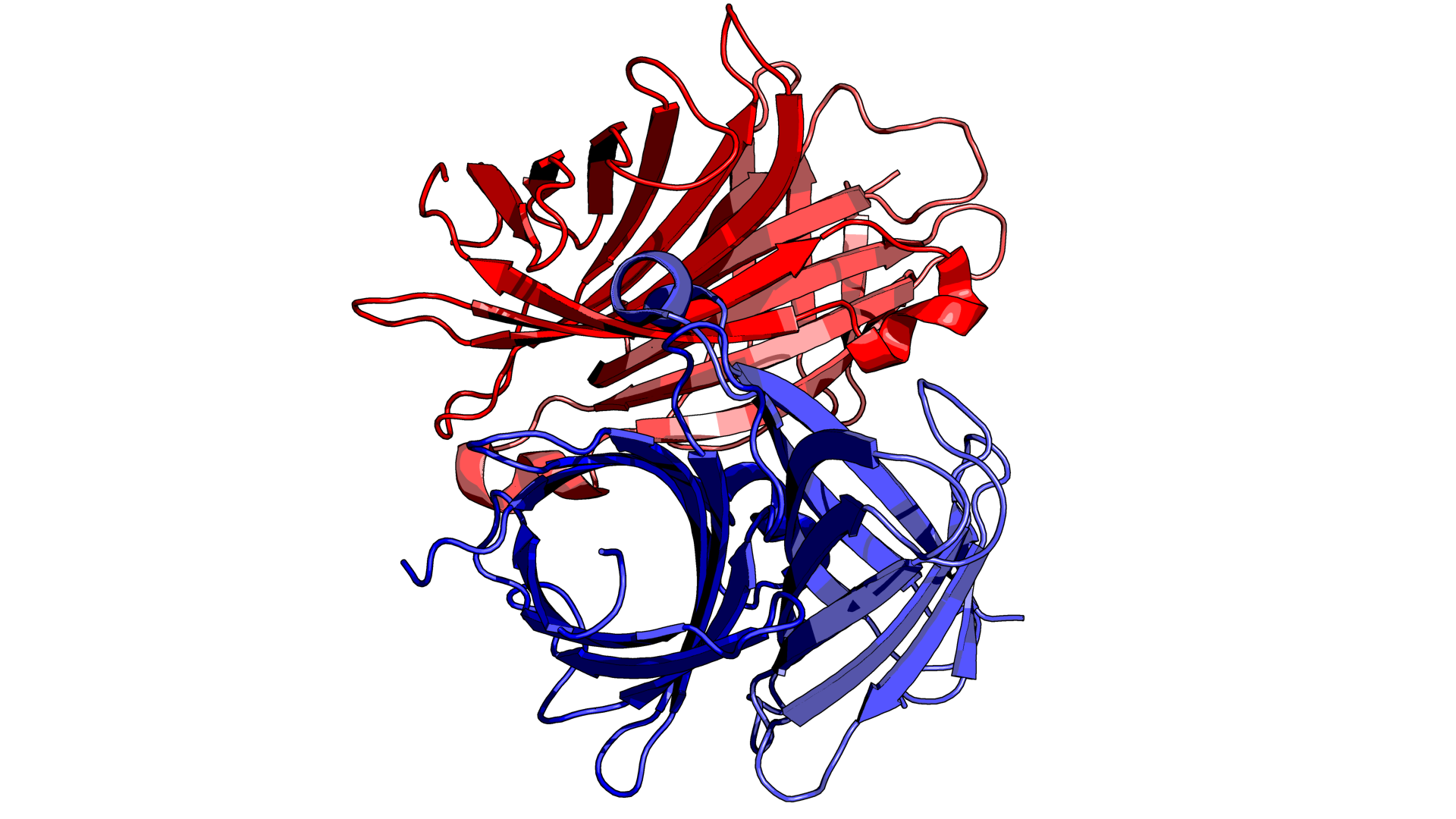
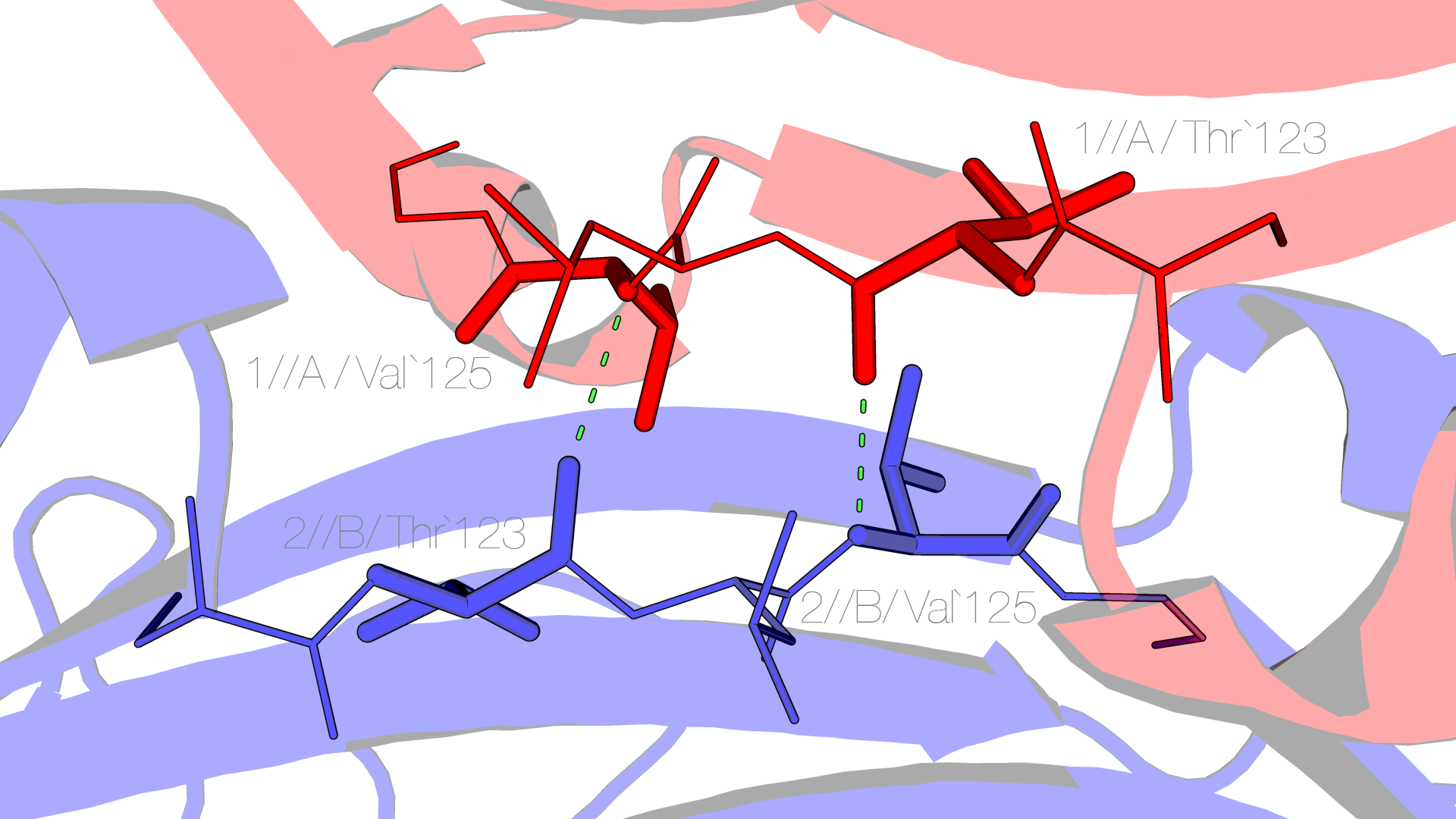
Между остатками разных копий асимметрической единицы можно найти водородные связи. Пример таких связей (A/Thr`123/O ··· B/VAL`125/N и B/Thr`123/O ··· A/VAL`125/N разных копий) приведён на следующем изображении:
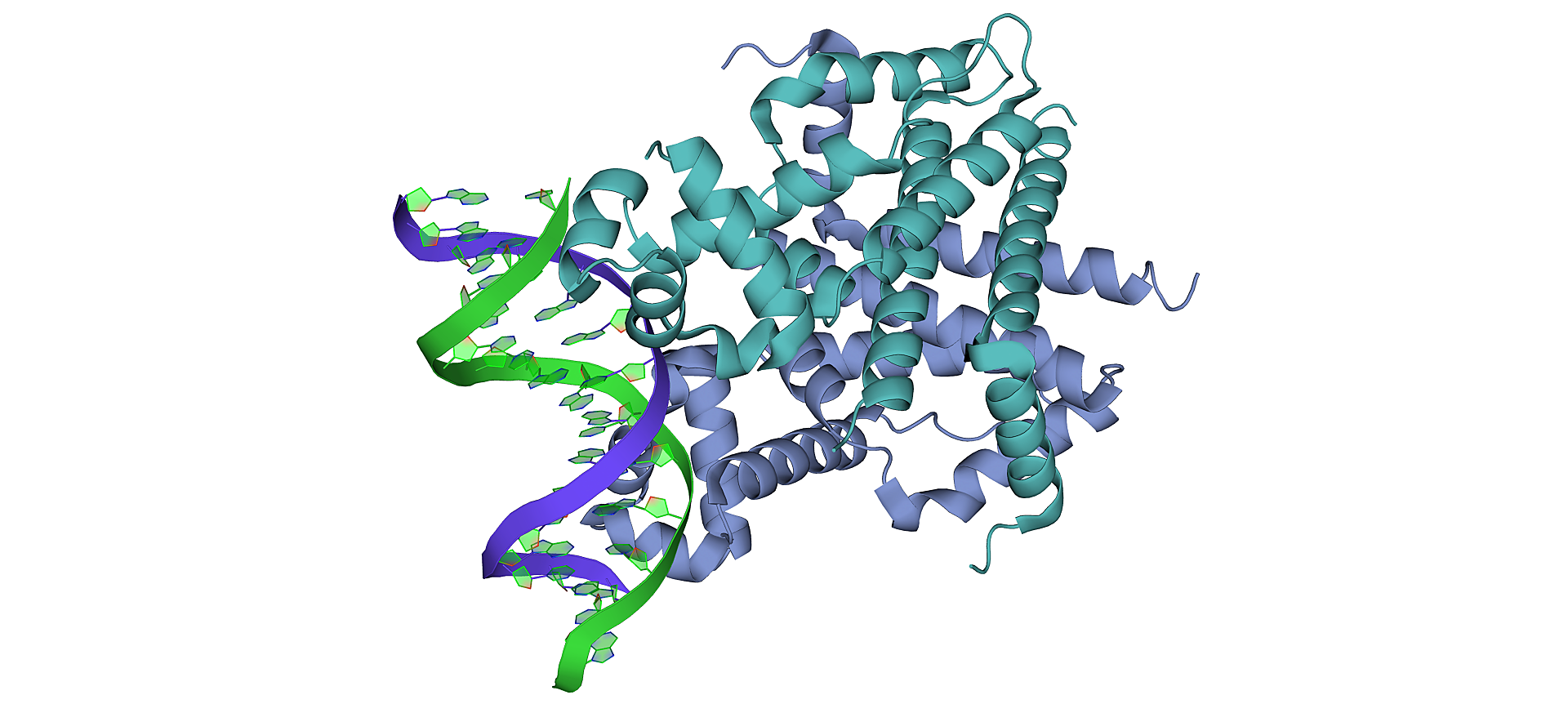
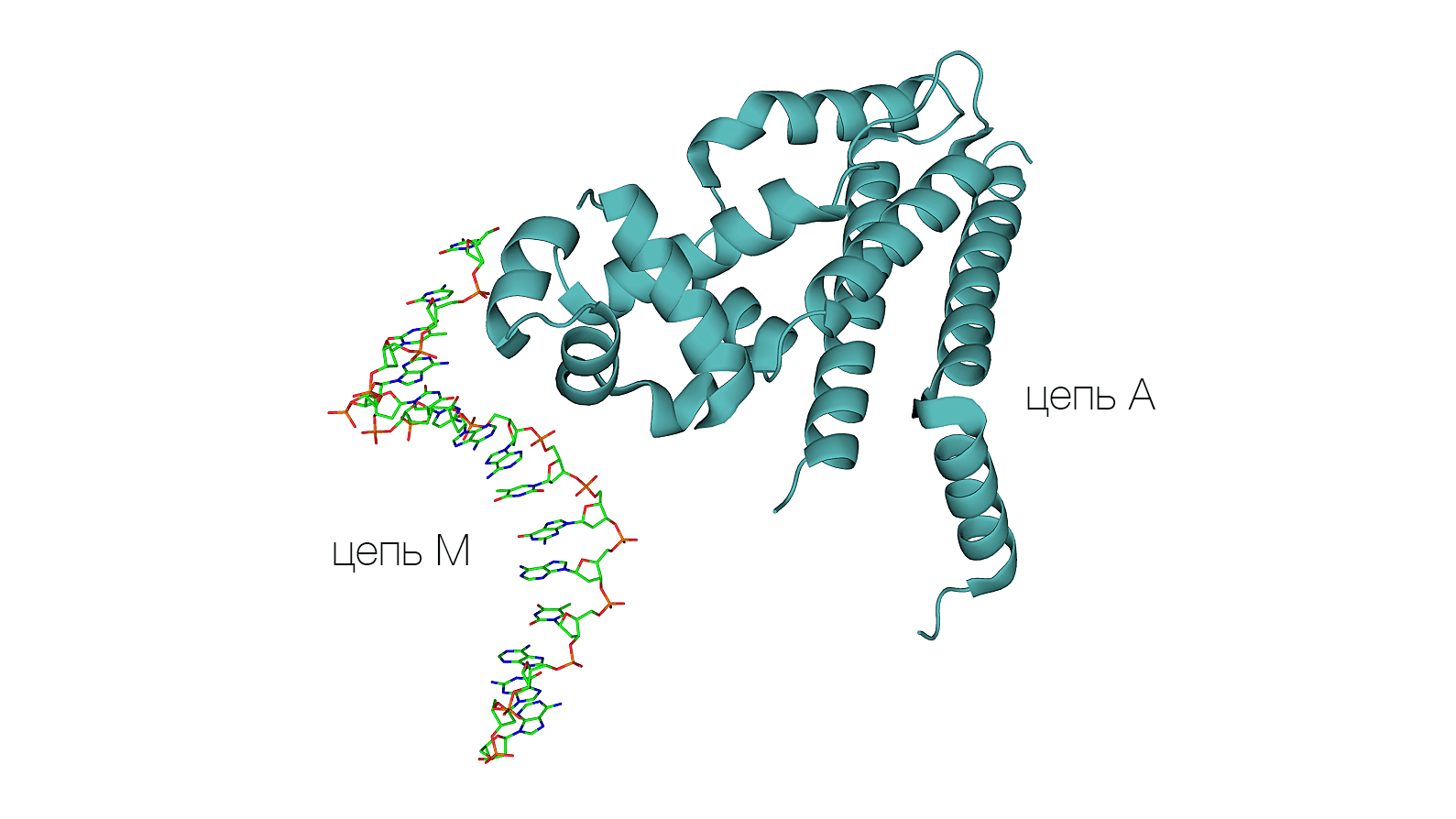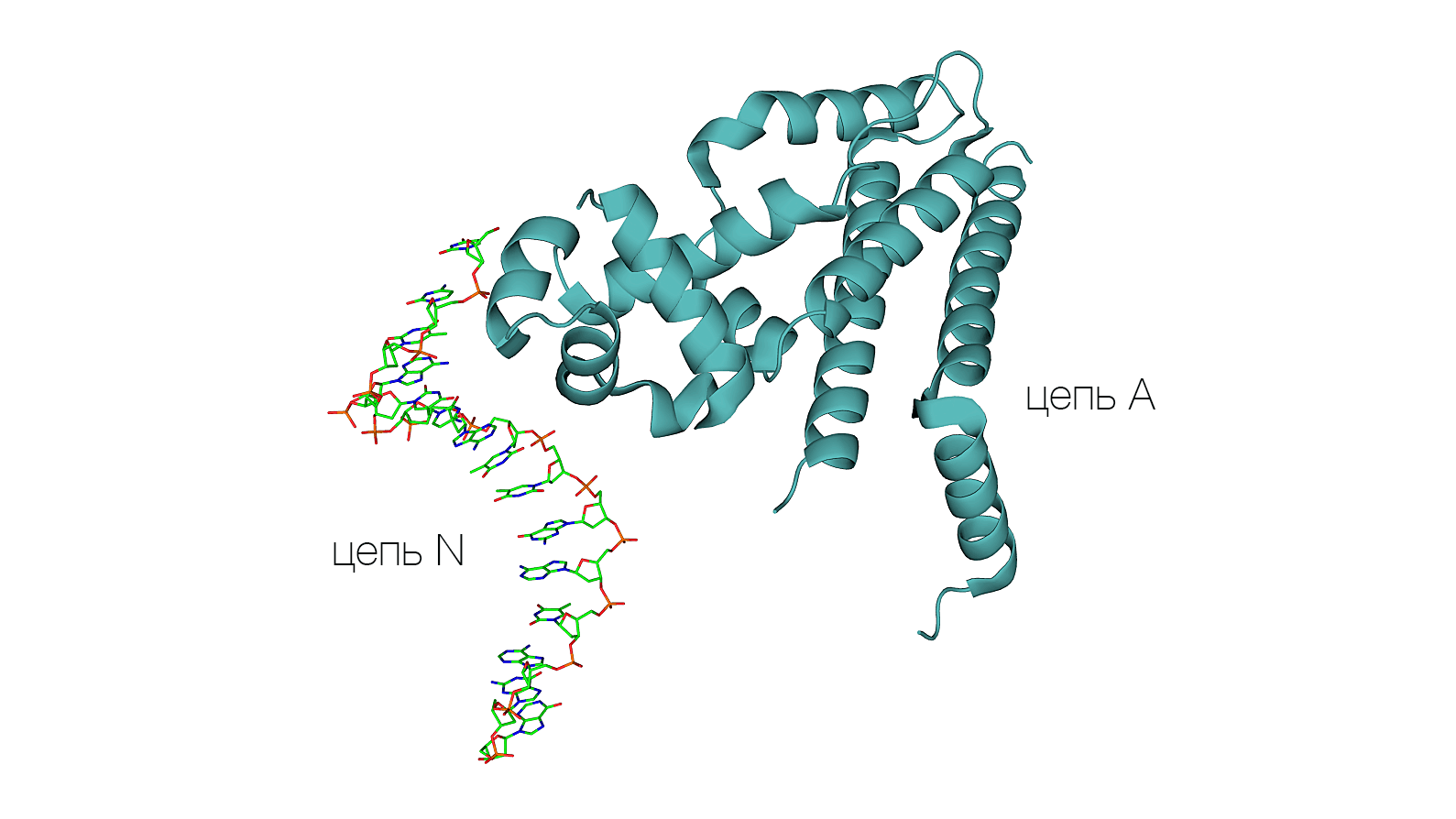
Анализ модели 1QPI
Структура 1QPI была разрешена в 2011 году (2.50 Å). Запись содержит три цепи: цепь A белка и цепи N и M ДНК. При этом цепи N и M обратно комплементарны друг другу и наложены друг на друга в модели структуры.
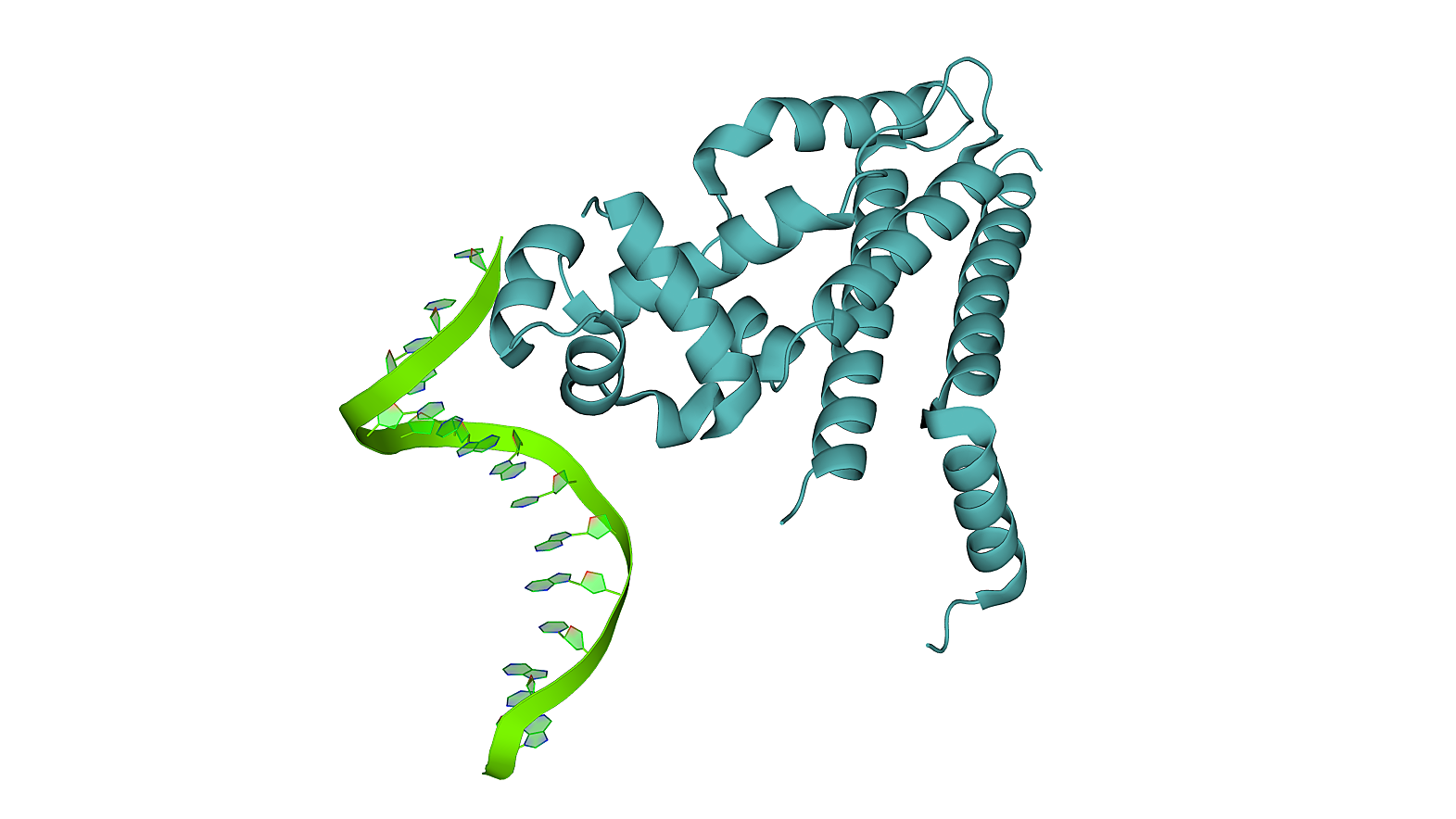
В файле 1qpi.pdb указаны преобразования, которые надо применить к цепям A, N и M, чтобы получить биологическую единицу:
BIOMOLECULE: 1 AUTHOR DETERMINED BIOLOGICAL UNIT: HEXAMERIC APPLY THE FOLLOWING TO CHAINS: M, N, A BIOMT1 1 1.000000 0.000000 0.000000 0.00000 BIOMT2 1 0.000000 1.000000 0.000000 0.00000 BIOMT3 1 0.000000 0.000000 1.000000 0.00000 BIOMT1 2 -1.000000 0.000000 0.000000 0.00000 BIOMT2 2 0.000000 1.000000 0.000000 0.00000 BIOMT3 2 0.000000 0.000000 -1.000000 45.87500
Можно заметить, что предложенное преобразование представляет собой поворот (на 180°) и трансляцию. Совместив две цепи ДНК в асимметрической единице и предложив указанное преобразование, авторы дают возможность получить таким образом биологическую единицу.
Координаты всех атомов (за исключением одного нуклеотида – восьмого в последовательности) в файле 1qpi.pdb полностью совпадают. При этом для атомов разных цепей указаны разные идентификаторы альтернативного положения (A для цепи M и B для цепи N). Соответственно, коэффициент заполнения для этих атомов записан равным 0.5:
# фрагмент файла для цепи M # occupancy
↓
ATOM 16 C6 A DC M 1 10.542 1.492 43.835 0.50 55.36 C
ATOM 17 P A DC M 2 14.633 -1.020 40.201 0.50 73.11 P
ATOM 18 OP1A DC M 2 16.033 -0.973 39.685 0.50 64.14 O
ATOM 19 OP2A DC M 2 13.643 -0.028 39.710 0.50 72.69 O
ATOM 20 O5'A DC M 2 14.025 -2.455 39.873 0.50 70.21 O
# фрагмент файла для цепи N
ATOM 321 C6 B DT N 1 10.542 1.492 43.835 0.50 55.36 C
ATOM 322 P B DC N 2 14.633 -1.020 40.201 0.50 73.11 P
ATOM 323 OP1B DC N 2 16.033 -0.973 39.685 0.50 64.14 O
ATOM 324 OP2B DC N 2 13.643 -0.028 39.710 0.50 72.69 O
ATOM 325 O5'B DC N 2 14.025 -2.455 39.873 0.50 70.21 O
Более того, позиции абсолютно идентичны даже для атомов разных химических элементов крайних остатков цепей:
↓ ↓
ATOM 14 N4 A DC M 1 6.999 1.132 44.427 0.50 41.83 N
... ↓ ↓
ATOM 319 O4 B DT N 1 6.999 1.132 44.427 0.50 41.83 O
↓ ↓
ATOM 300 N6 A DA M 15 -4.411 -1.850 0.149 0.50 67.08 N
... ↓ ↓
ATOM 604 O6 B DG N 15 -4.411 -1.850 0.149 0.50 67.08 O
Таким образом, в записи 1QPI наблюдается наложение двух цепей ДНК с помощью альтернативных конформаций атомов, а возможность восстановления двухцепочечной ДНК предоставлена в виде преобразований для получения биологической единицы. Удачность решения такой формы записи для координат атомов ДНК кажется сомнительной. Файл 1qpi.pdb1 биологической единицы содержит ожидаемую «двухцепочечную» ДНК и две цепи белка. Правда, получается, что цепей ДНК в полученной таким образом структуре четыре (цепи M и N были наложены друг на друга в асимметрической единице, и к ним применили описанное выше преобразование симметрии).
Реальной же картиной, по-видимому, является «нерегулярное» положение цепей ДНК, так как, согласно статье авторов структуры, при взаимодействии исследуемого белка с ДНК важна последовательность CTATCA*TGATAG (и не важны центральный или концевые нуклеотиды); а в этом смысле цепи N и M идентичны.