Учебный сайт
Бредихина Данилы
- VII
- Оценка качества 3D структур
Поиск несоответствий в PDB файлах
Модель 7GPB
7GPB представляет собой модель структуры гомотетрамера гликогенфосфорилазы с разрешением 2.90 Å. Статья об исследовании, в рамках которого была построена данная модель, датируется 1991 годом. Последние изменения были произведены в 2011 году (поле REVDAT в файле .pdb), однако, насколько можно судить из записей в поле Revision History на странице PDB, они не касались каких-либо деталей структуры.
В модели 7GPB внимание на себя обращают, в частности, триптофаны, например Trp67 в цепи D. На изображении ниже различимо сильное нарушение планарности боковой цепи триптофана:
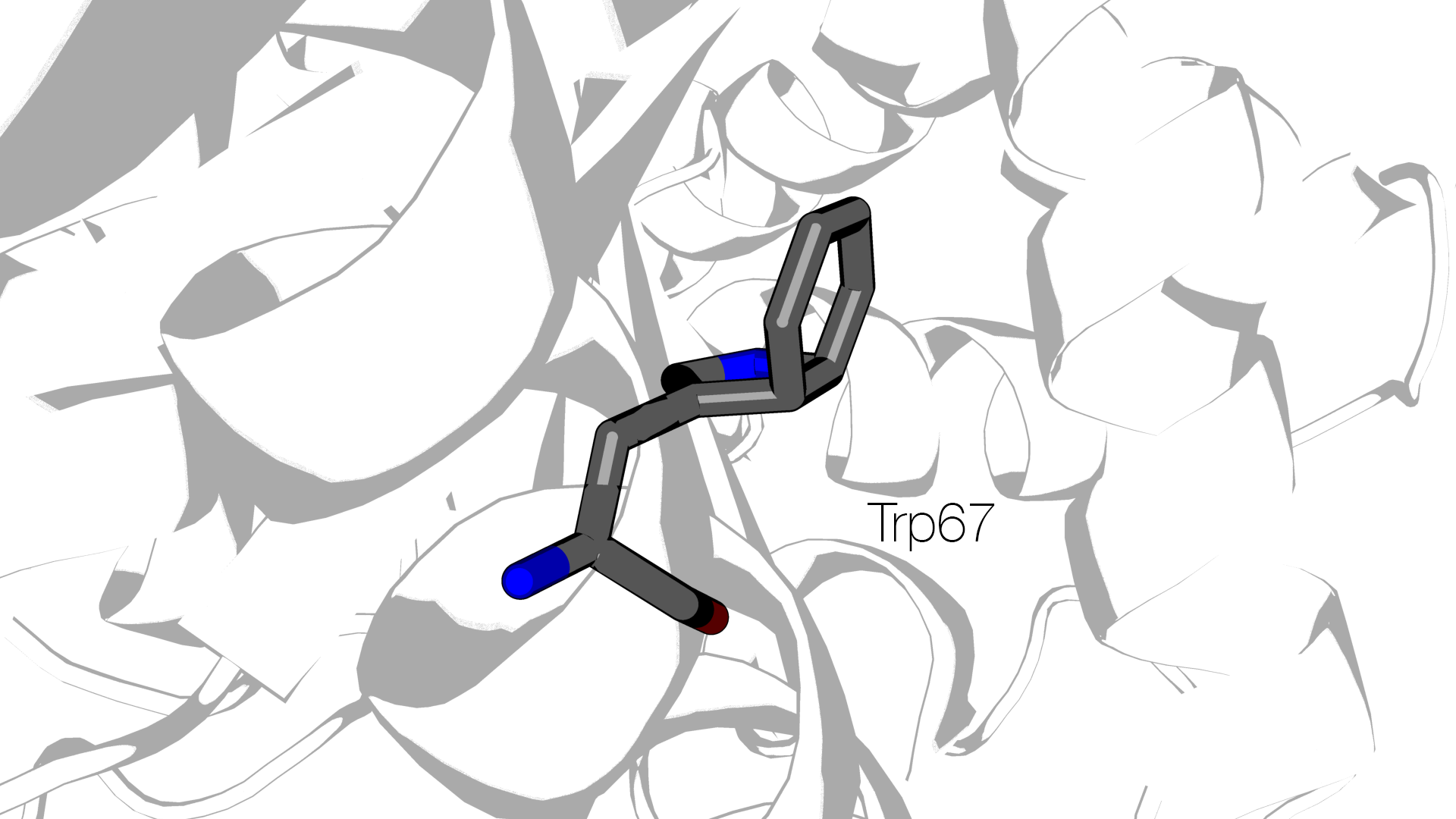
Температурный фактор для атомов бензольного кольца индола у триптофана 67 цепи D значимо выше, чем для, например, триптофана 825 той же цепи (48.97 – 54.15 у Trp67 против 17.33 – 21.80 у Trp825). Можно сказать, что наблюдаемый случай – пример противоречия законам химии, допущенного при создании модели.
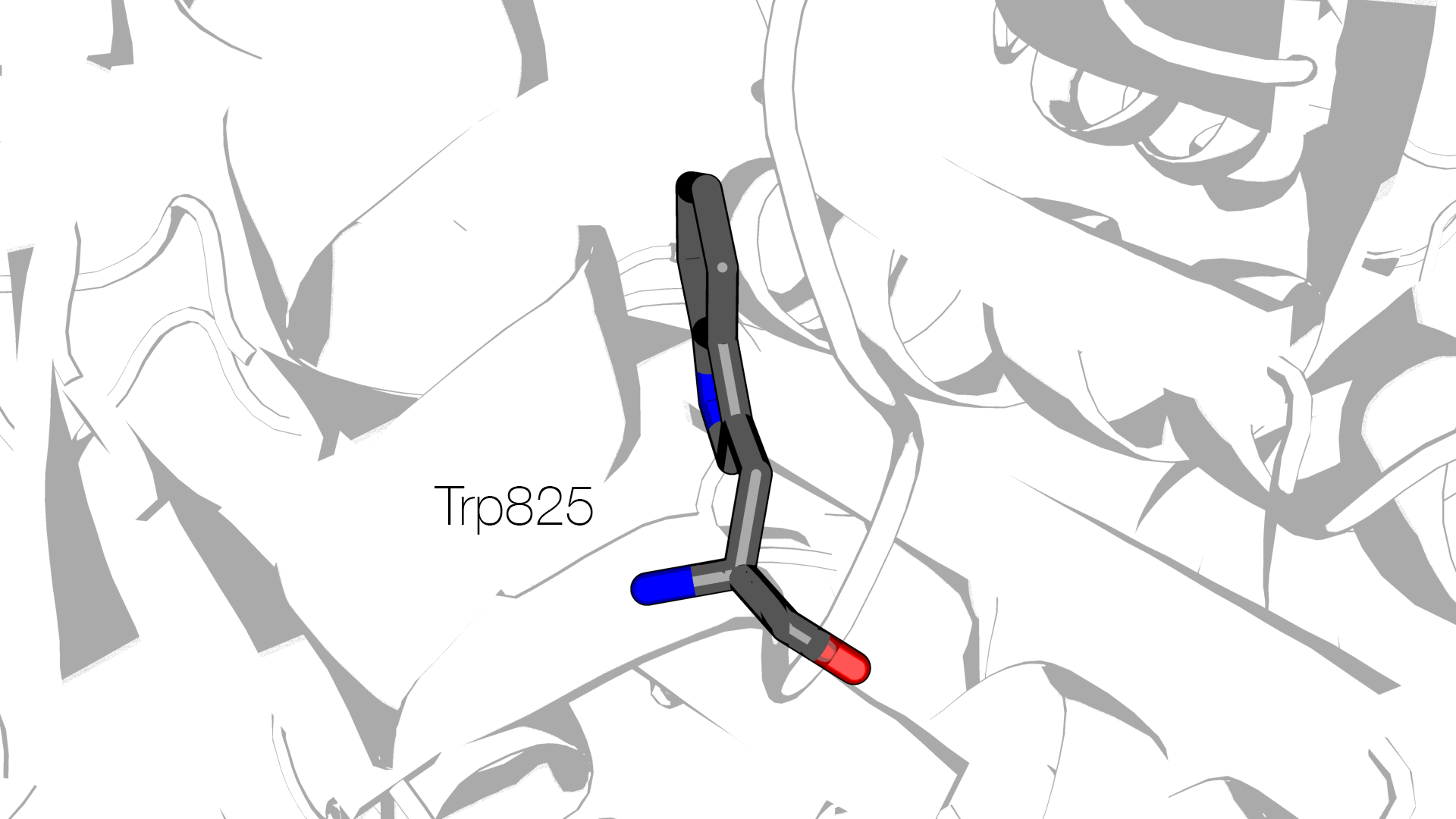
Стоит отметить, что в отчёте для 7GPB в базе PDBREPORT присутствует ошибка Side chain planarity problems, в которой на первой позиции обнаруживается как раз Trp67 цепи D.
Модель 5RXN
5RXN представляет собой модель структуры рубредоксина Clostridium pasteurianum с разрешением 1.20 Å. Структура была расшифрована в 1980-е годы. В структуре модели можно обнаружить два диастереомера треонина – (2S, 3R) и (2S, 3S), лишь первый из которых типичен для белков:
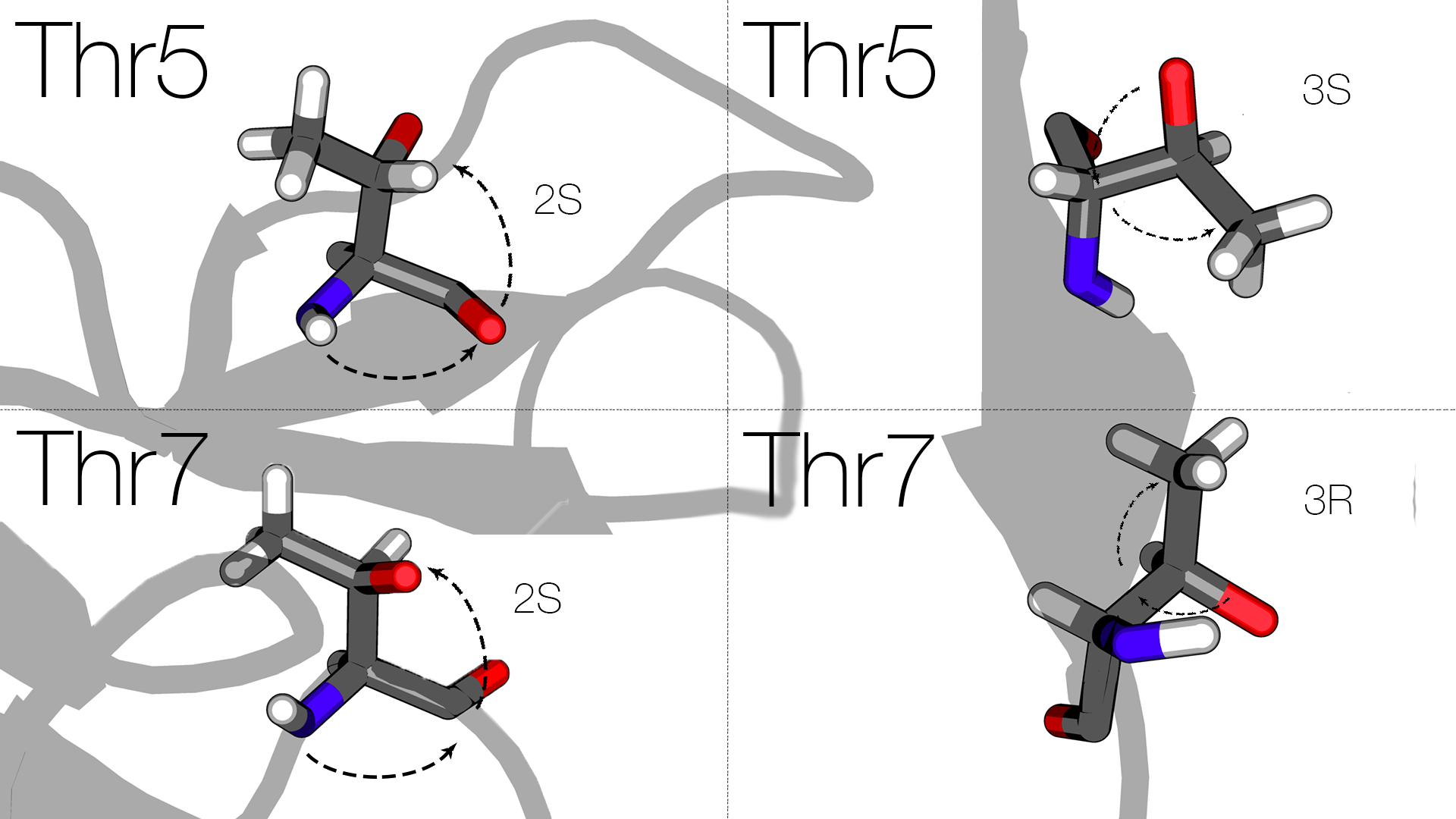
Температурные факторы для атомов боковой цепи Thr5 [(2S, 3S)] имеют более высокие значения, нежели, например, для Thr7 или Thr28 [оба (2S, 3R)]. (Так, для атома HB у Thr5 температурный фактор имеет значение 27.00, в то время как у Thr7 и Thr28 – 15.60 и 15.00, соответственно).
Отчёт в PDBREPORT для 5RXN содержит следующую ошибку:
Error: Threonine nomenclature problem The threonine residues listed in the table below have their O-gamma-1 and C-gamma-2 swapped. 5 THR ( 5-) A
Таким образом, можно сделать заключение о необоснованном выборе стереоизомерии (2S, 3S) для остатка Thr5 в структуре 5RXN.
Помимо треонина, хиральностью обладает (из числа протеиногенных аминокислот) изолейцин. (Треонин и изолейцин относятся к так называемым C-β branched аминокислотам, к которым также принадлежит валин).
Модель 1GT0
Модель 1GT0 представляет собой комплекс ДНК с доменами POU и HMG транскрипционных факторов. Эта структура расшифрована в 2003 году, последние исправления датируются ноябрём 2012 года. Разрешение – 2.60 Å. В цепи C (домен POU) отсутствуют аминокислоты с 78 по 96.
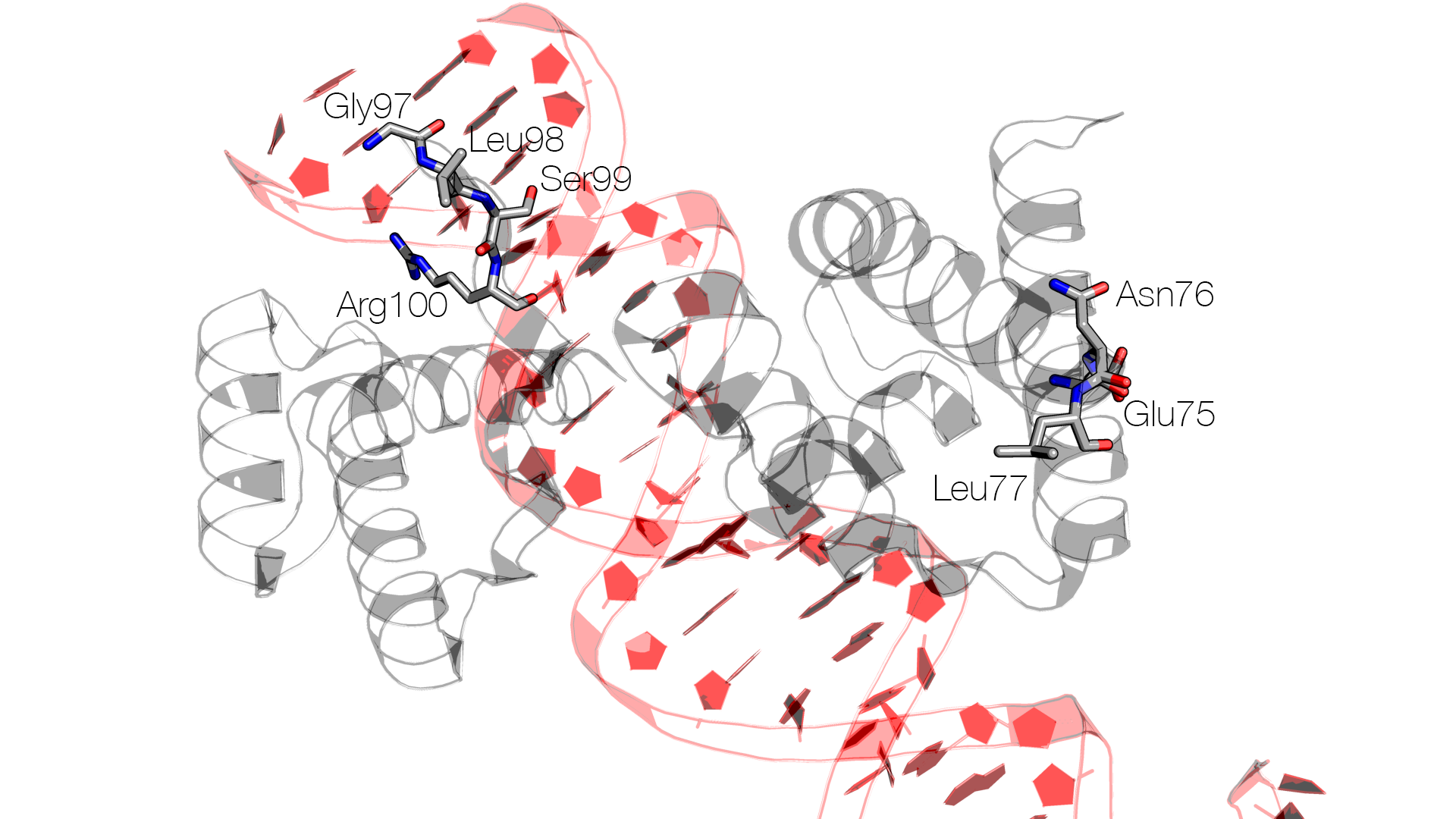
Температурные факторы для остатков 75 - 77 и 97 – 100 (по «краям» разрыва) имеют высокие значения (от 80.17 до 97.70). По визуализации электронной плотности создаётся впечатление случайного выбора положения боковой цепи Arg100:
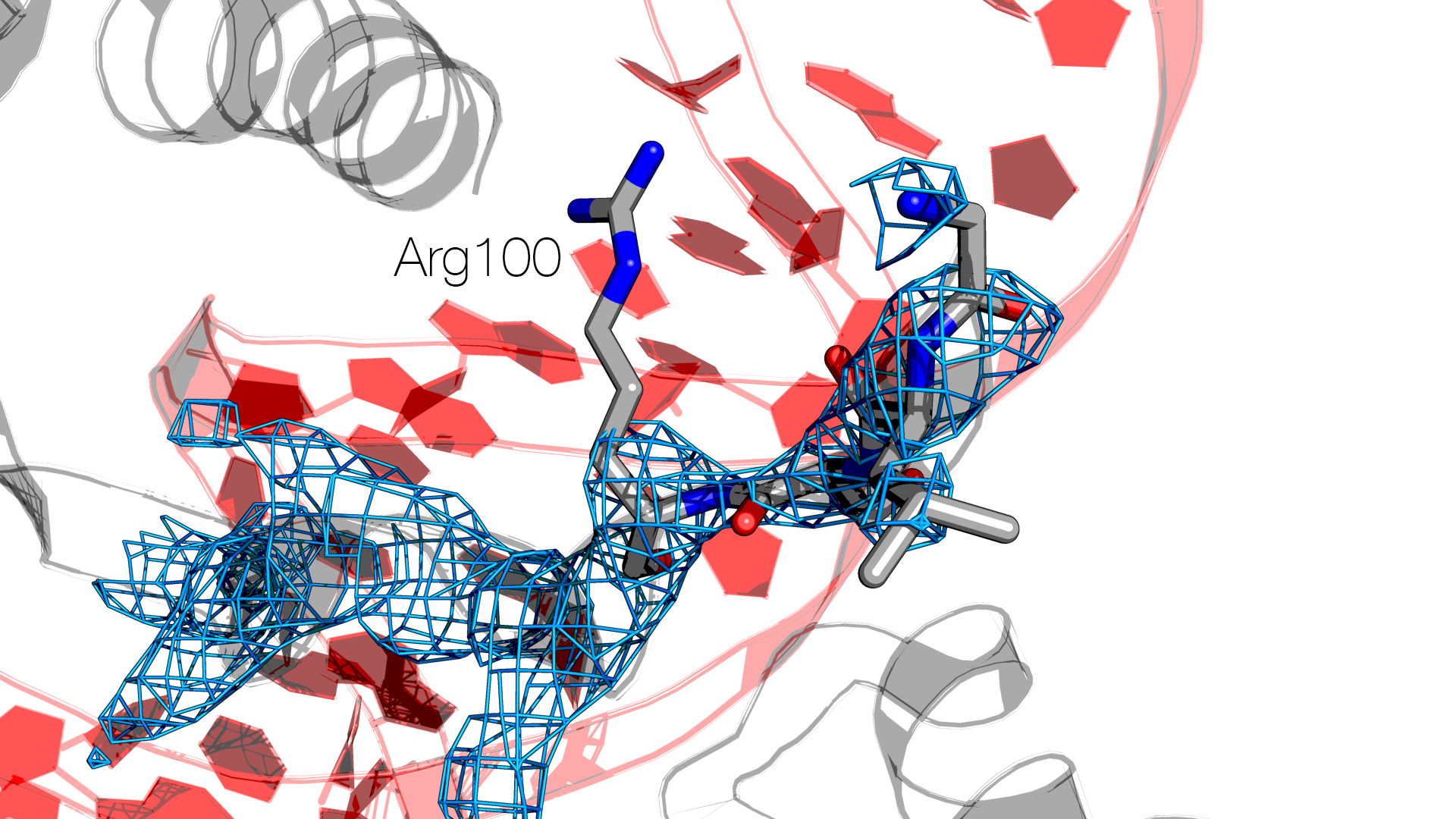
Модель 2B5A
Структура 2B5A разрешена в 2005 году (последнее редактирование – в 2011) с разрешением 1.54 Å. В цепи C этой модели остаток Gln21 имеет две альтернативные конформации. Это видно на изображениях ниже:
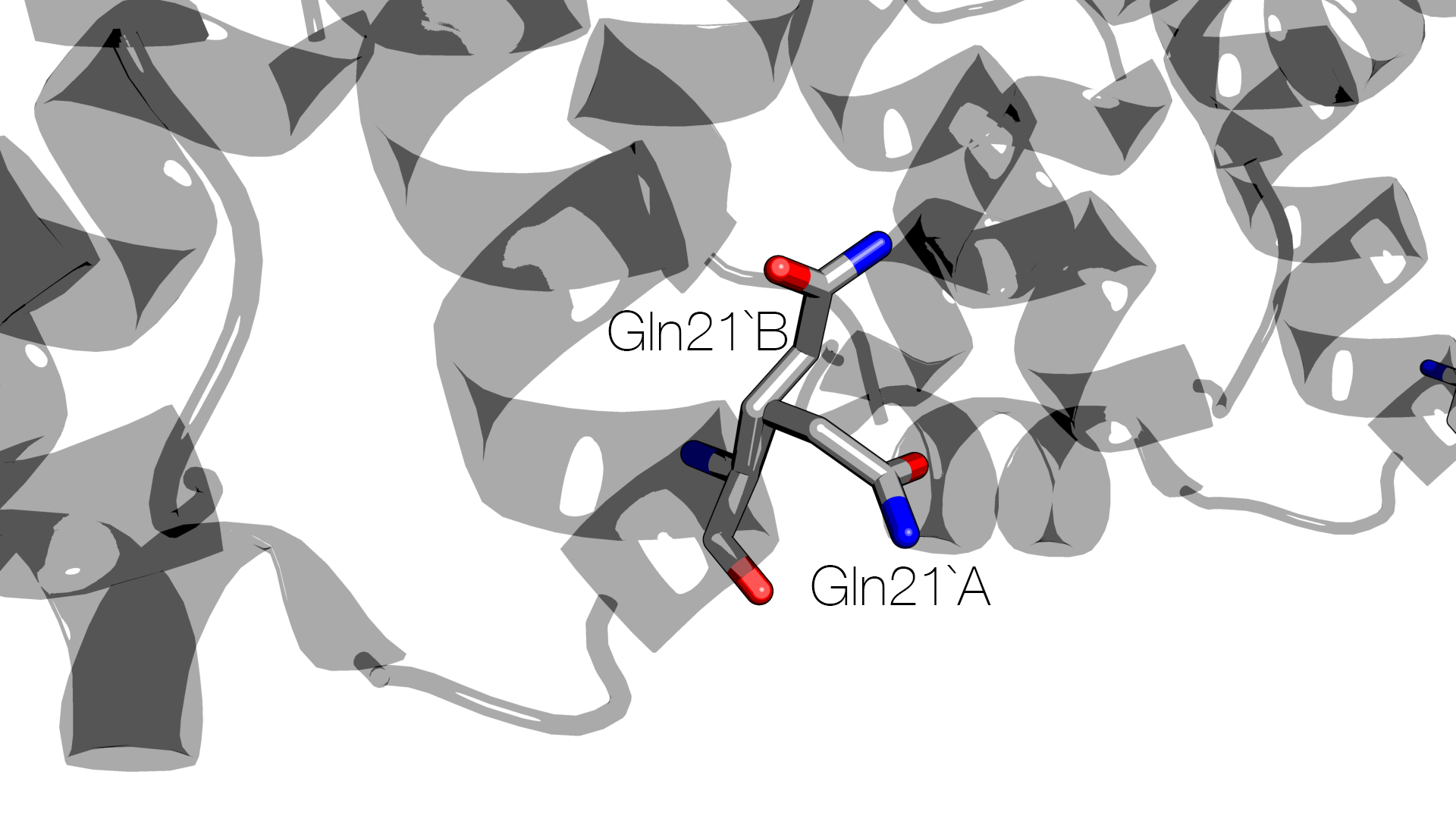
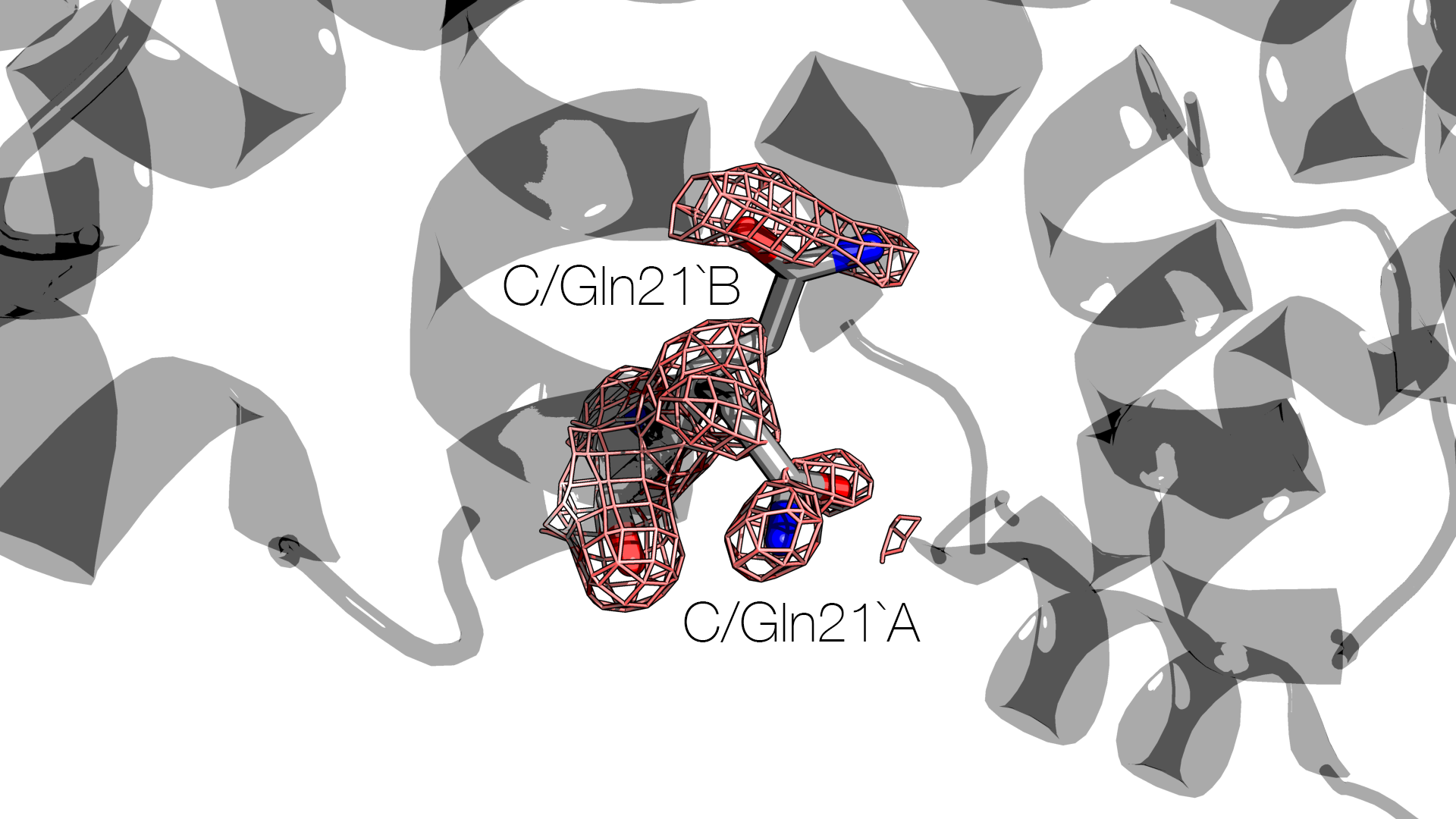
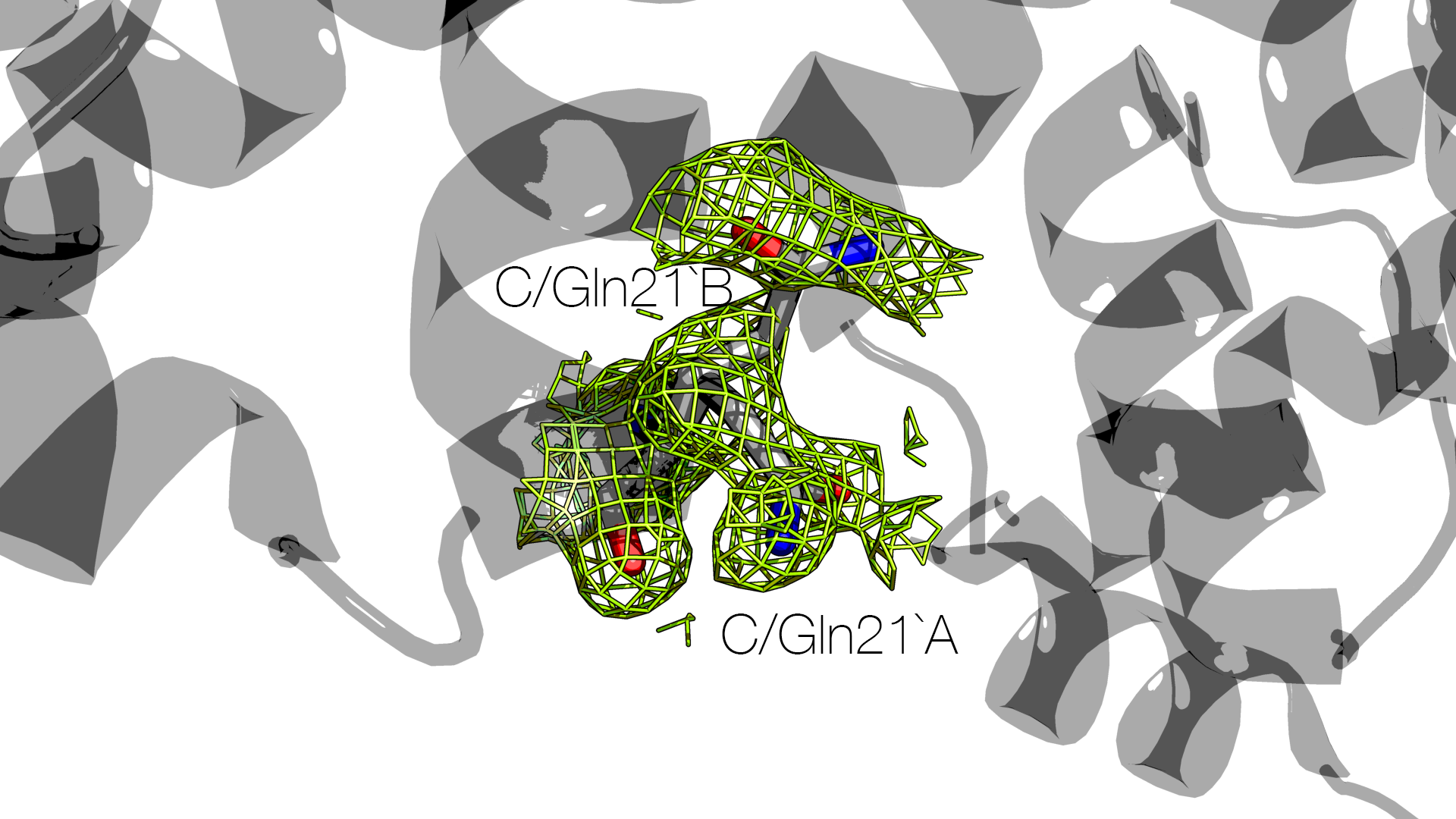
Для сравнения – изображения для Gln21 цепи A:
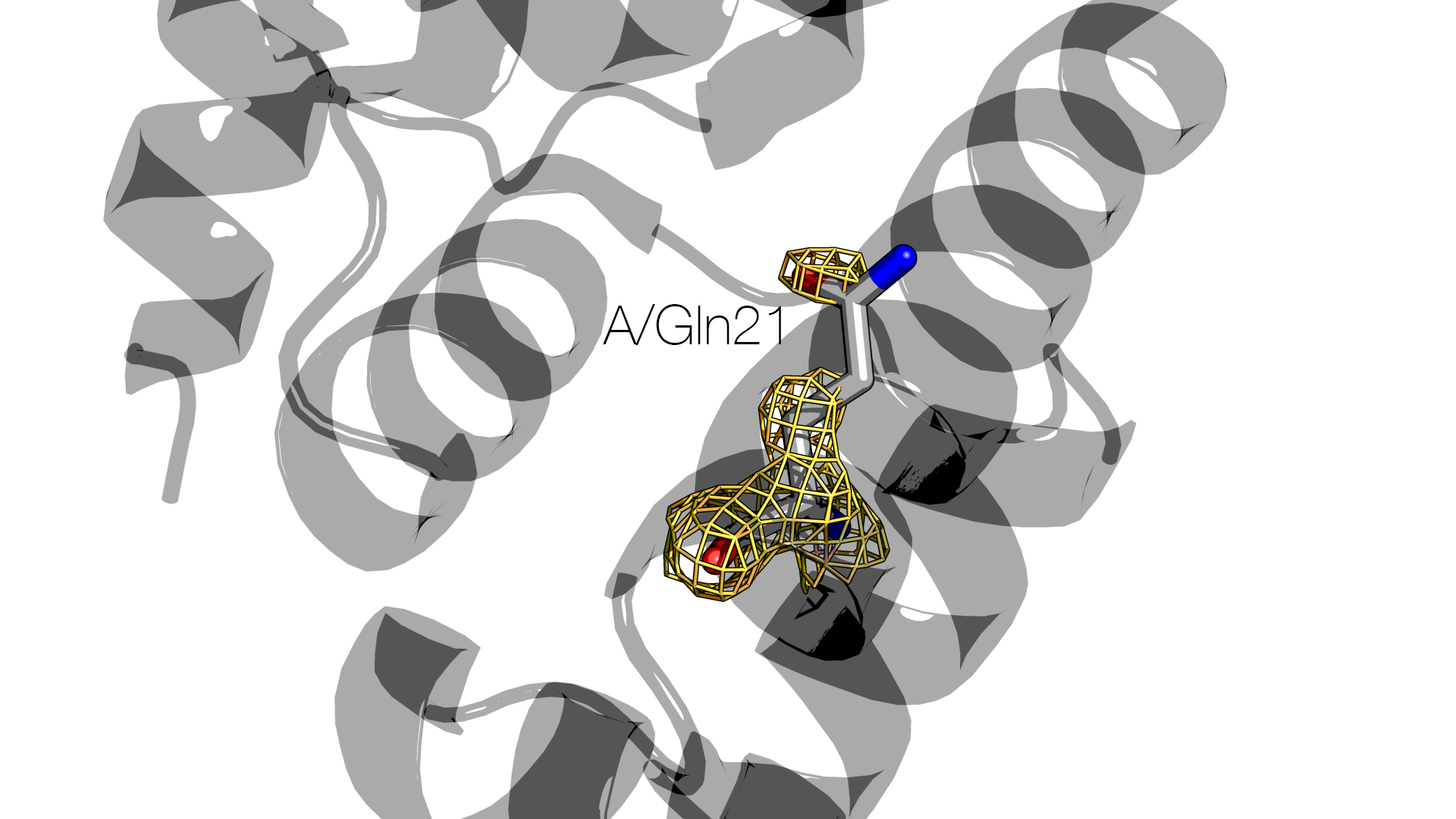
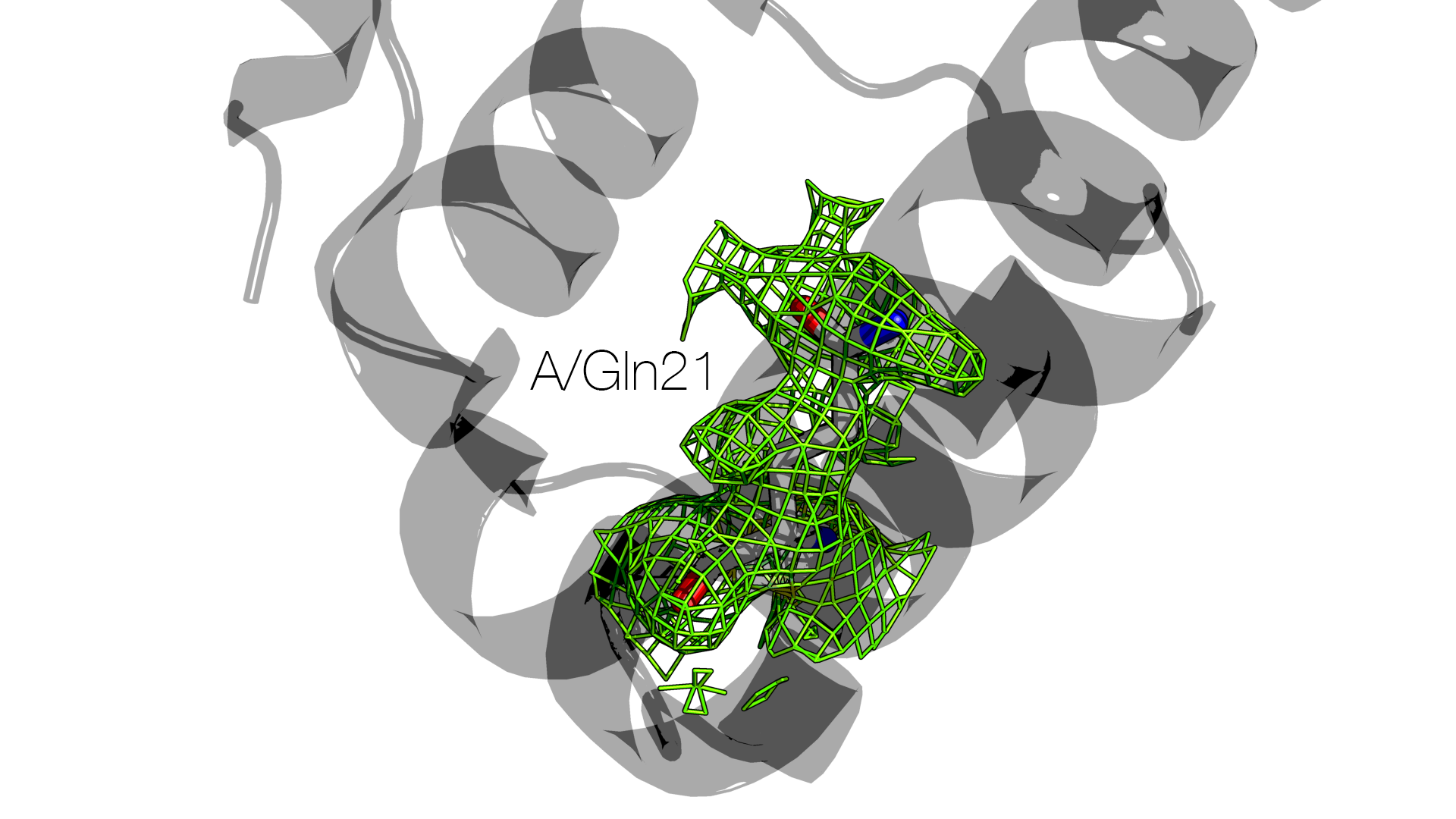
В файле .pdb альтернативные конформации Gln21 цепи C обозначены следующим образом (альтеркод A или B перед названием остатка):
... ATOM 1409 CB AGLN C 21 17.931 32.039 33.379 0.50 20.64 C ATOM 1410 CB BGLN C 21 17.973 31.950 33.643 0.50 22.06 C ATOM 1411 CG AGLN C 21 17.034 33.216 33.781 0.50 20.74 C ATOM 1412 CG BGLN C 21 16.993 31.438 32.609 0.50 23.71 C ...
Остатки Gln21 всех четырёх цепей (A, B, C и D) перечислены в отчёте для модели 2B5A в базе PDBREPORT как имеющие необычное пространственное окружение (an unusual packing environment; для вычисления среднего окружения используются остатки того же типа в «хороших» PDB файлах).
Модель 1RC2
Модель аквапорина Z 1RC2 с разрешением 2.50 Å была получена в 2003; последние исправления датируются 2011 годом. В файле PDB можно обнаружить молекулы воды, не связанные с белком. Так, в файле .pdb приведён список молекул растворителя, которые расположены на расстоянии более 5 Å от ближайшей белковой цепи. Атомы кислорода этих молекул показаны на следующем изображении:
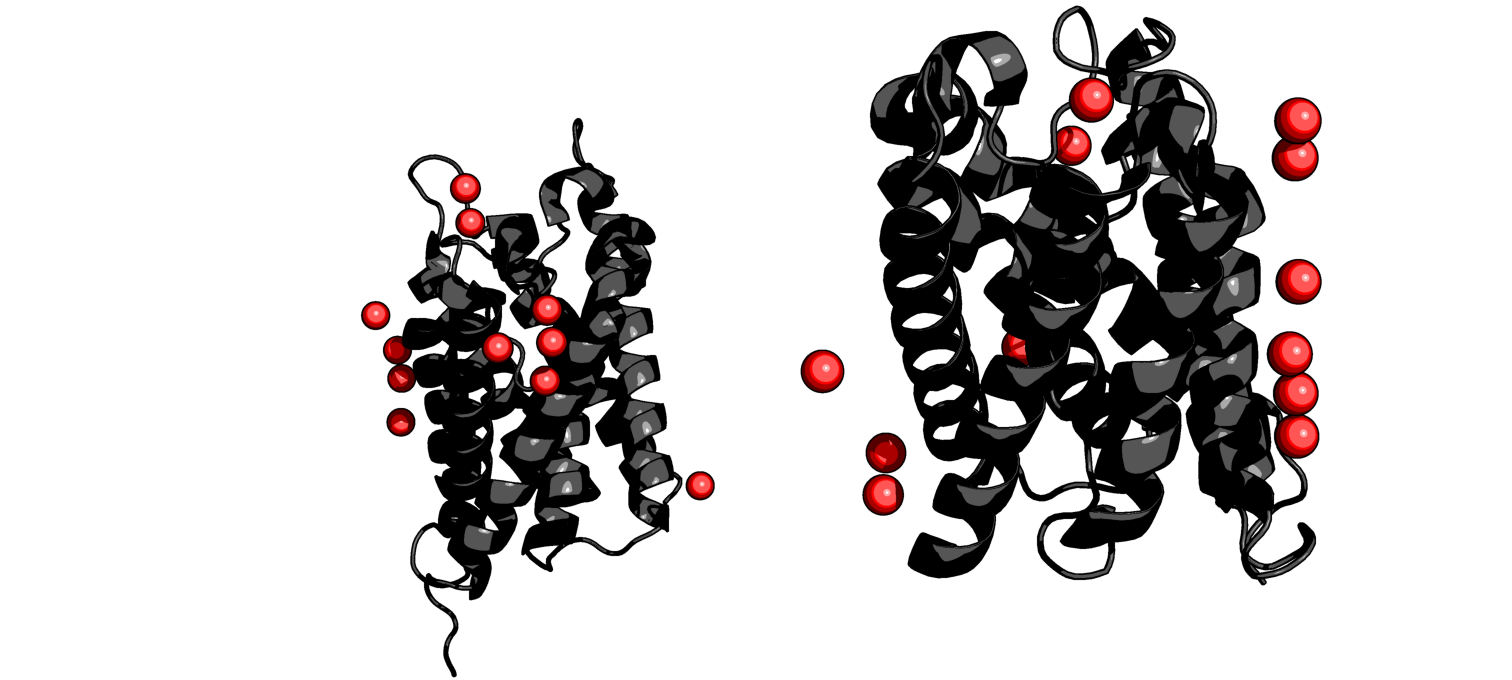
Стоит отметить также наличие в модели 1RC2 большого числа атомов (для 43 остатков из 462) со значениями температурного фактора >100. Как указано в отчёте для модели 1RC2 в базе PDBREPORT, B-factors above 100.0 are a good indicator that the location of that atom is meaningless. Этот отчёт также содержит список из 16 молекул воды, не участвующих в формировании водородных связей (Water molecules without hydrogen bonds); для 118 остатков конформация остова «редкая» (Unusual backbone conformations), что порождает сомнения в точности модели.
Модель 1DLP
Структура гомогексамера 1DLP с разрешением 3.30 Å была расшифрована в 2000 году. В файле .pdb можно обнаружить атомы с коэффициентом заполнения, равным 0 (эти атомы перечислены в файле разделах MISSING ATOM и ZERO OCCUPANCY ATOM), например:
... # occupancy
↓
ATOM 1004 CG ASN A 136 58.627 90.110 34.403 0.00 20.00 C
ATOM 1005 OD1 ASN A 136 58.461 89.077 33.787 0.00 20.00 O
ATOM 1006 ND2 ASN A 136 58.300 90.272 35.704 0.00 20.00 N
...
Для этого остатка – Asn136 цепи A – очевидно нарушена его химическая структура:
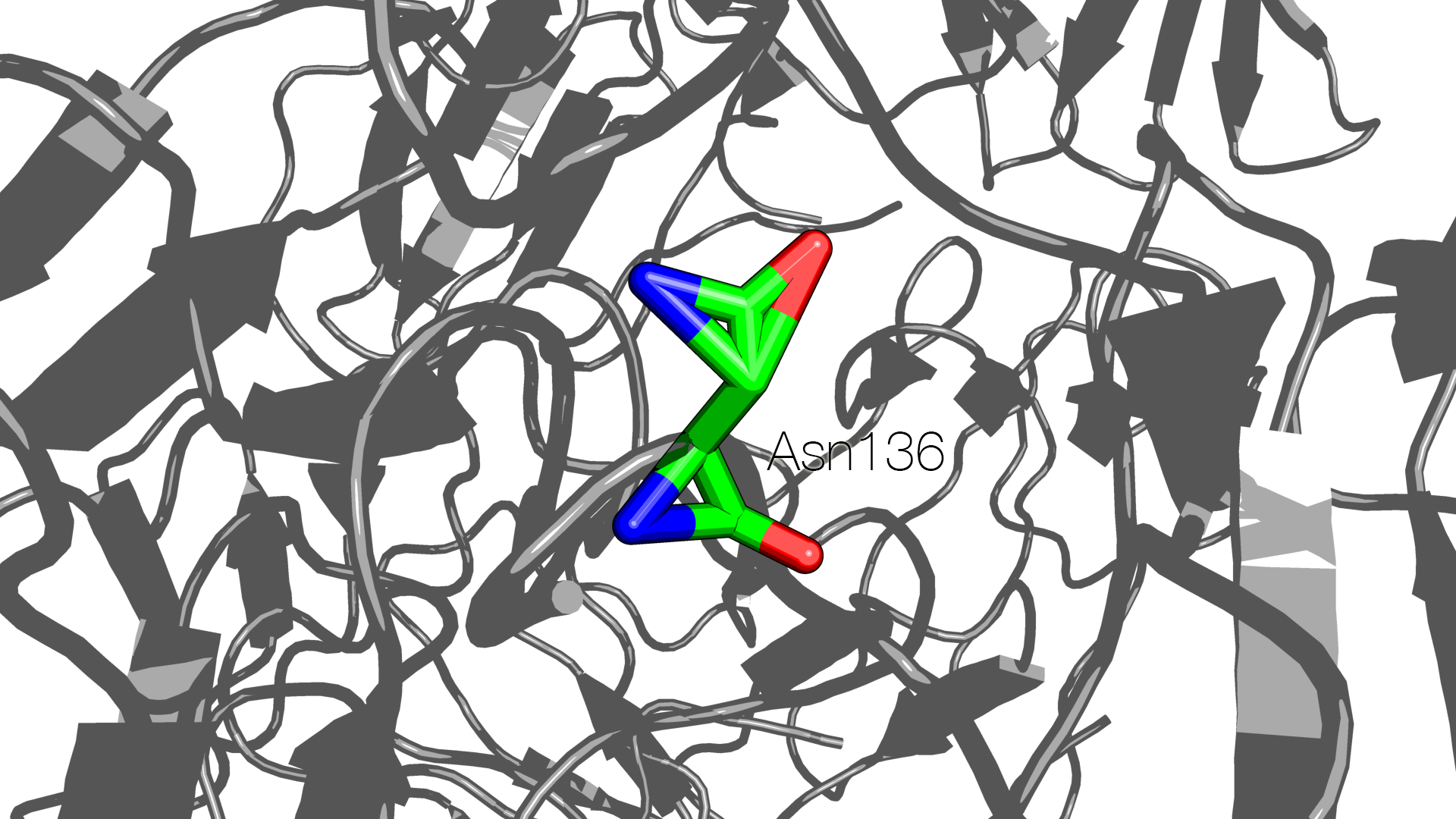
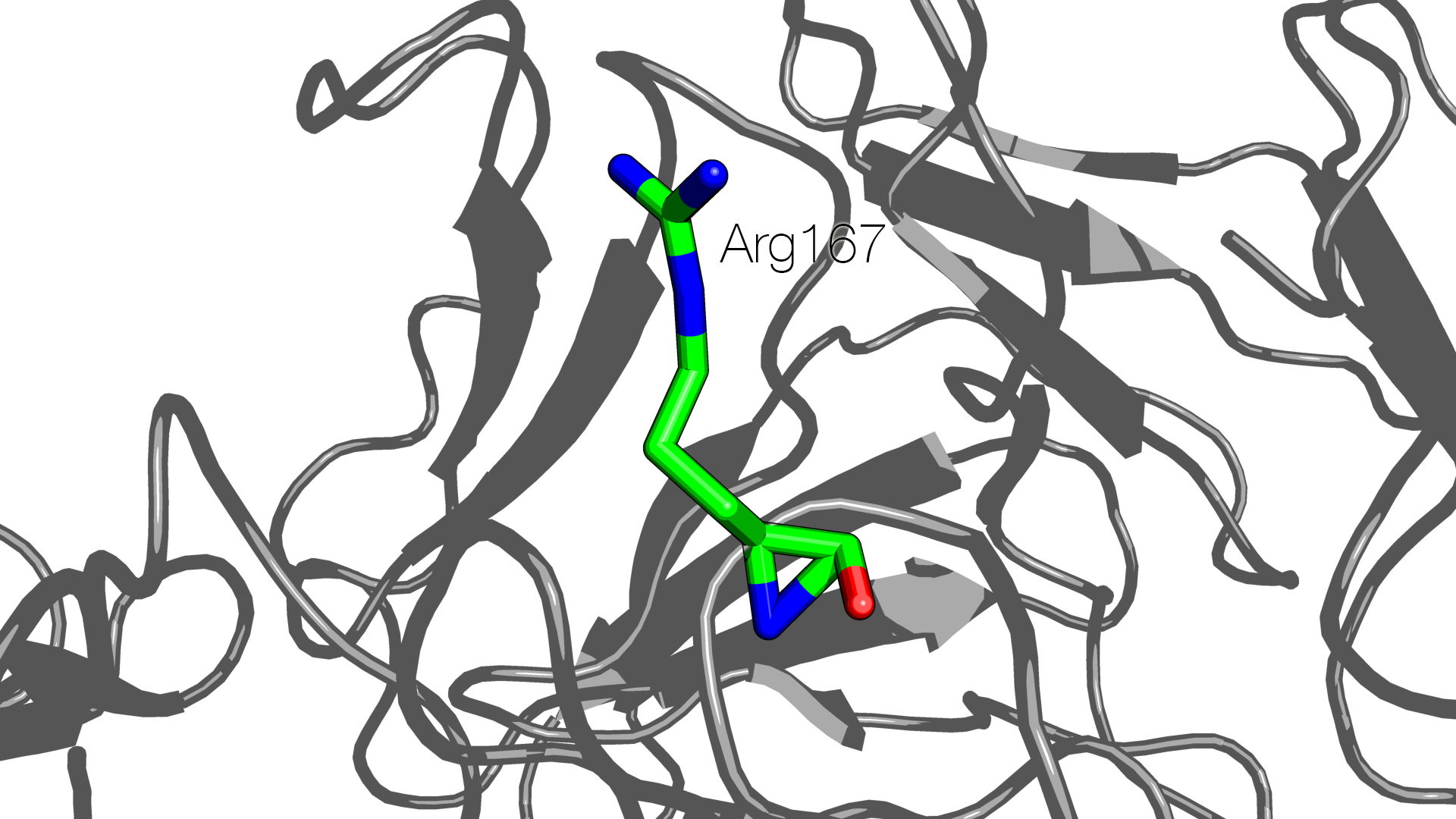
Похожая ситуация наблюдается, например, для Arg167 цепи C:
... # occupancy
↓
ATOM 4716 CB ARG C 167 80.188 77.637 46.951 0.00 20.00 C
ATOM 4717 CG ARG C 167 81.160 77.095 45.943 0.00 20.00 C
ATOM 4718 CD ARG C 167 80.497 76.065 45.054 0.00 20.00 C
ATOM 4719 NE ARG C 167 80.793 74.648 44.964 0.00 20.00 N
ATOM 4720 CZ ARG C 167 81.179 73.412 44.666 0.00 20.00 C
ATOM 4721 NH1 ARG C 167 81.808 73.151 43.492 0.00 20.00 N
ATOM 4722 NH2 ARG C 167 80.921 72.404 45.532 0.00 20.00 N
...
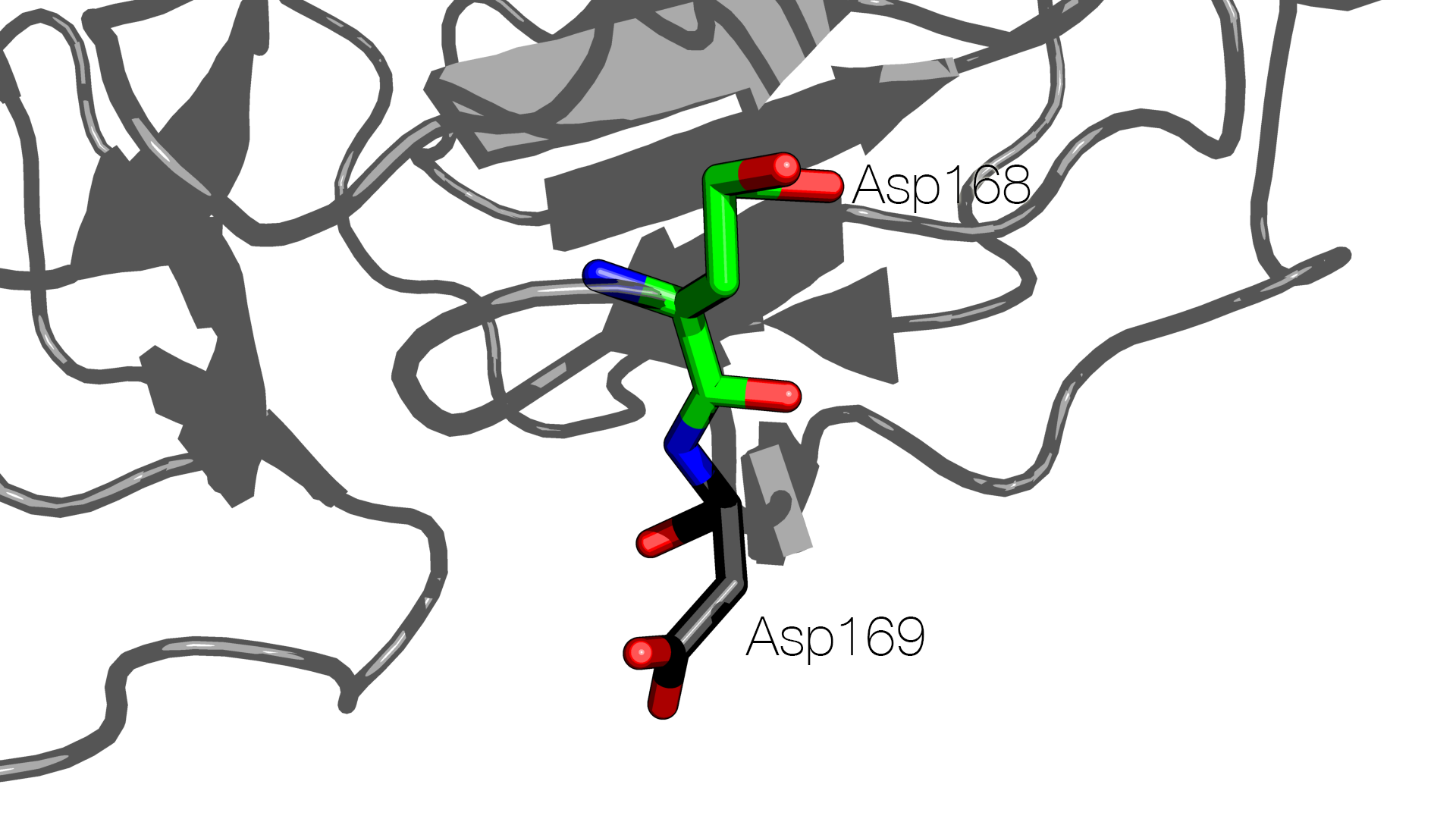
Подобных ситуаций в модели 1DLP можно найти достаточное количество. В качестве очередного и последнего примера – Asp168 цепи C с неправильным, с химической точки зрения, расположением атомов Oδ1 и Oδ2 (соседний Asp169 приведён на изображении ниже для сравнения):
... # occupancy
↓
ATOM 4729 OD1 ASP C 168 73.065 77.542 45.924 0.00 20.00 O
ATOM 4730 OD2 ASP C 168 73.949 75.666 45.211 0.00 20.00 O
...
Положение 59 остатков в эксперименте определить не удалось (MISSING RESIDUES). Большое число остатков на картах Рамачандрана в отчёте в базе PDBREPORT расположено вне предпочитаемых и разрешённых участков. Как указано в отчёте, if too many plus- signs fall outside the contoured areas then the molecule is poorly refined (or worse). Для 167 остатков конформация полипептидного остова модели является редкой (Unusual backbone conformations).
B-фактор и occupancy при разных разрешениях
С помощью Advanced search на сайте PDB был произведён поиск структур разрешения до 1 Å и от 2.0 Å до 2.5 Å (метод получения – X-RAY). Для каждой из двух групп была выбрана модель: 1US0 (0.66 Å) и 4R6V (2.35 Å). Обе структуры являются мономерами; длина полипептидной цепи составляет 316 остатков и 323 остатка, соответственно.
Извлечь коэффициенты заполнения (occupancy) и температурные факторы (B-факторы) из файлов можно, например, следующим образом:
# 55 - 60 Occupancy. cat 4r6v.pdb | egrep ^ATOM | cut -c55-60 > 4r6v.occ cat 1us0.pdb | egrep ^ATOM | cut -c55-60 > 1us0.occ # 61 - 66 Temperature factor. cat 4r6v.pdb | egrep ^ATOM | cut -c61-66 > 4r6v.b cat 1us0.pdb | egrep ^ATOM | cut -c61-66 > 1us0.b
Выполнить подсчёт значений и визуализировать полученные данные можно с помощью скрипта:
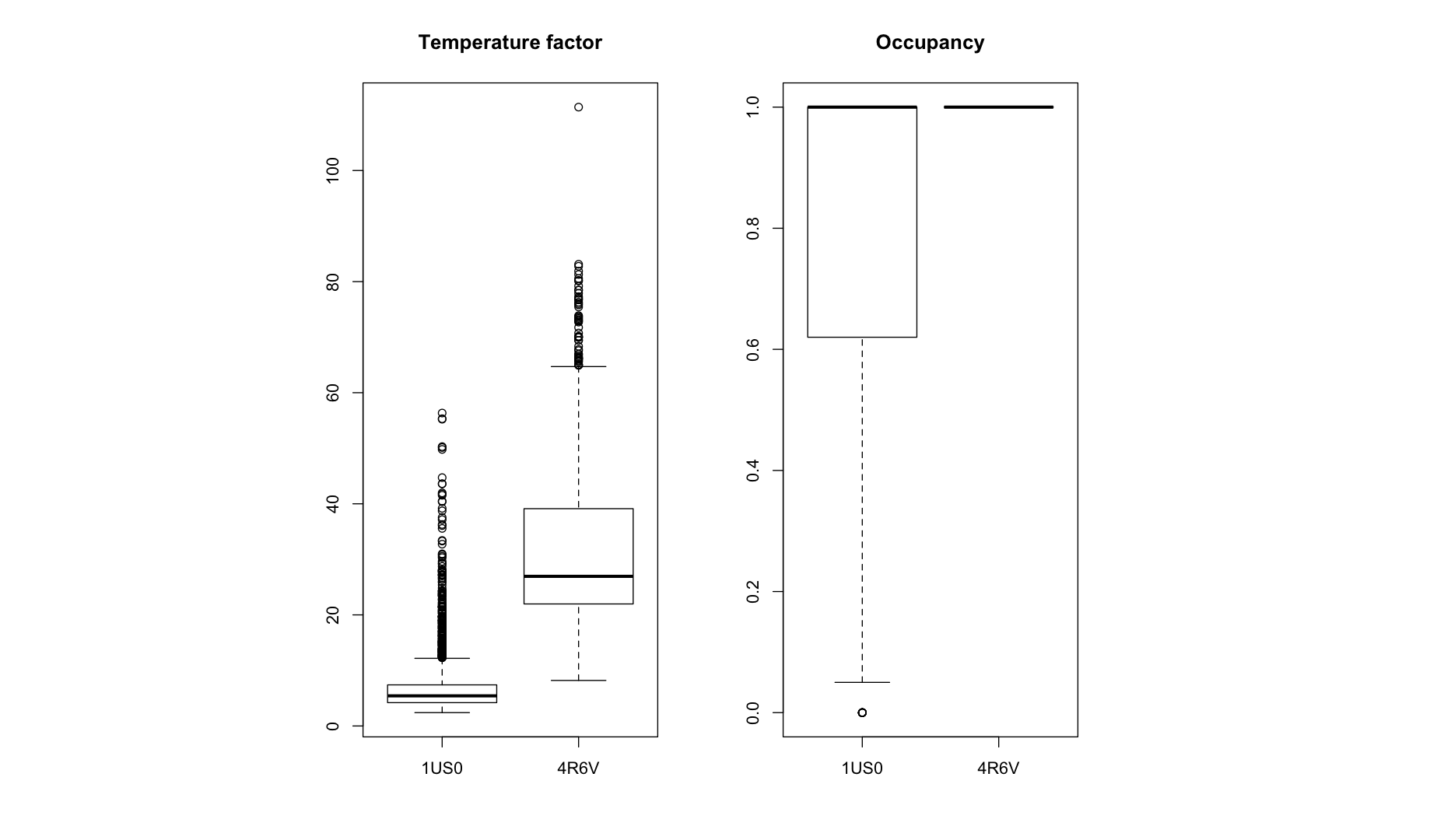
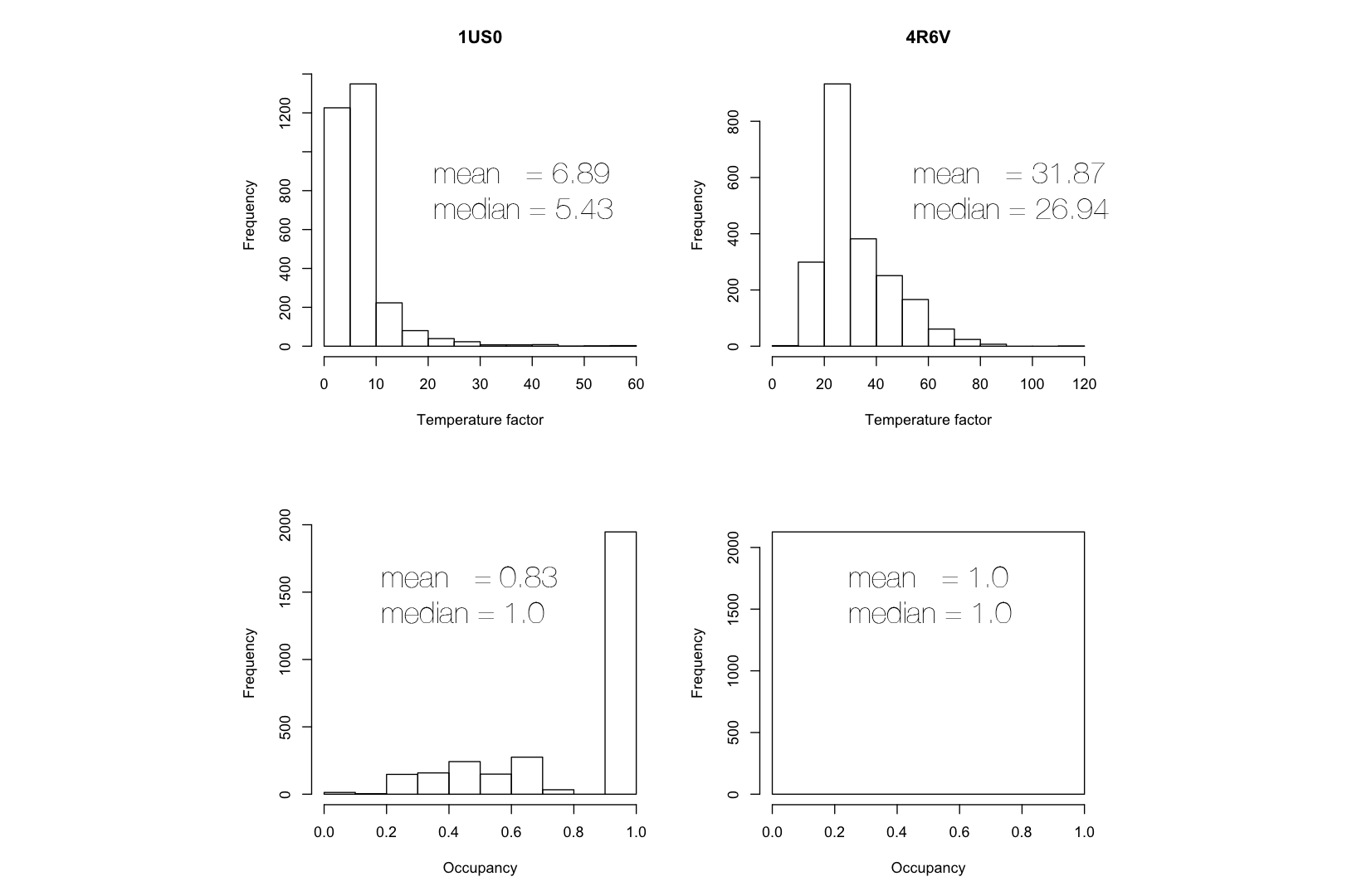
Как видно, температурные факторы для менее точной модели структуры в среднем имеют более высокие значения. При этом если для менее точной модели структуры коэффициенты заполнения всех атомов равны единице, то с увеличением точности проявляется неоднозначность положения отдельных атомов и появляются коэффициенты заполнения меньше единицы.
(Так, в 1US0.pdb 472 атома имеют по 2 альтернативных положения, для ещё двух атомов указано наличие трёх альтернативных положений; 11 атомов в этой модели имеют коэффициенты заполнения 0.00, при этом они не имеют альтернативных положений.)

Ссылки и файлы
- PDB файлы: 7GPB, 5RXN, 1GT0, 2B5A, 1RC2, 1DLP
- Скрипты для 7GPB, 5RXN, 1GT0, 2B5A, 1RC2, 1DLP
- Файл 2b5a.omap
- Файл 1gt0.omap
- База данных PDBREPORT
- Файл 1us0.pdb
- Файл 4r6v.pdb
- Скрипт для сравнения данных