Учебный сайт
Бредихина Данилы
- VII
- Дополнительные задания
Определение вторичной структуры
Программы DSSP и Stride
Для определения вторичной структуры белка 1UWZ были использованы программы DSSP и Stride. Границы спиралей и тяжей в выдаче программ (DSSP, Stride) оказалась близки к таковым, указанным в pdb-файле модели структуры 1UWZ. Изображение последних было получено с использованием следующих команд и приведено ниже (для цепи A белка):
set cartoon_discrete_colors, 1 color gray color red, ss h color lime, ss s
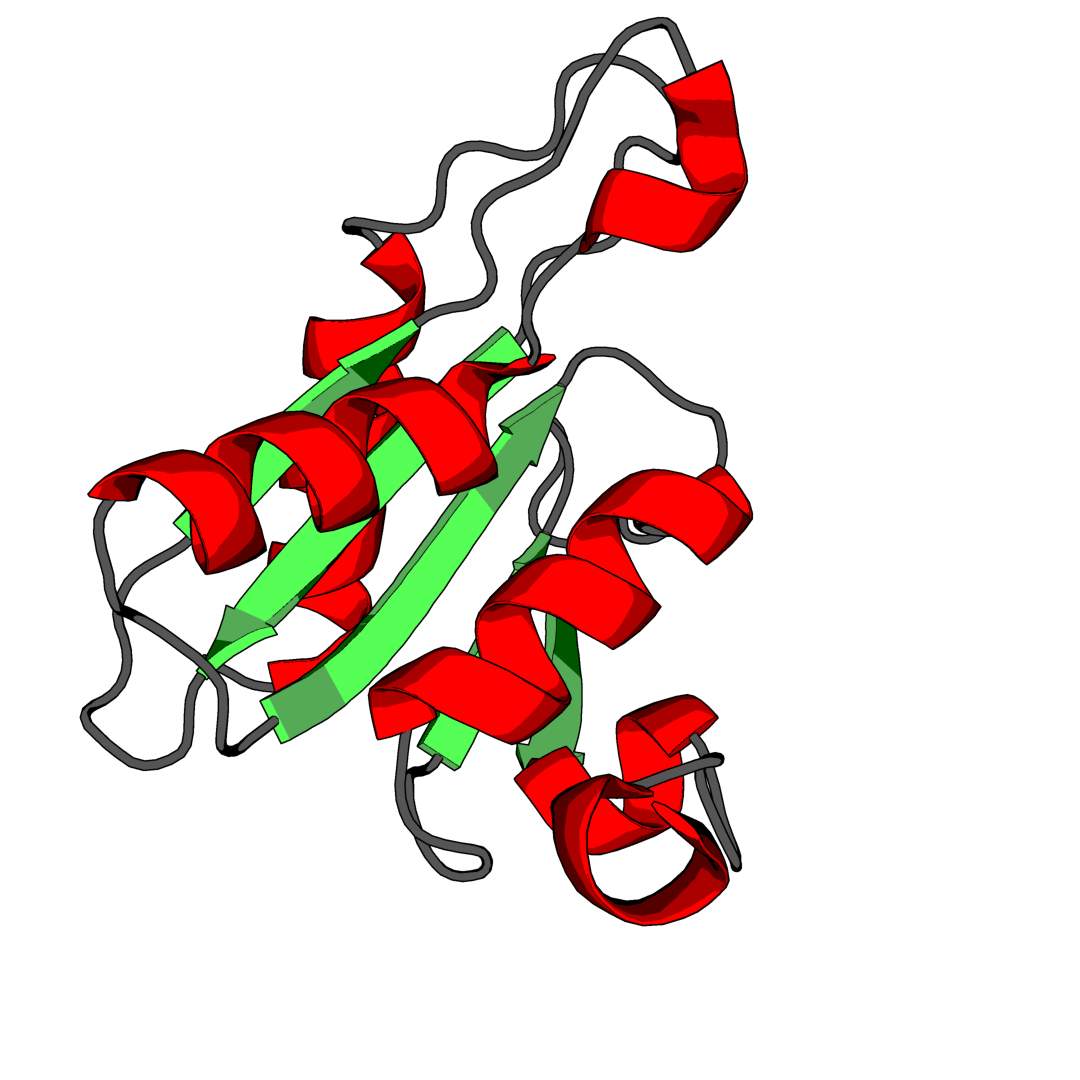
Программа DSSP определила границы α-спиралей в структуре следующим образом (для цепи A; для цепи B результаты аналогичны): 3 – 14, 54 – 65, 87 – 96, 117 – 120 (приведены номера аминокислотных остатков, обозначающих границы элемента). Результаты программы Stride отличаются лишь в границе одной спирали: 3 – 15, 54 – 65, 87 – 96, 117 – 120. Полученные границы можно сравнить с границами аннотации вторичной структуры в pdb-файле: 2 – 15, 53 – 65, 86 - 97, 117 – 121.
Аналогичное сравнение можно проделать для определённых β-тяжей. Соответствующие границы приведены в таблице 1.
Видно, что границы β-тяжей, определённые программами, в точности соответствуют таковым в pdb-файле, а предсказание границ α-спиралей меньше соответствует pdb-файлу. При этом алгоритмы DSSP и Stride показали сравнимое качество предсказания, а отличие каждой границы неверного предсказания от границы в pdb-файле составляет один аминокислотный остаток.
| № элемента вторичной структуры | Результат DSSP (номера остатков) | Результат Stride (номера остатков) | Аннотация в pdb-файле (номера остатков) |
| α-спираль | |||
| 1 | 3 – 14 | 3 – 15 | 2 – 15 |
| 2 | 54 – 65 | 54 – 65 | 53 – 65 |
| 3 | 87 – 96 | 87 – 96 | 86 – 97 |
| 4 | 117 – 120 | 117 – 120 | 117 – 121 |
| β-тяж | |||
| 1 | 26 – 32 | 26 – 32 | 26 – 32 |
| 2 | 37 – 41 | 37 – 41 | 37 – 41 |
| 3 | 70 – 77 | 70 – 77 | 70 – 77 |
| 4 | 102 – 106 | 102 – 106 | 102 – 106 |
| 5 | 112 – 116 | 112 – 116 | 112 – 116 |
| 310 спирали | |||
| 1 | 48 – 50 | 48 – 50 | 47 – 51 |
| 2 | 127 – 129 | 127 – 129 | 126 – 130 |
Помимо отмеченных в таблице спиралей 310, из найденных редких элементов вторичной структуры можно привести β-мостики (β-bridges): согласно выдаче обеих программ, их образуют остатки с номерами 18, 43, 52.
Сервис SheeP
С помощью SheeP была построена карта β-листа в цепи A структуры 1UWZ (были использованы параметры по умолчанию):

По карте видно, что выбранный β-лист состоит из пяти тяжей. Изображение его в структуре приведено ниже.
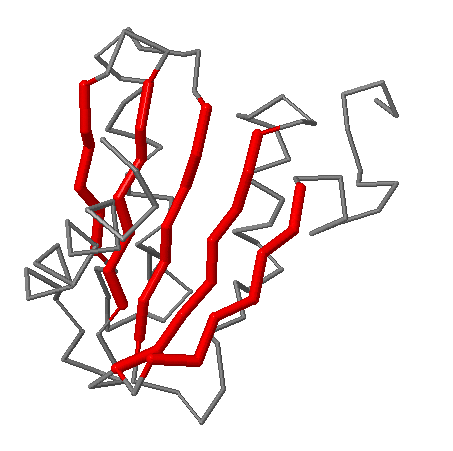
Один столбец карты соответствует одному «хребту» (crest) β-листа. Один из таких «хребтов», соответствующий аминокислотам в третьем столбце карты (остаткам Met115, Val103, Leu73, Leu30, Tyr38), показан на следующем изображении:
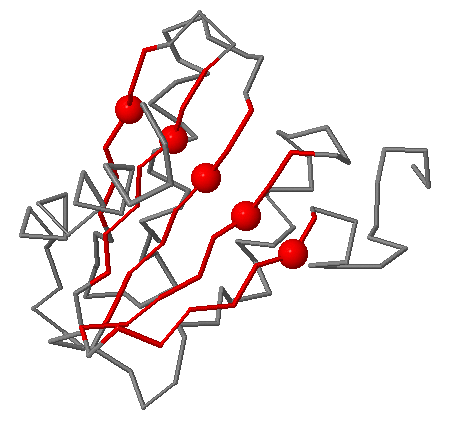
Выбранный β-лист имеет сходный процент гидрофобных аминокислотных остатков с каждой стороны листа. Это также видно на изображении гидрофобных кластеров, определённых с помощью сервиса CluD (см. далее).
Совмещение структур
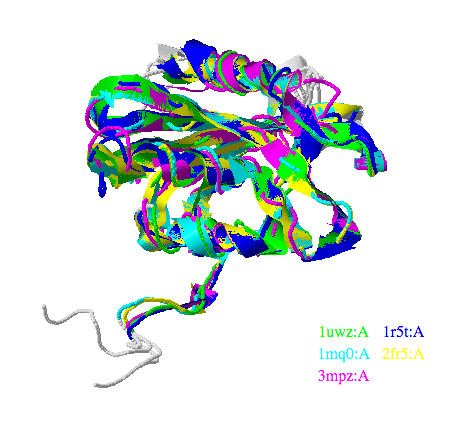
Совмещение структур 1UWZ и структурных гомологов
С помощью сервиса PDBeFold были выбраны 4 структурных гомолога 1UWZ: 1R5T, 1MQ0, 2FR5, 3MPZ. Для пяти структур – 1UWZ и перечисленных выше – были загружены выравнивание последовательностей по совмещению структур (попарное и множественное) и само совмещение структур:
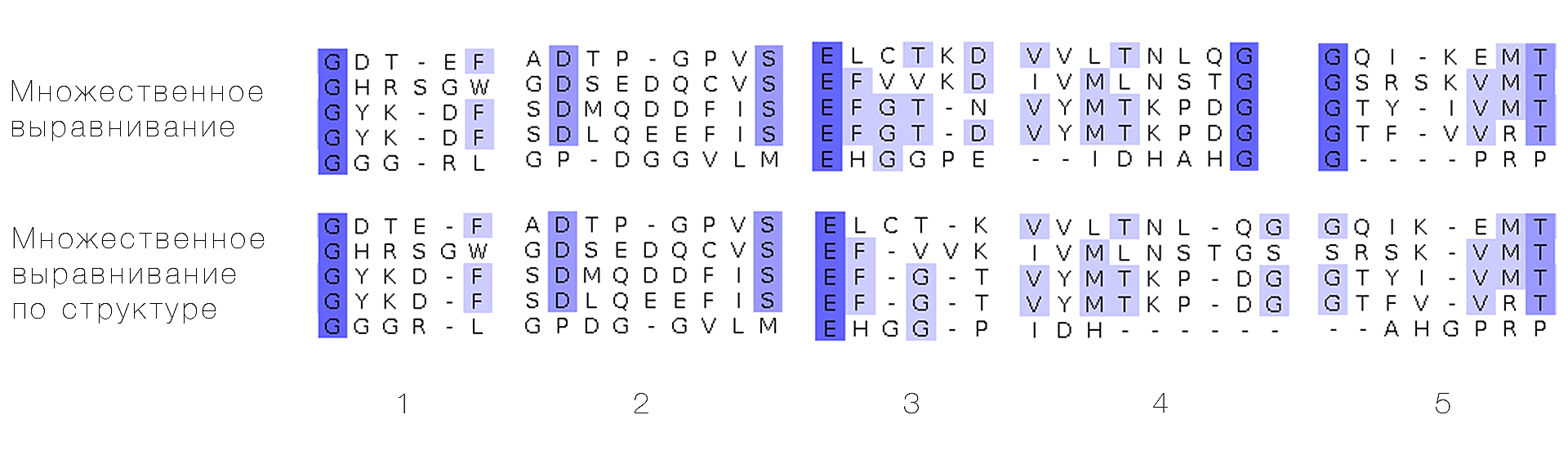
Выравнивание по структуре (большие буквы в файле с выравниванием) можно сравнить с выравниванием последовательностей, выполненным, например, в программе JalView (с помощью Tcoffee со стандартными параметрами):

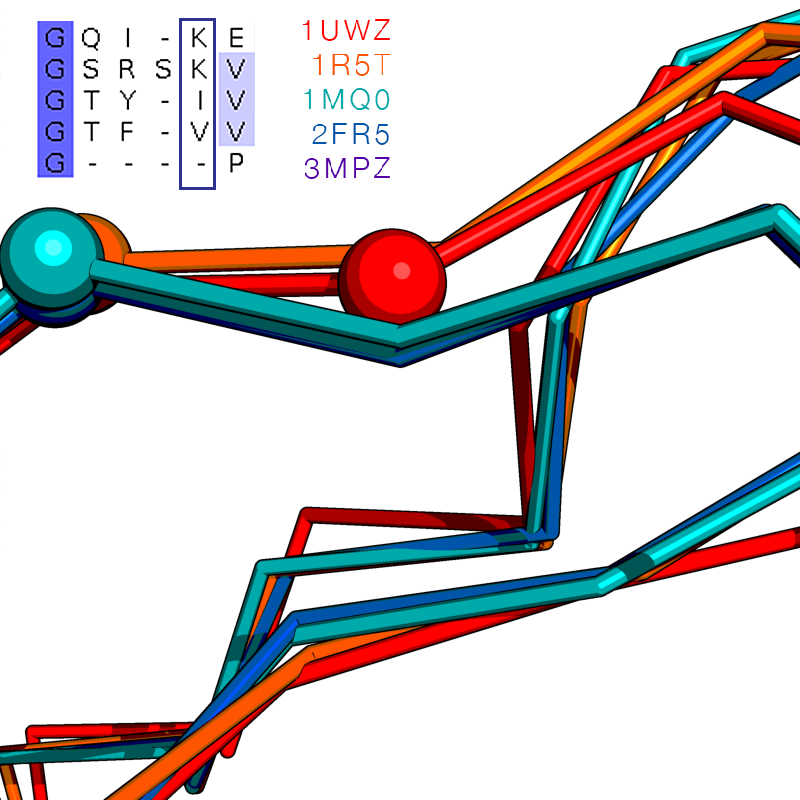
При сравнении множественного выравнивания последовательностей и выравнивания последовательностей по структуре удаётся выявить несколько различий. Так, против 1R5T:Ser77 во множественном выравнивании во всех остальных последовательностях находятся гэпы, однако этот остаток выровнен в выравнивании по структуре, а против гэпов стоит остаток 1R5T:Gly78. Этот и некоторые другие несоответствия отображены ниже. Можно отметить, что все найденные несоответствия наблюдаются именно в неструктурированных участках (или иногда захватывают граничные остатки элементов вторичной структуры) и отсутствуют в спиралях или β-тяжах.
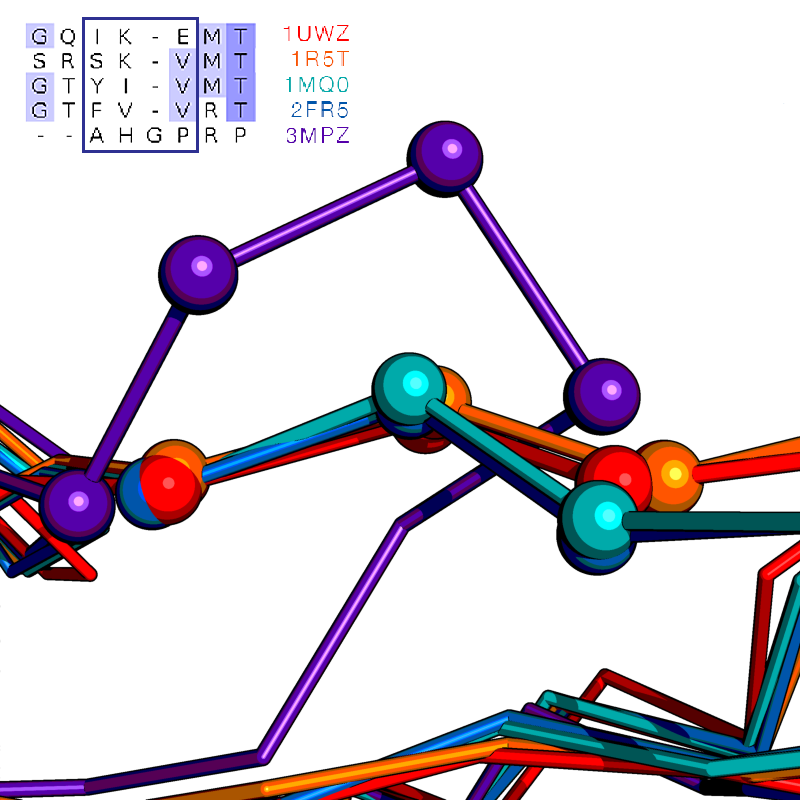
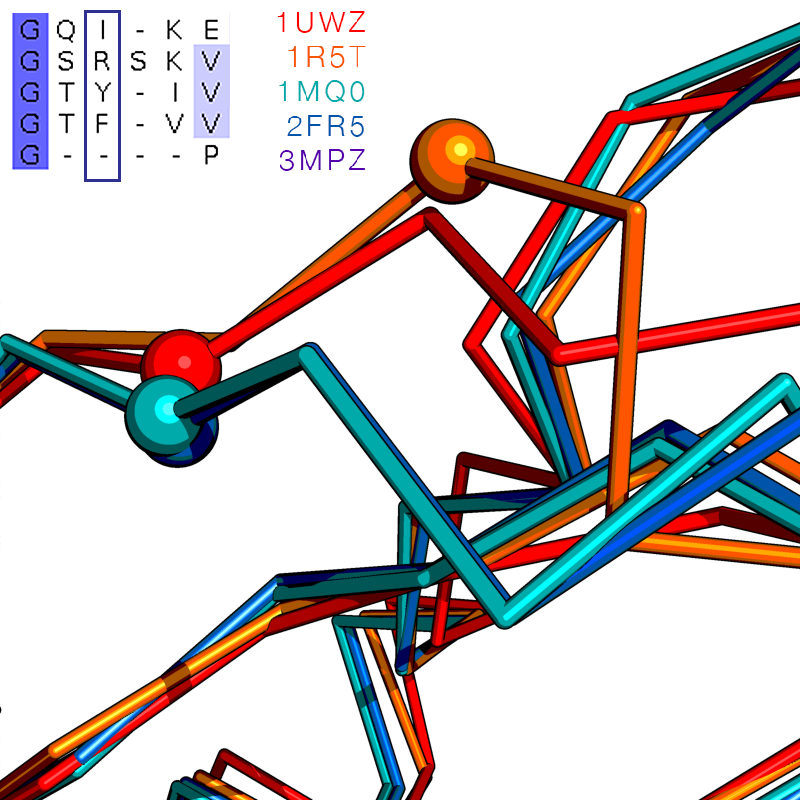
Рассмотрим, например, фрагмент выравнивания под номером 5 на рисунке выше. Ниже приведены изображения совмещения всех структур, фрагмента выравнивания, выполненного по структуре, а также фрагмента множественного выравнивания (выравненные атомы отмечены в шаровой модели). Можно заключить, что выравнивание по структуре для этого фрагмента более правдоподобно, нежели выравнивание по последовательности.

Совмещение по заданному выравниванию
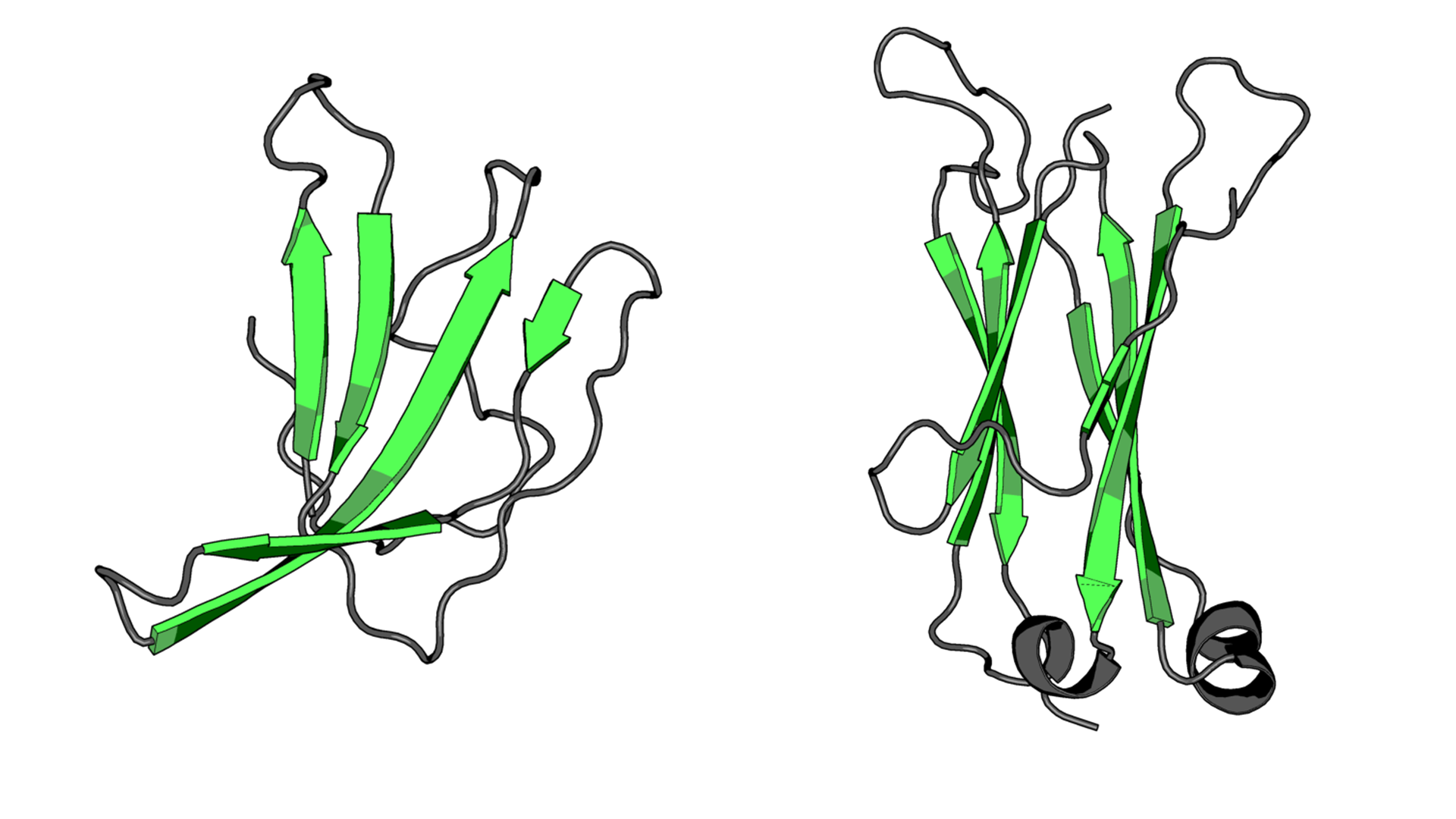
Структуры константного домена человеческого T-клеточного рецептора из цепи α и из цепи β, а именно 1oga, region d: 118-202 и 1oga, region e: 119-245, были сохранены в формате pdb: t_alpha.pdb, t_beta.pdb:
С помощью сервиса SheeP были получены карты β-листов в этих доменах:
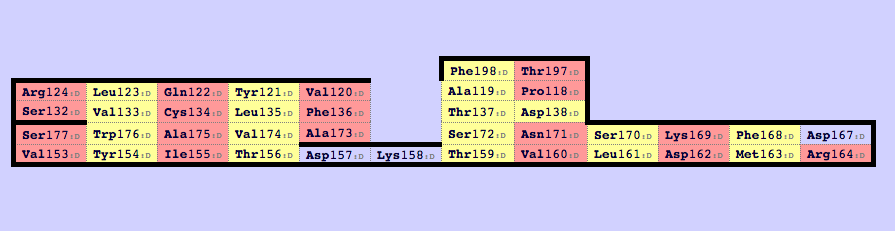
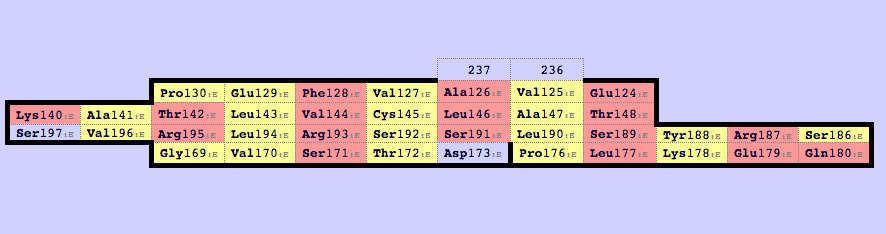
По видимому, лист с картой map 0 в цепи β соответствует листу в цепи α. Карты этих листов приведены на изображениях выше «в одной ориентации». При построении выравнивания последовательностей этих β-тяжей консервативные остатки цистеина в них задают выравнивание центрального тяжа. Остатки, спаренные с консервативным цистеином, задают выравнивание «соседних» тяжей. Таким образом можно построить выравнивание (записано в файле t-alpha_and_t-beta.mfa).
Полученную информацию о выровненных остатках можно использовать для совмещения структур в PyMol:
pair_fit \ t_alpha & resi 134 & n. ca, t_beta & resi 145 & n. ca, \ t_alpha & resi 122 & n. ca, t_beta & resi 127 & n. ca, \ t_alpha & resi 175 & n. ca, t_beta & resi 192 & n. ca, \ t_alpha & resi 155 & n. ca, t_beta & resi 172 & n. ca, \ t_alpha & resi 133 & n. ca, t_beta & resi 144 & n. ca, \ t_alpha & resi 135 & n. ca, t_beta & resi 146 & n. ca
Совмещение сохранено в соответствующем файле:
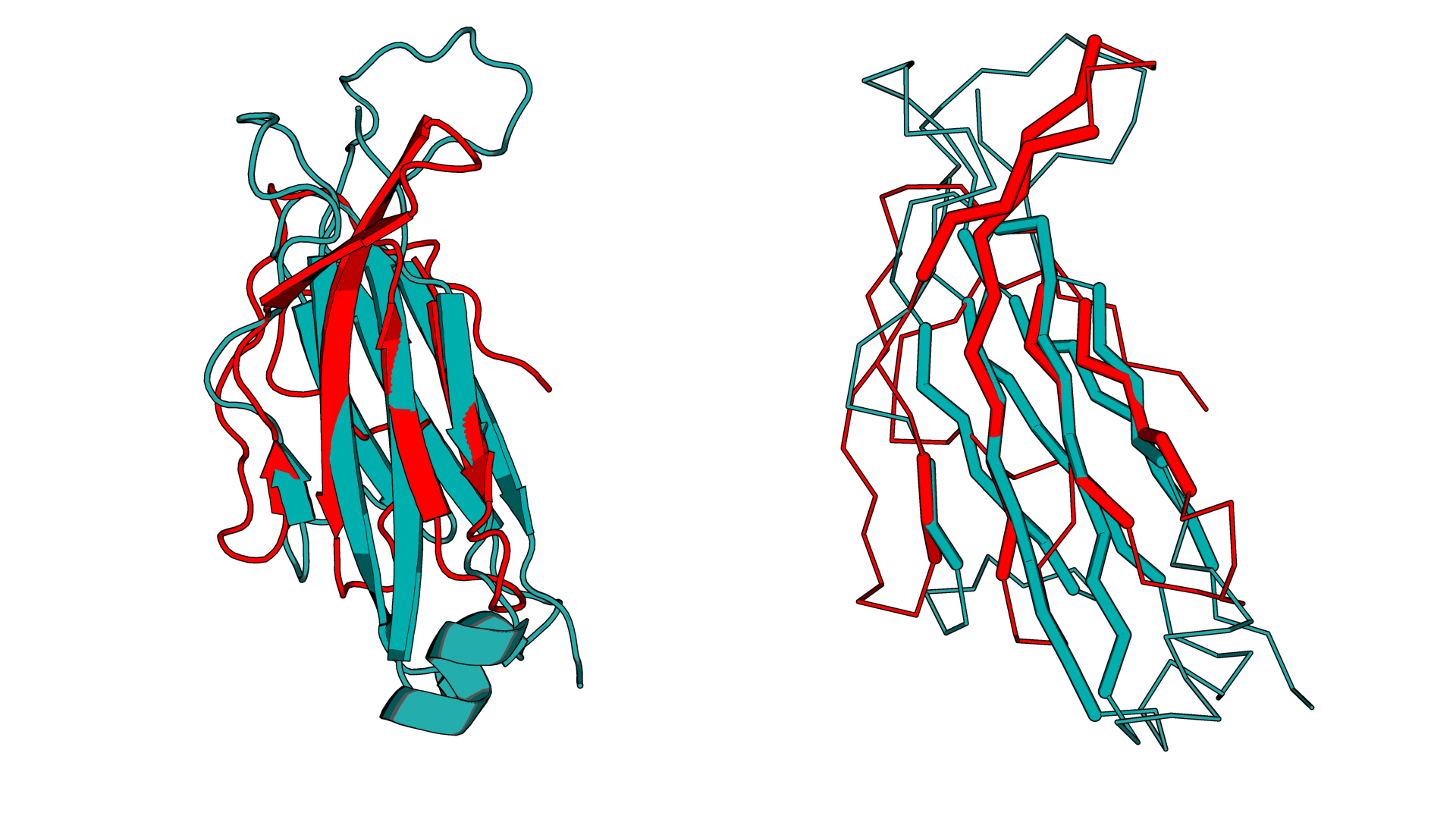
На изображении совмещения видно, что общий ход полипептидной цепи в пространстве совпадает. Т. е. топологии сходны. Можно также обратить внимание на то, что каждой петле в одной структуре соответствует петля в другой.
Нахождение гидрофобных кластеров
С помощью сервиса Clud в структуре 1UWZ был произведён поиск гидрофобных кластеров. Со значениями 5.0 и 5 для порога расстояния и размера кластера, соответственно, было найдено 5 кластеров, которые обозначены на изображении ниже:
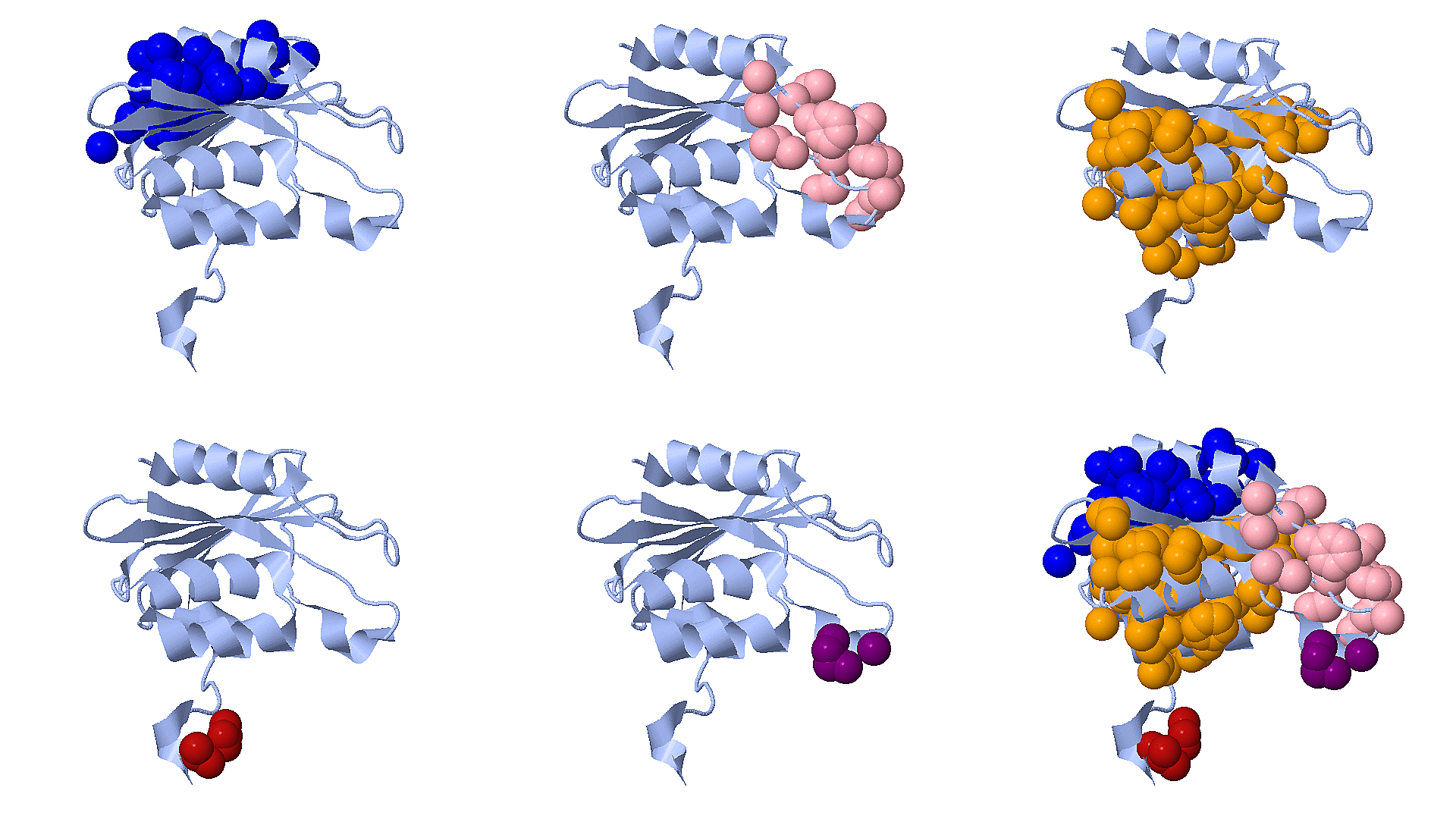
Пожалуй, можно сказать, что найденные кластеры отвечают группам (с одним и более) элементов вторичной структуры белка, определяемым визуально.
Поиск гидрофобных кластеров в димере белка 3Q8T (цепях A и B структуры с мотивом coiled coil) позволяет различить гидрофобные кластеры в участках контакта двух спиралей (значения для порога расстояния и размера кластера – 4.5 и 3, соответственно):
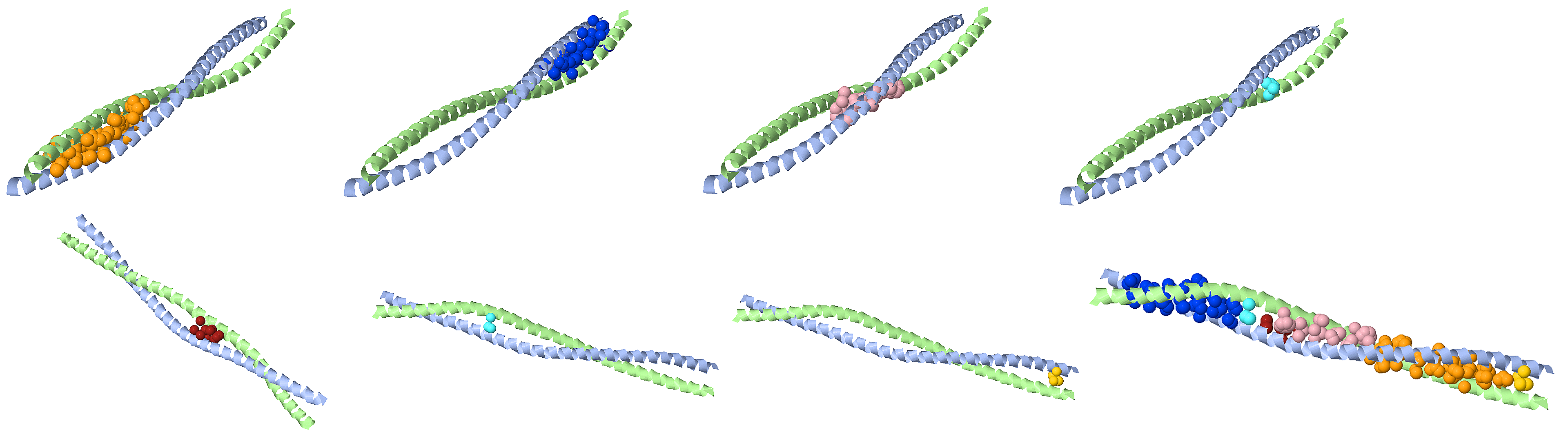
Построение поверхности
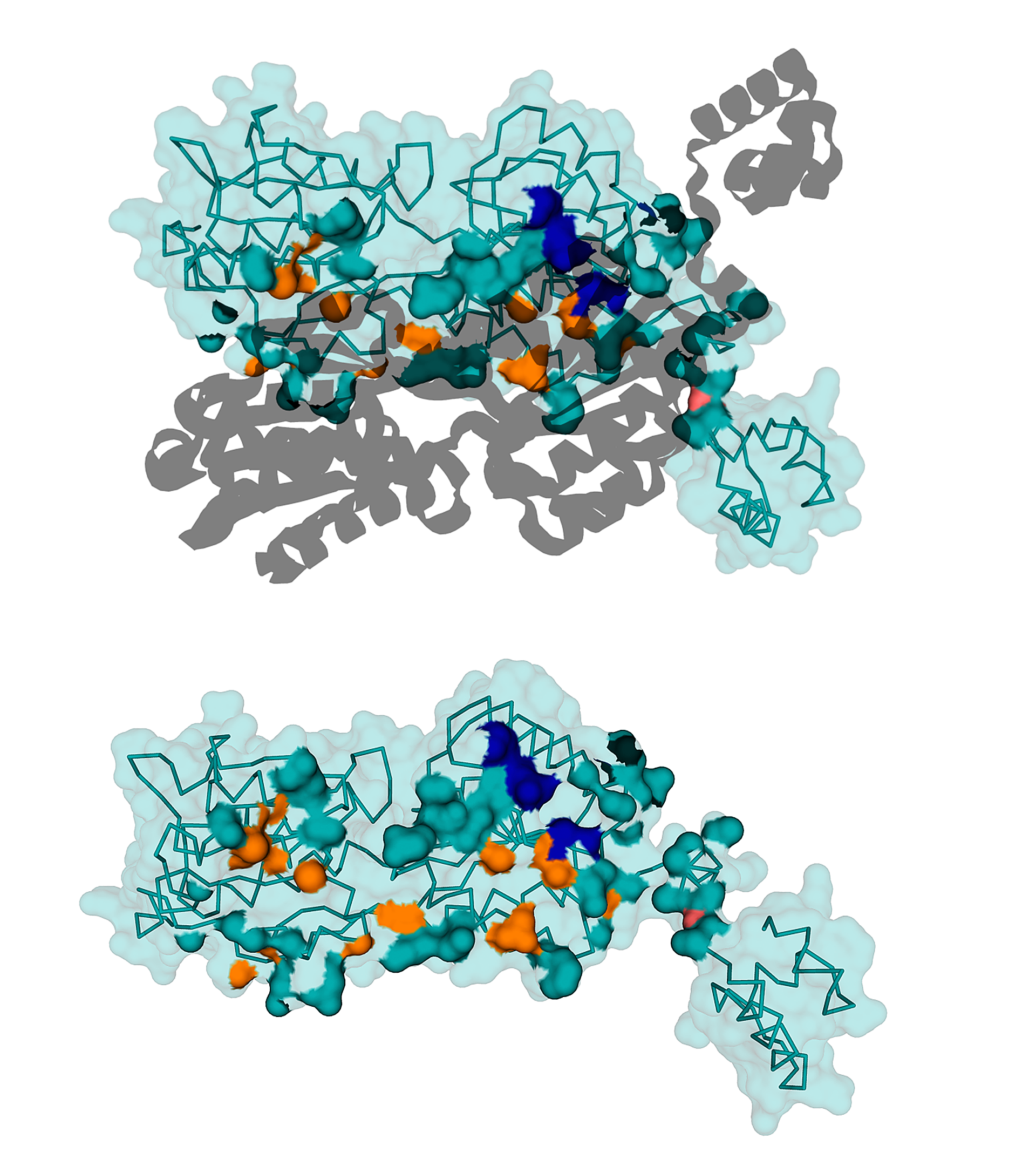
Для комплекса димера пуринового репрессора с ДНК (биологическая единица 1JFT) с использованием средств программы PyMol были созданы изображения, демонстрирующие контакт мономеров белка и между собой и контакт белка с ДНК. Контакты поверхности мономера белка с симметричным мономером и с двойной спиралью ДНК выделены цветом на изображениях ниже.
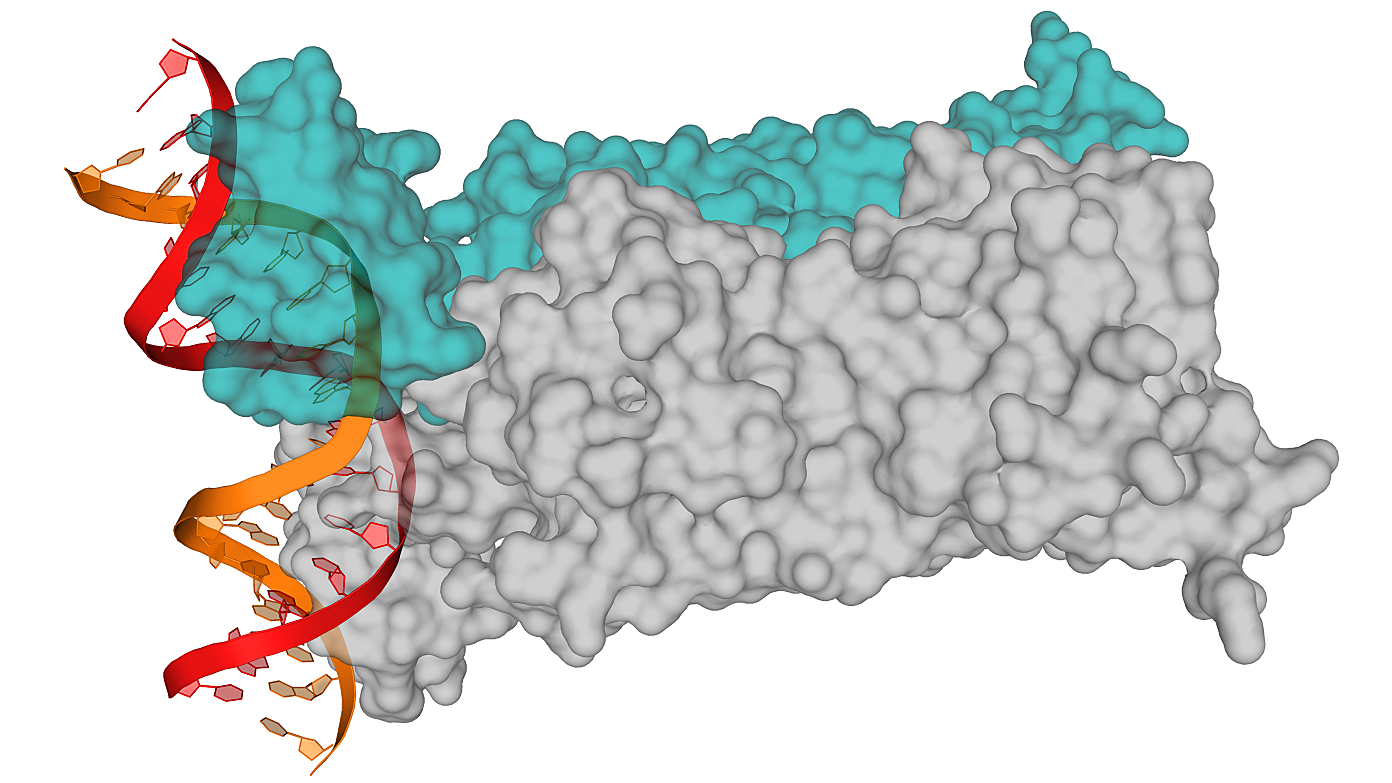
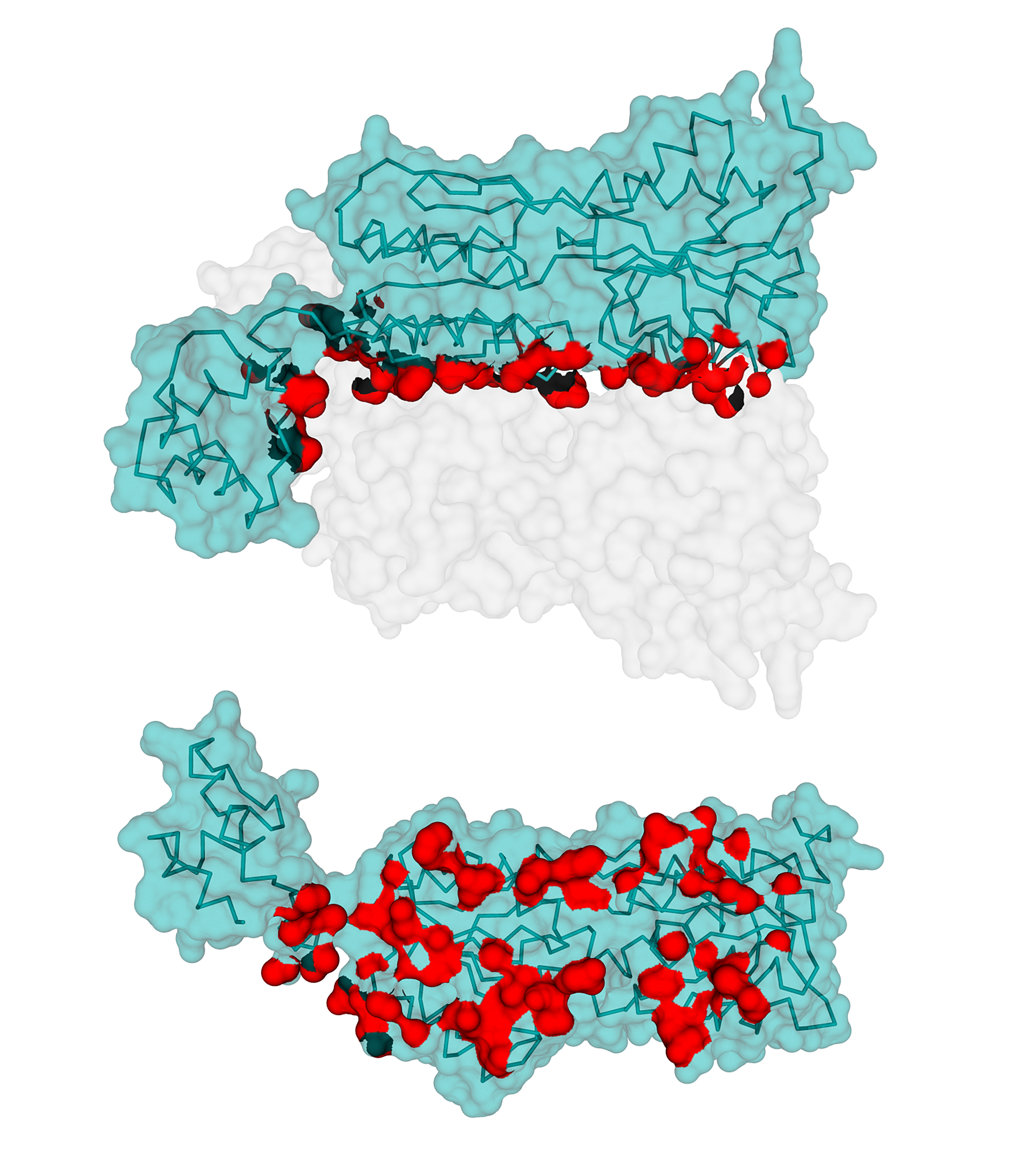
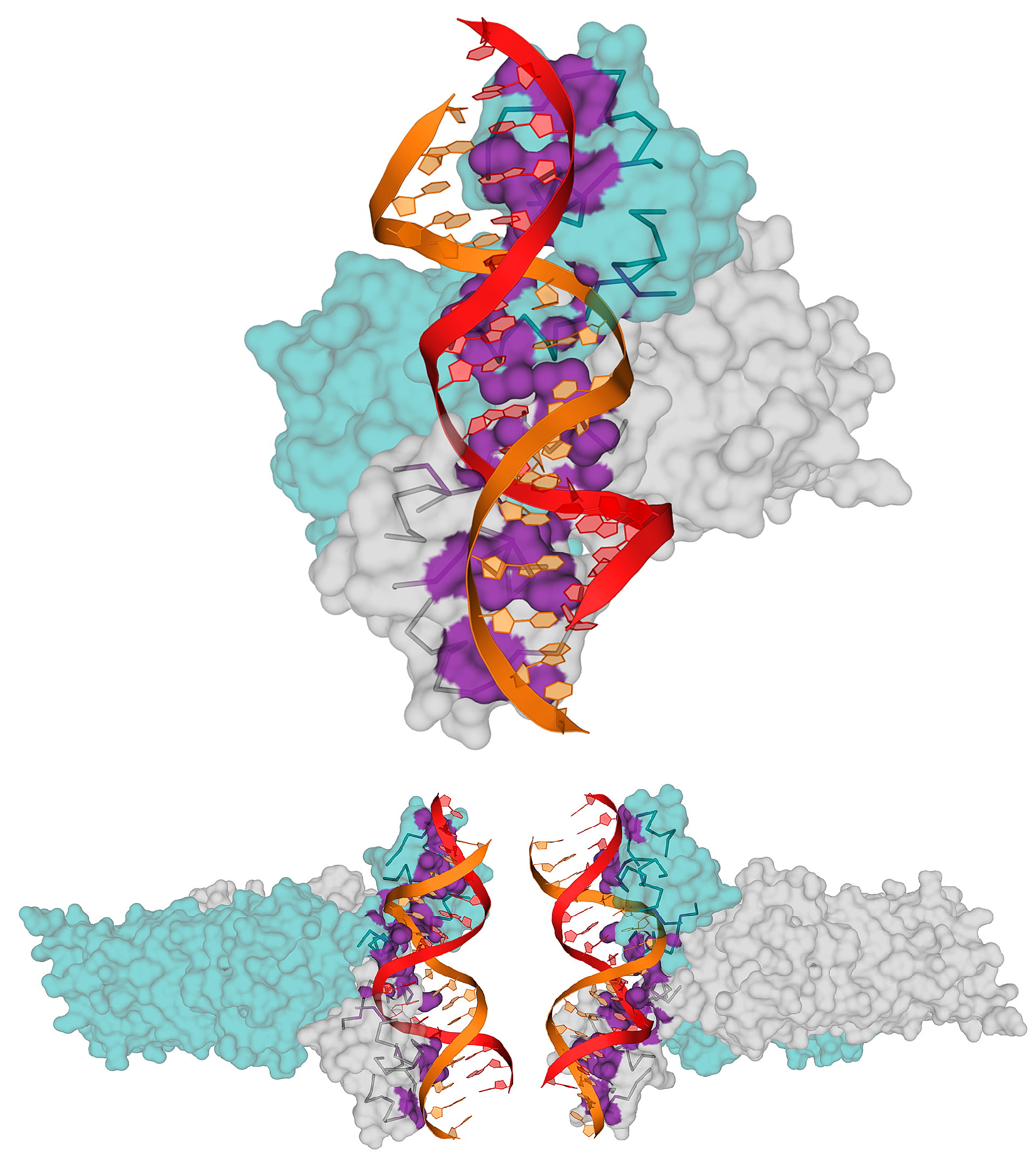
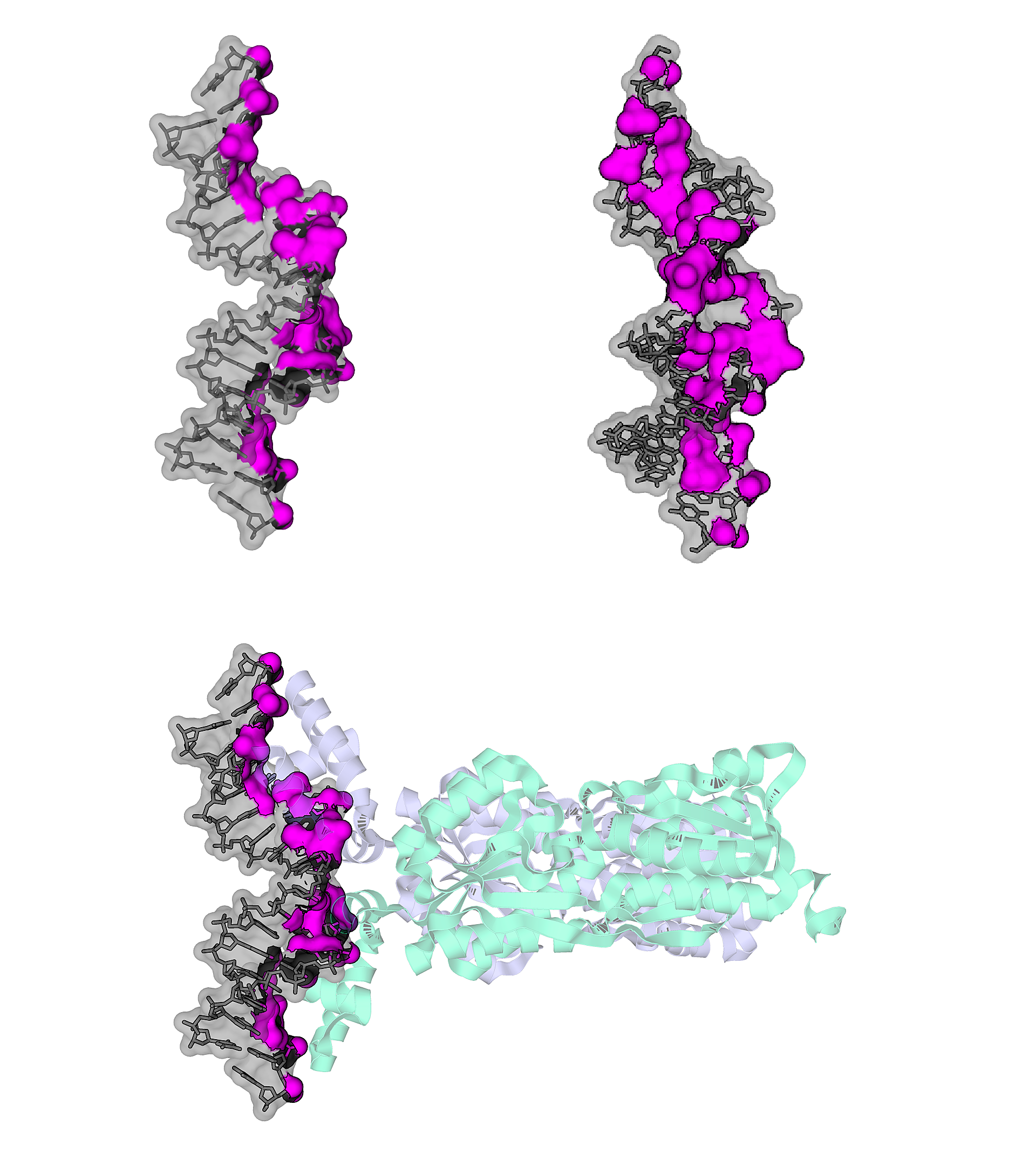
С помощью сервиса Clud в структуре 1JFT были определены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка (distance threshold – 5 Å). На изображении ниже показана поверхность контакта мономера белка с симметричным мономером; поверхность, относящаяся ко входящим в найденные гидрофобные кластеры атомам, выделена синим, оранжевым и пастельно-розовым цветами (разные цвета соответствуют разным кластерами; приведены вид «сбоку» и вид «снизу»):
Сравнение доменов SCOP и Pfam
Для белка цитидиндезаминазы Bacillus subtilis (PDB ID 1UWZ, цепь A гомодимера) были выделены домены Pfam и SCOP. Ниже эти домены обозначены в последовательности белка:
# Pfam domain: |-----------------------------------------------------------
# SCOP domain: |===========================================================
MNRQELITEALKARDMAYAPYSKFQVGAALLTKDGKVYRGCNIENAAYSMCNCAERTALF
# In PDB file: ++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
# --------------------------------------------|
# =====================================================================|
KAVSEGDTEFQMLAVAADTPGPVSPCGACRQVISELCTKDVIVVLTNLQGQIKEMTVEELLPGAFSSEDLHDERKL
# ++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
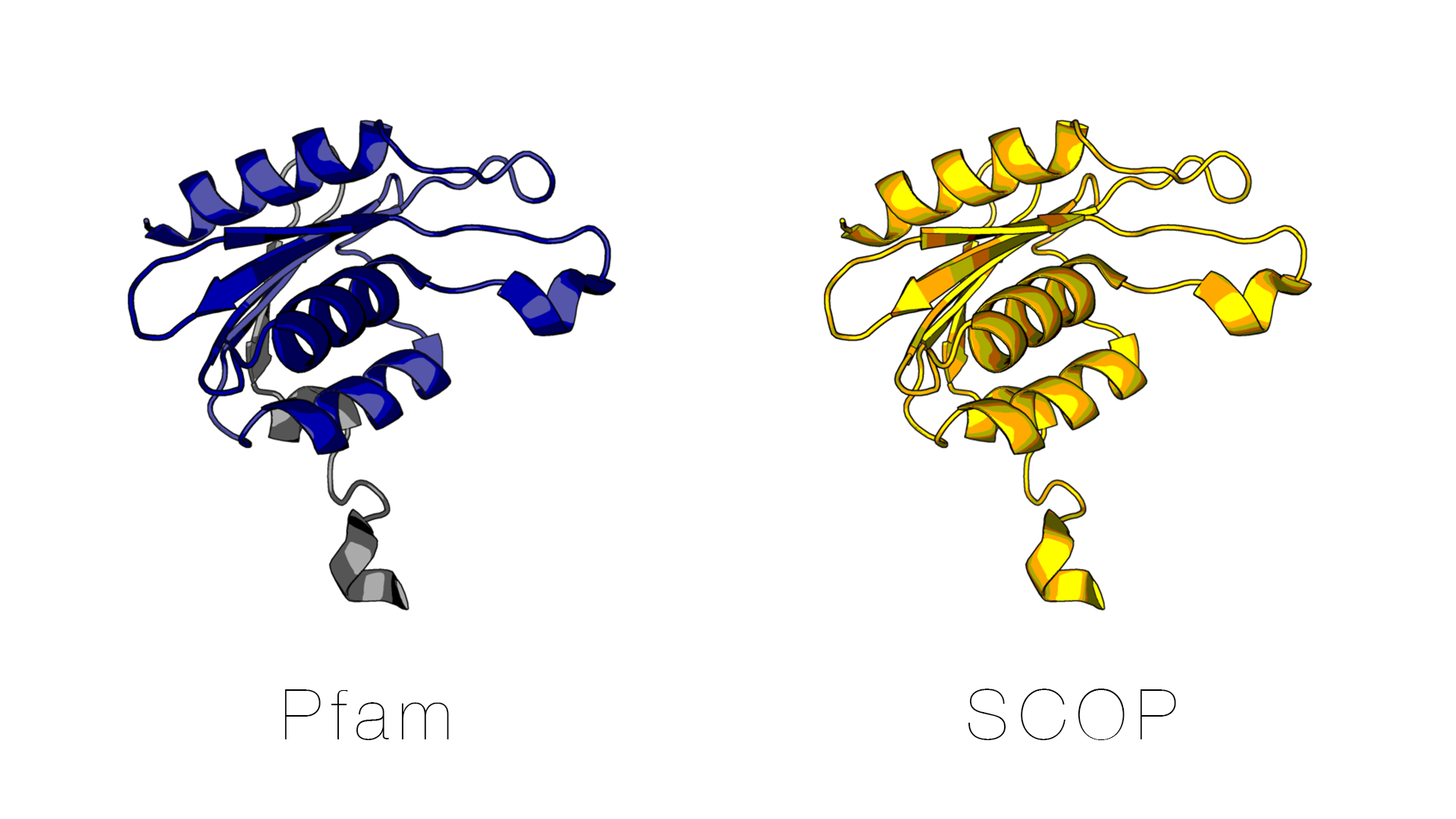
Видно, что домен в SCOP (d1uwza_) совпадает с последовательностью белка из файла структуры монодоменной цитидиндезаминазы 1uwz.pdb. Границы домена согласно Pfam (dCMP_cyt_deam_1) несколько короче.
Границы доменов также можно обозначить на структуре:
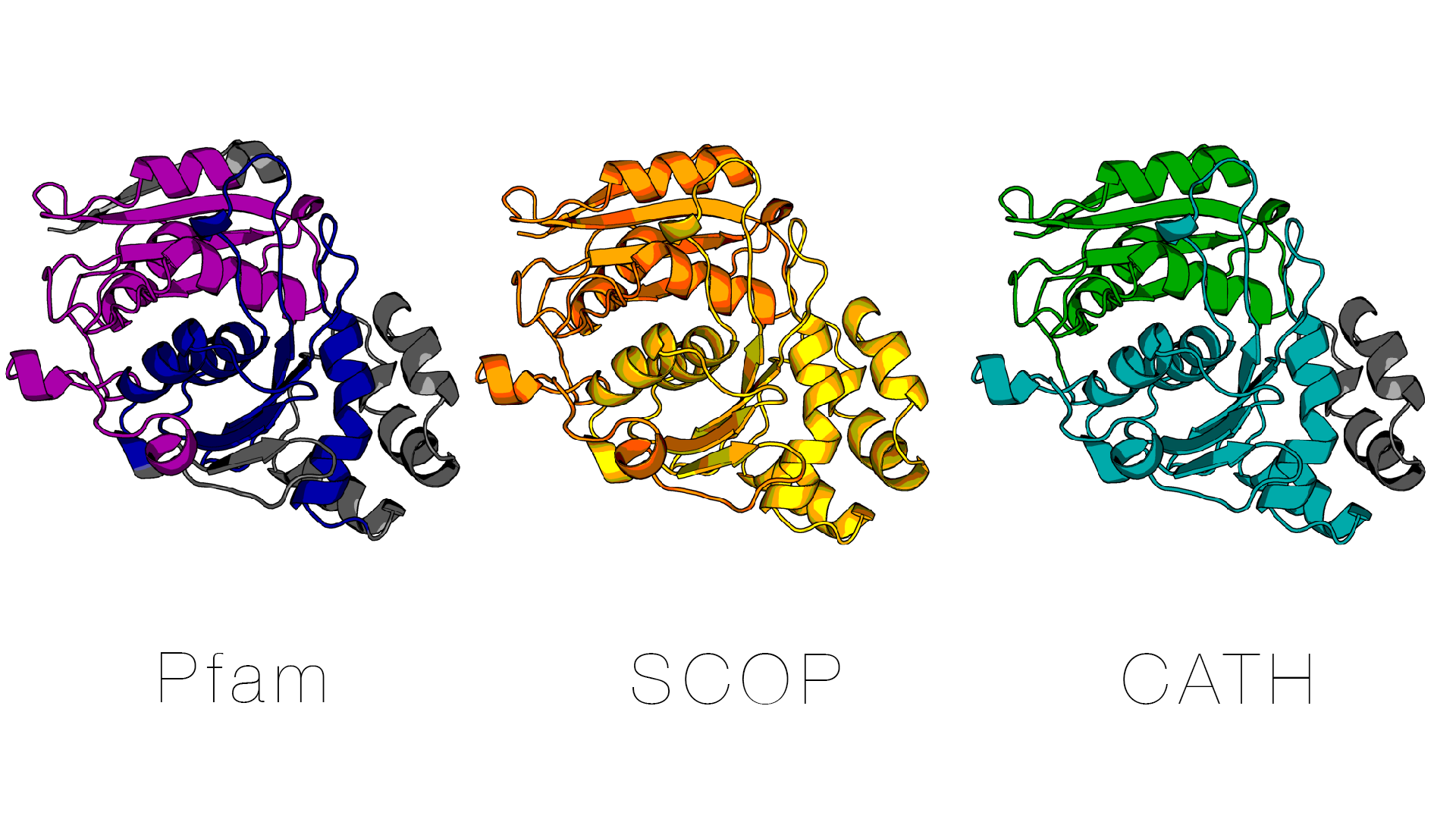
Более интересным представляется рассмотрение двухдоменной цитидиндезаминазы, например из Escherichia coli (PDB ID 1AF2). Согласно SCOP, в белке два домена: d1af2a1 и d1af2a2; Pfam выделяет домены dCMP_cyt_deam_1 и dCMP_cyt_deam_2. Домены SCOP и Pfam, а также CATH (1af2A01, 1af2A02) отмечены ниже в последовательности белка:
# Pfam domain: 1---
# SCOP domain: 1=================================================
# CATH domain: 1*********************
MHPRFQTAFAQLADNLQSALEPILADKYFPALLTGEQVSSLKSATGLDED
# In PDB file: ++++++++++++++++++++++++++++++++++++++++++++++++++
# --------------------------------------------------------------- # Pfam
# =============================================================== # SCOP
# *************************************************************** # CATH
ALAFALLPLAAACARTPLSNFNVGAIARGVSGTWYFGANMEFIGATMQQTVHAEQSAISHAWL
# +++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
# -----------------------------1 2------------------- # Pfam
# ====================================1 # SCOP
# 2========================= # SCOP
# **********************************1 # CATH
SGEKALAAITVNYTPCGHCRQFMNELNSGLDLRIHLPGREAHALRDYLPDAFGPKDLEIKTLL
# +++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
# --------------------------------------------------------------- # Pfam
# =============================================================== # SCOP
# 2*********************************************************** # CATH
MDEQDHGYALTGDALSQAAIAAANRSHMPYSKSPSGVALECKDGRIFSGSYAENAAFNPTLPP
# +++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++
# ---------------------------------------2 # Pfam
# ======================================================2 # SCOP
# ******************************************************2 # CATH
LQGALILLNLKGYDYPDIQRAVLAEKADAPLIQWDATSATLKALGCHSIDRVLLA
# +++++++++++++++++++++++++++++++++++++++++++++++++++++++
В этом случае домены Pfam примерно соответствуют доменам SCOP, но также несколько короче их по длине. Границы доменов в CATH отличаются от таковых в SCOP. Домены Pfam, SCOP и CATH в структуре показаны разным цветом на изображении ниже.