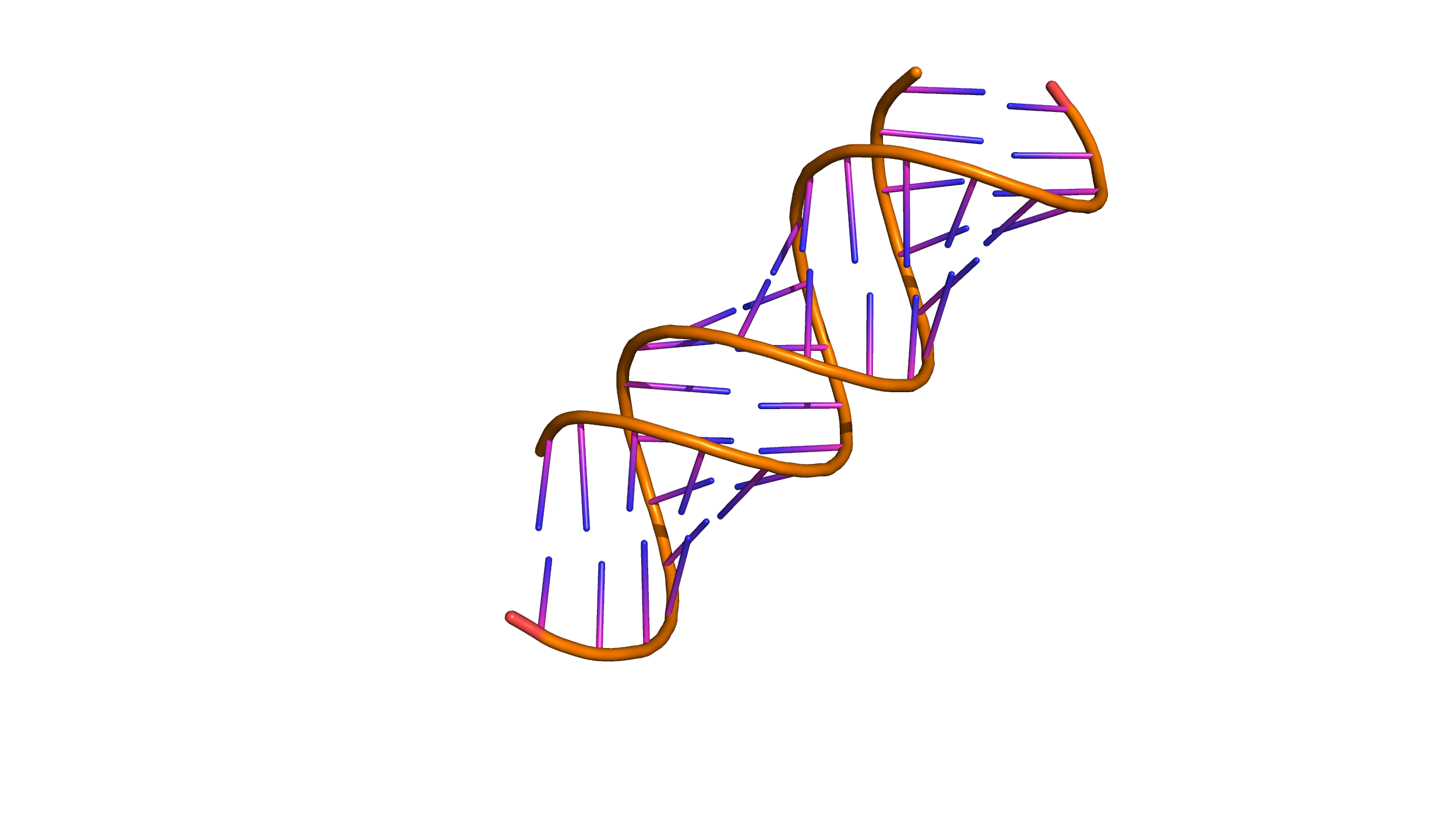
Задание 1
A-форма ДНК
gatc-a.pdb
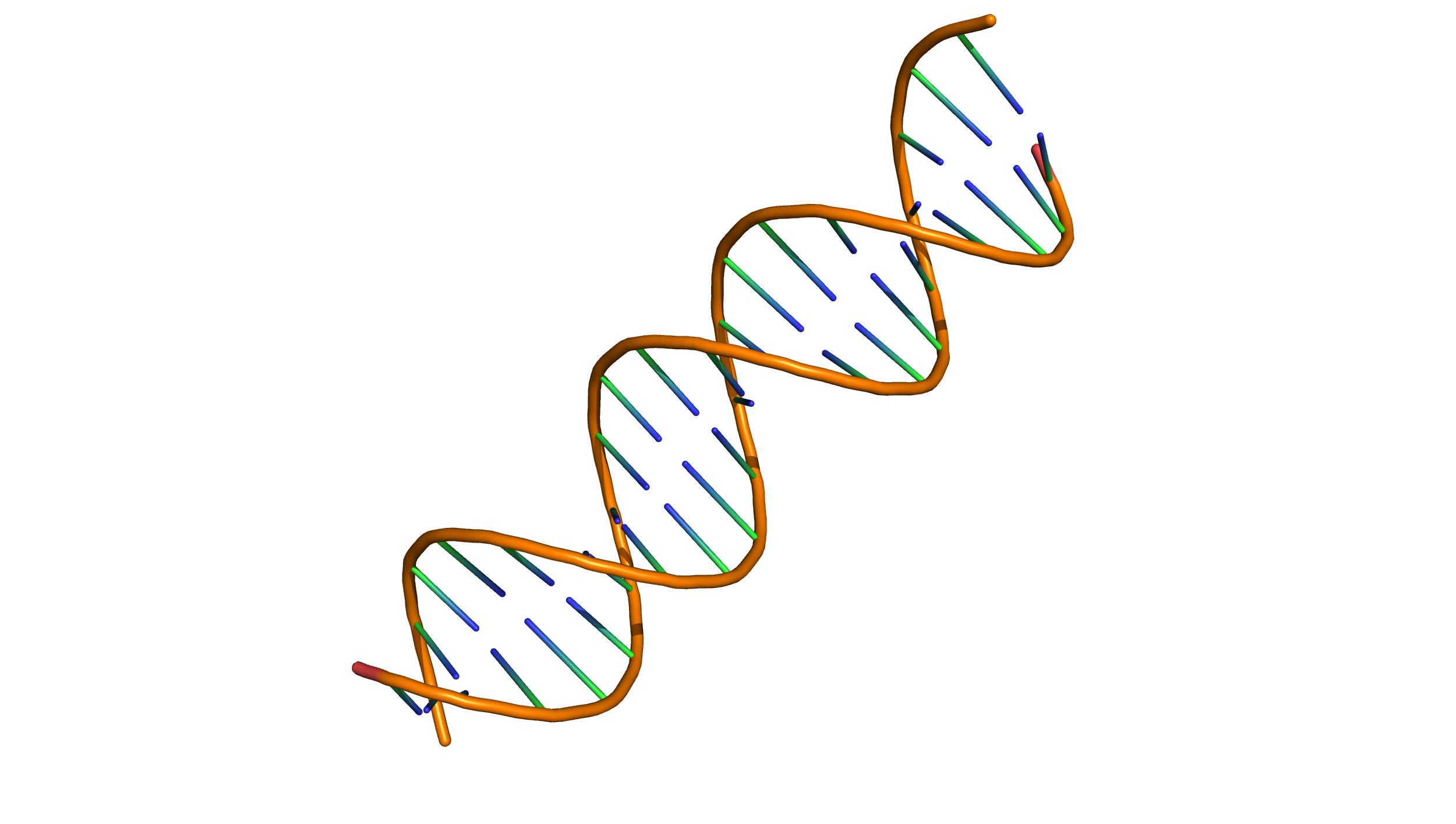
B-форма ДНК
gatc-b.pdb
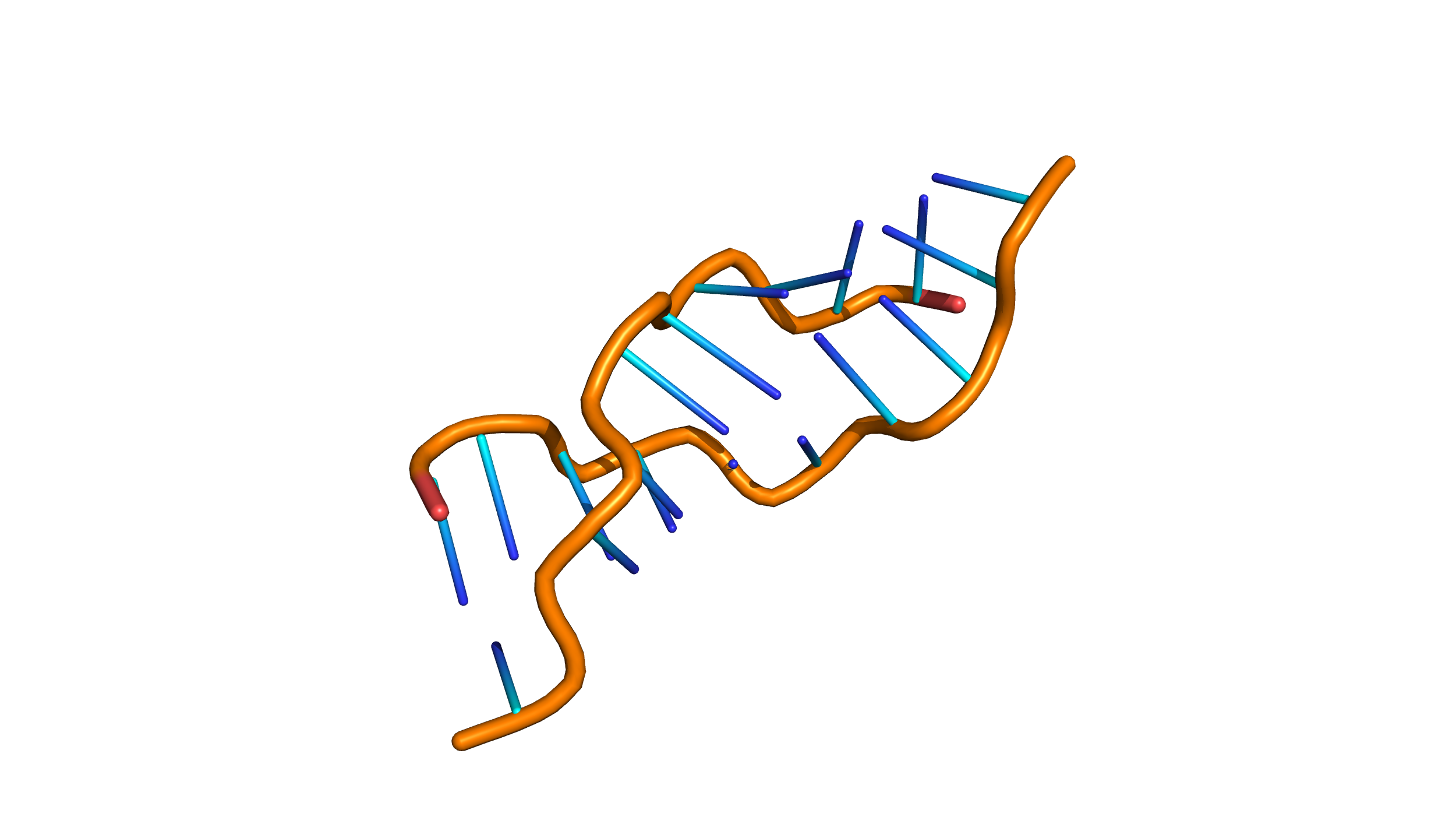
Z-форма ДНК
gatc-z.pdb
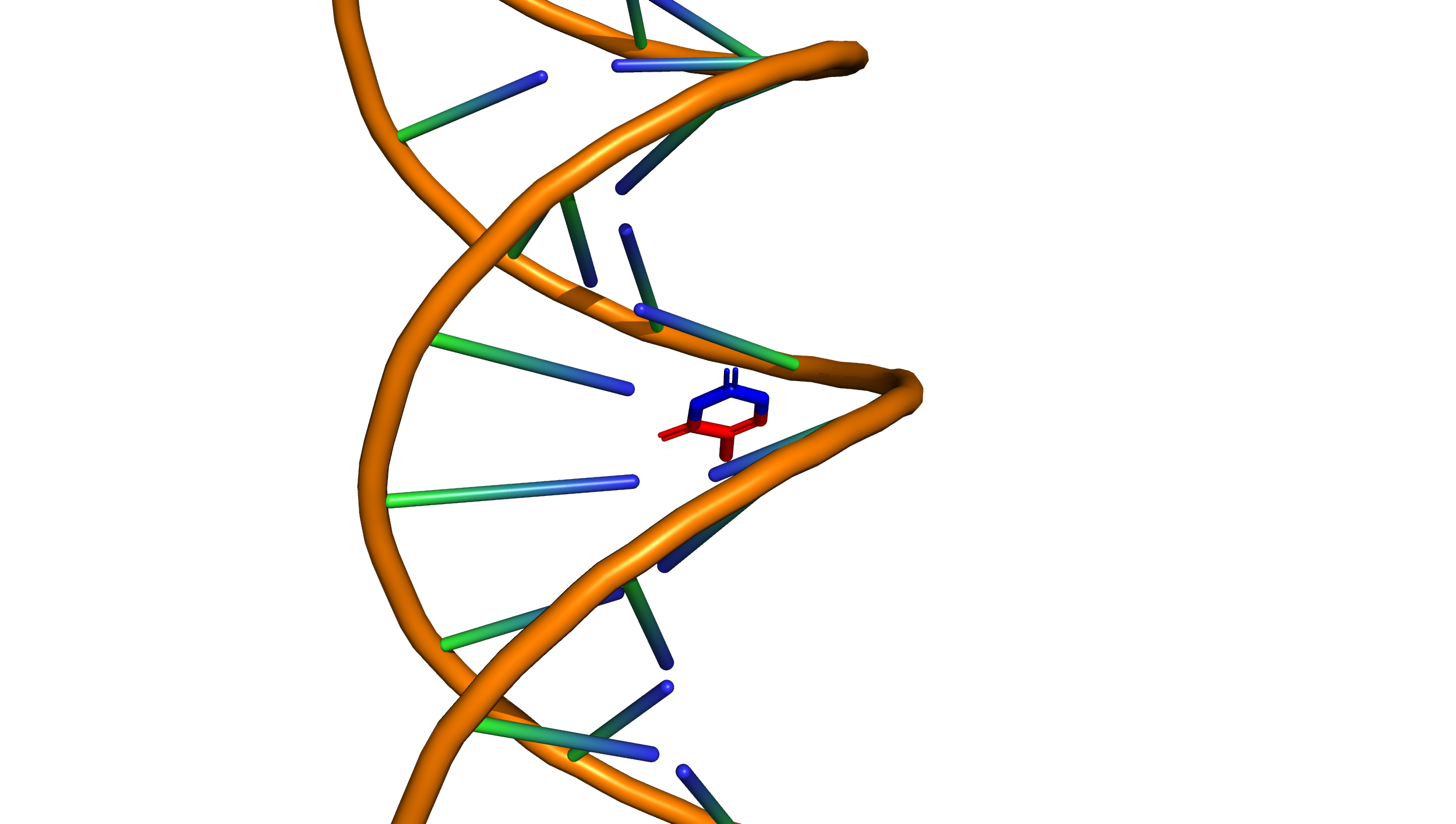
Задание 2
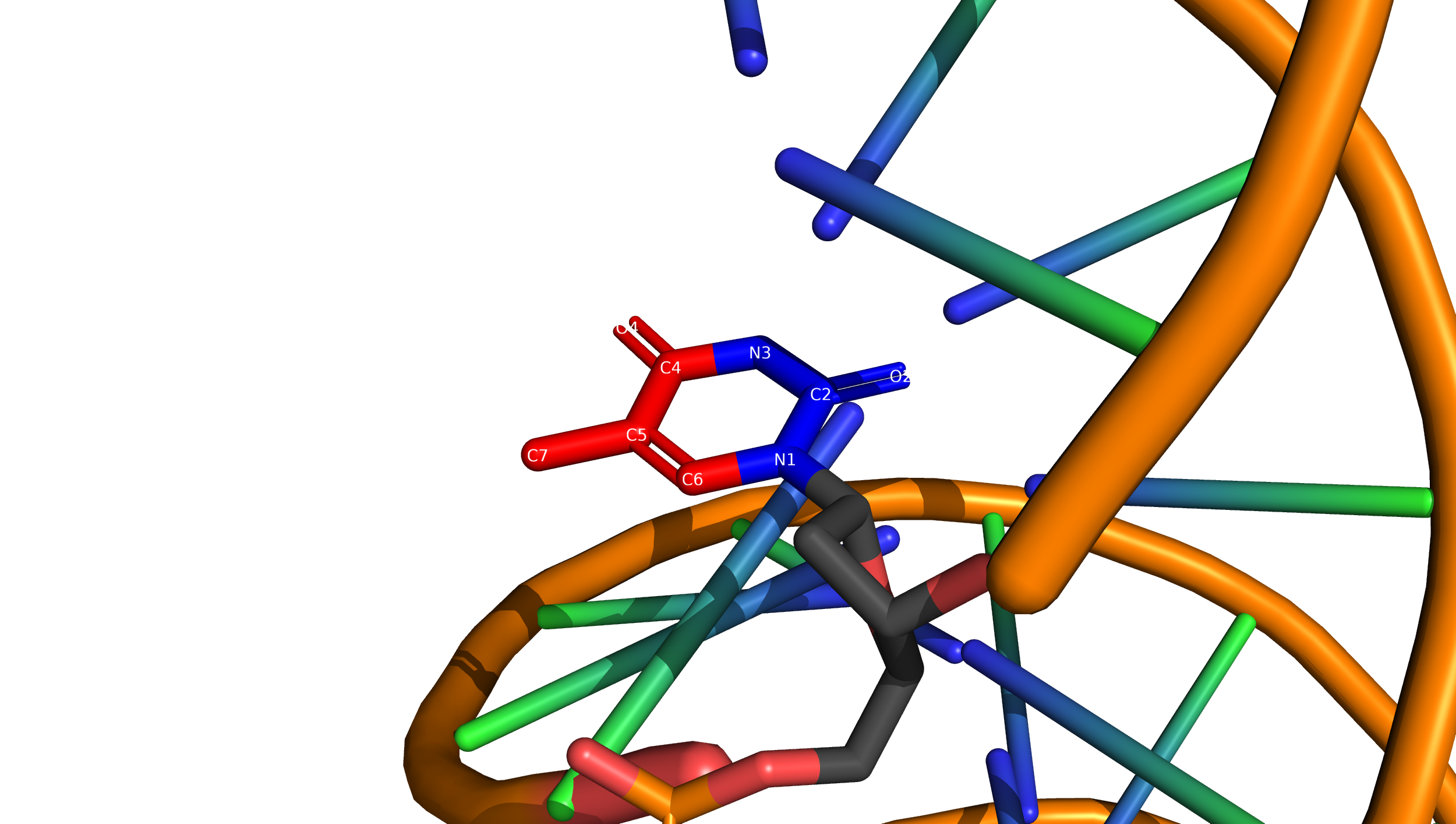
На рисунках показано одно из азотистых оснований (тимин) в структуре B-ДНК. Атомы синего цвета (N1, C2, O2, N3) смотрят в сторону малой бороздки, а атомы красного цвета (C4, O4, C5, C6, C7) в сторону большой
| A-форма | B-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.0 | 33.8 | 45.6 (в случае C-G динуклеотидов) |
| Число оснований на виток | 12 | 10 | 12 (то же самое) |
| Ширина большой бороздки (Å) | 18.5 (от цитозина) | 20.6 (от гуанина) | 18.3? (от цитозина) |
| Ширина малой бороздки (Å) | 9.6 (от гуанина) | 13.2 (от цитозина) | 9.9 (от цитозина) |
Задание 3
На примере 1EHZ (дрожжевая фенилаланиновая тРНК) разбираемся как работают программы find_pair и analyze из пакета 3DNA. Предварительно протонируем структуру. После применения конвейера find_pair -t mytrna.pdb stdout | analyze нас интересует файл mytrna.out, где мы хотим найти информацию о взаимодействии н.п.
Strand I Strand II Helix
1 (0.008) ....>-:...1_:[.RG]G-----C[.RC]:..72_:-<.... (0.006) |
2 (0.006) ....>-:...2_:[.RC]C-----G[.RG]:..71_:-<.... (0.013) |
3 (0.021) ....>-:...3_:[.RG]G-----C[.RC]:..70_:-<.... (0.018) |
4 (0.018) ....>-:...4_:[.RG]G-*---U[.RU]:..69_:-<.... (0.015) |
5 (0.018) ....>-:...5_:[.RA]A-----U[.RU]:..68_:-<.... (0.011) |
6 (0.011) ....>-:...6_:[.RU]U-----A[.RA]:..67_:-<.... (0.020) |
7 (0.008) ....>-:...7_:[.RU]U-----A[.RA]:..66_:-<.... (0.016) x
8 (0.013) ....>-:..41_:[.RU]U-----A[.RA]:..29_:-<.... (0.019) |
9 (0.010) ....>-:..42_:[.RG]G-----C[.RC]:..28_:-<.... (0.007) |
10 (0.025) ....>-:..43_:[.RG]G-----C[.RC]:..27_:-<.... (0.011) |
11 (0.026) ....>-:..45_:[.RG]G-**--C[.RC]:..25_:-<.... (0.011) |
12 (0.005) ....>-:..11_:[.RC]C-----G[.RG]:..24_:-<.... (0.013) |
13 (0.012) ....>-:..12_:[.RU]U-----A[.RA]:..23_:-<.... (0.030) |
14 (0.013) ....>-:..13_:[.RC]C-----G[.RG]:..22_:-<.... (0.017) |
15 (0.025) ....>-:..14_:[.RA]A-**--U[.RU]:...8_:-<.... (0.016) |
16 (0.021) ....>-:..15_:[.RG]G-**+-C[.RC]:..48_:-<.... (0.015) x
17 (0.026) ....>-:..19_:[.RG]G-----C[.RC]:..56_:-<.... (0.014) +
18 (0.014) ....>-:..33_:[.RU]U-**--A[.RA]:..36_:-<.... (0.015) +
19 (0.024) ....>-:..50_:[.RU]U-----A[.RA]:..64_:-<.... (0.014) |
20 (0.030) ....>-:..51_:[.RG]G-----C[.RC]:..63_:-<.... (0.016) |
21 (0.020) ....>-:..52_:[.RU]U-----A[.RA]:..62_:-<.... (0.016) |
22 (0.027) ....>-:..53_:[.RG]G-----C[.RC]:..61_:-<.... (0.012) |
Note: This structure contains 5[4] non-Watson-Crick base-pairs.
****************************************************************************
Detailed H-bond information: atom-name pair and length [ O N]
1 G-----C [3] O6 - N4 2.83 N1 - N3 2.88 N2 - O2 2.84
2 C-----G [3] O2 - N2 3.01 N3 - N1 2.97 N4 - O6 2.86
3 G-----C [3] O6 - N4 2.94 N1 - N3 2.87 N2 - O2 2.76
4 G-*---U [2] O6 - N3 2.91 N1 - O2 2.82
5 A-----U [2] N6 - O4 3.15 N1 - N3 2.88
6 U-----A [2] N3 - N1 2.78 O4 - N6 3.04
7 U-----A [2] N3 - N1 2.73 O4 - N6 2.80
8 U-----A [2] N3 - N1 2.87 O4 - N6 3.06
9 G-----C [3] O6 - N4 3.15 N1 - N3 2.98 N2 - O2 2.77
10 G-----C [3] O6 - N4 2.92 N1 - N3 2.98 N2 - O2 3.02
11 G-**--C [1] N2 * N4 3.94
12 C-----G [3] O2 - N2 2.70 N3 - N1 2.82 N4 - O6 3.00
13 U-----A [2] N3 - N1 2.95 O4 - N6 2.81
14 C-----G [3] O2 - N2 2.73 N3 - N1 2.90 N4 - O6 2.88
15 A-**--U [2] N7 - N3 2.74 N6 - O2 3.00
16 G-**+-C [2] N1 - O2 2.78 N2 - N3 2.83
17 G-----C [3] O6 - N4 3.03 N1 - N3 2.93 N2 - O2 2.82
18 U-**--A [1] N3 - OP2 2.80
19 U-----A [2] N3 - N1 2.79 O4 - N6 2.81
20 G-----C [3] O6 - N4 2.89 N1 - N3 2.80 N2 - O2 2.74
21 U-----A [2] N3 - N1 2.82 O4 - N6 2.96
22 G-----C [3] O6 - N4 2.95 N1 - N3 2.87 N2 - O2 2.80
****************************************************************************
1) Координаты стеблей (номера нуклеотидов): I) 1-6 : 72-76; II) 41-45,11-14 : 29-22,8; III) 50-53 : 64-61
2) Неканонические пары оснований: 4 : 69 (G:U; 2 водородные связи); 45 :25 (G:C; 1 связь); 14 : 8 (A:U; 2 связи(но неканоничные)); 15 : 48 (GC; 2 связи); 33 : 36 (U:A; 1 связь)
3) Стабилизация третичной структуры (изолированные пары): 19 : 56 (G:C); 33 : 36 (U:A)