Пр7
Задание 1
Выбранный организм - серая крыса (norway rat), Rattus norvegicus на латинском
Ссылка на запись на сайте NCBI
Задание 2
| Идентификатор GenBank | GCA_036323735.1 |
| Идентификатор RefSeq | GCF_036323735.1 |
| Уровень сборки генома | Chromosome |
| Общий размер генома (п.н.) | 2.8 млрд |
| Число фрагментов генома в сборке | 76 |
| Параметры N50 и L50 для всех элементов (контигов, скэффолдов) | Scaffold N50 - 137 Mb; Scaffold L50 - 8; Contig N50 - 64.3 Mb; Contig L50 - 15 |
Задание 4
(hemoglobin) AND (genomic dna[Filter] OR mrna[Filter]) - ищем все геномные днк или мрнк, где упоминается гемоглобин - 113634 записей
(hemoglobin) AND (genomic dna[Filter] OR mrna[Filter]) AND (genbank[Filter]) - в генбанке - 75359 записей
(hemoglobin) AND (genomic dna[Filter] OR mrna[Filter]) AND (refseq[Filter]) - в рефсеке - 15360 записей
для человека (в конец AND human[Organism]):
genbank and gene - 1479 записей
genbank and mrna - 9390 записей
refseq and gene - 43 записей
refseq and mrna - 151 записей
Пр8
Задание 1
δ-субъединица АТФ-синтазы (fasta)
id мрнк, кодиирующей этот белок: NM_139106.2; id гена, содержащего эту мрнк: 245965 (с него синтезируются 5 мрнк, кодирующие разные изоформы этого белка)
последовательность днк, от начала до конца CDS этого белка, координаты: 10211514:10214308; (ncbi gene < id -> graphics -> find < 10211514-10214308)
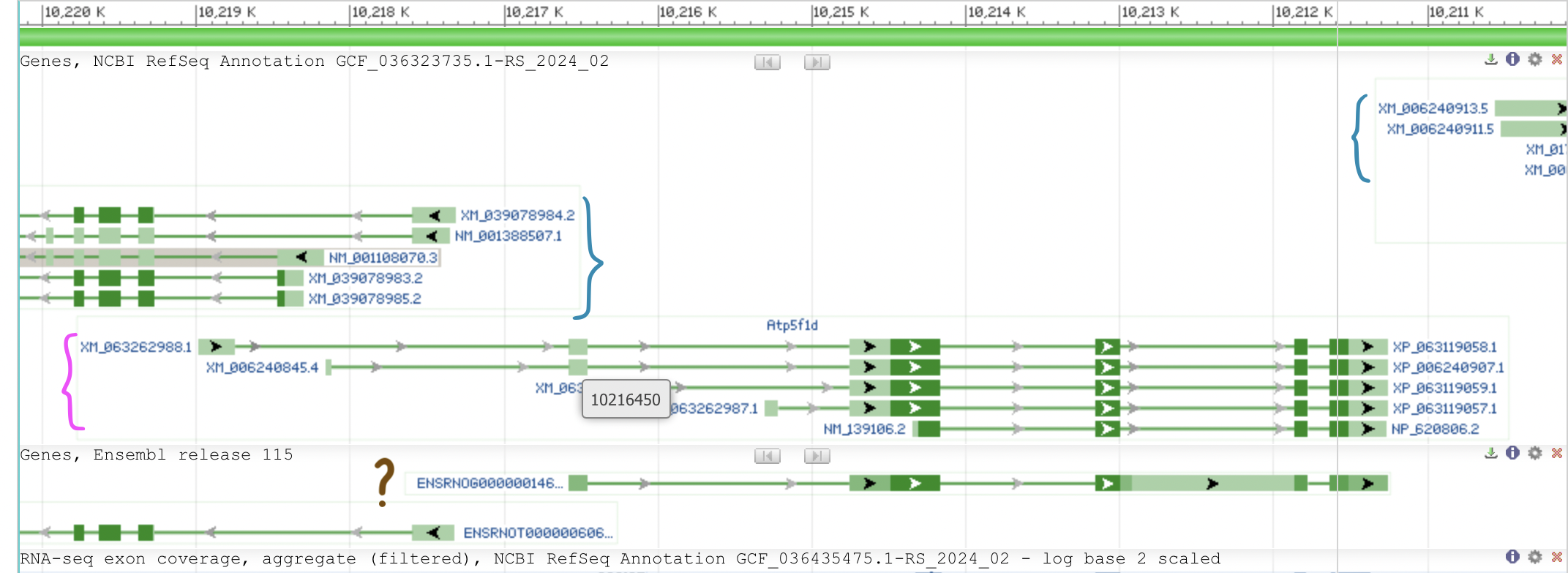
фиолетовые - мрнк, синтезированные с нашего гена, голубые - соседние. квадратиками - экзоны. под знаком вопроса вероятно наш ген, но он почему-то обрезан с одной стороны
Задание 2
Гомологичные последовательности ищем у Apoidea (пчелы), потому что они относительно далеки от крыс и я люблю пчел. база данных refseq_genomes
blastn не нашел совсем ничего, даже при минимальной длине слова 7, минимальных штрафах и e-value<0.05. у пауков тоже ничего не нашел, зато нашел у кошачьих, но их использовать нельзя. видимо последовательности днк слишком сильно разошлись. я попробовал поискать через blastp, и он нашел 19 совпадений, но этого тоже не требовалось. tblastx выдает 51 совпадение, так что дело не в том, что у меня неправильная последовательность днк. я это к тому, что разошлись именно последовательности днк, а не белковые
tblastn выдал 38 результатов. его я использовал, потому что он работает быстрее чем tblastx, и у меня есть последовательность белка. всего кстати в базе данных 38 сборок относящихся к Apoidea. ниже графический результат. видим, что совпадени в основном в середине. видимо в середине находится финкционально важный домен, раз он так похож у далеких организмов

Задание 3
команда для индексирования генома крысы:
makeblastdb -in /Users/macbook/Downloads/ncbi_dataset_genome/ncbi_dataset/data/GCF_036323735.1/GCF_036323735.1_GRCr8_genomic.fna -dbtype nucl
команда на поиск гомологичных с рРНК e.coli участков у крысы
blastn -task blastn -query /Users/macbook/Downloads/rRNA_ecoli.fasta -db /Users/macbook/Downloads/ncbi_dataset_genome/ncbi_dataset/data/GCF_036323735.1/GCF_036323735.1_GRCr8_genomic.fna -outfmt 7 -evalue 0.001 > ecolirat.txt
файл с табличкой
рисунок:
