Учебный сайт Саши Якушевой |
||||
Второй семестр |
||||
Главная |
Семестры |
Обо мне |
Ссылки |
|
|
Освоение навыков работы в BLAST.
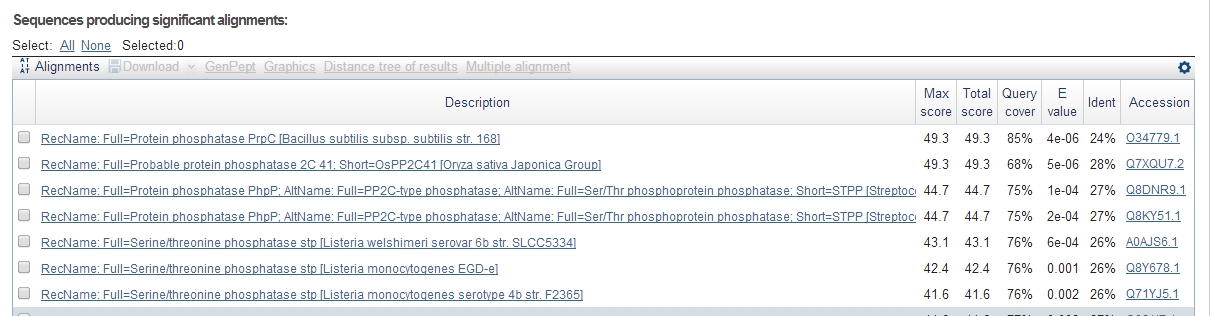
Рисунок 1. Результат поиска в BLAST. Далее нужно было выбрать гомолога, я выбрала белок с AC Q8Y678.1. Serine/threonine phosphatase stp из Listeria monocytogenes EGD-e. С помощью Align two or more sequences в BLAST надо было получить карту локального сходства. 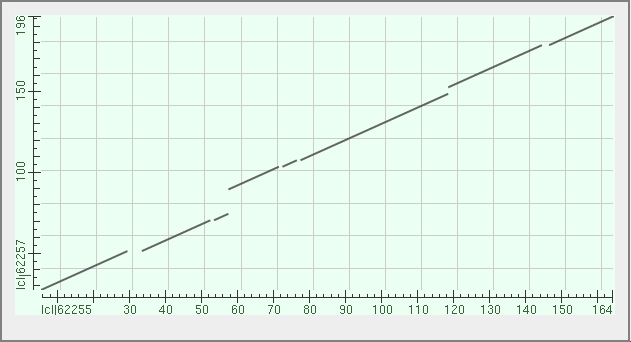
Рисунок 2. Матрица локального сходства. Дальше нужно было проверить, сколько есть гомологов среди эукариот. Для это в качестве параметров на вход в pBLAST надо задать eukaryotes (taxid:2759). В базе данных nr было найдено 100 гомологов. Среди которых 7 были выбраны для дальнейшей работы. 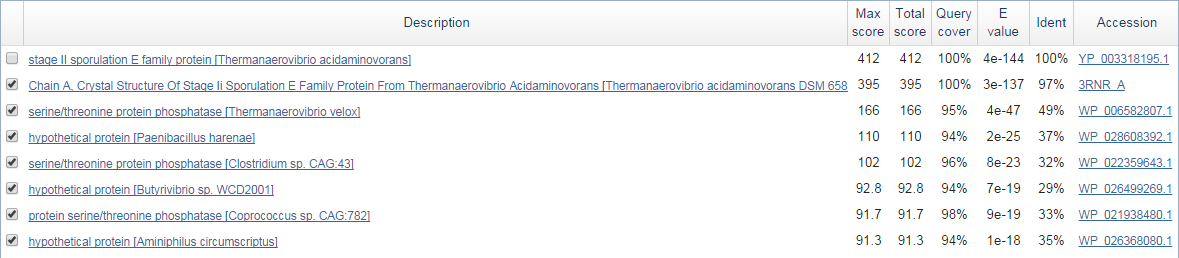
Рисунок 3. Выбранные гомологи. Далее было построено множественное выравнивание и проанализированно с помощью программы Jalview. Результат представлен на рисунке 4, в формате fasta по ссылке и в формате jar по ссылке.  Рисунок 4. Множественное выравнивание, полученное в Jalview. © Саша Якушева, 2013
|