Построение парных выравниваний. Поиск по сходству
Исходные данные
Был дан белок (Sequence ID: YP_184698.1) археи Thermococcus kodakarensis KOD1[1]. Необходимо было выполнить несколько заданий.
Задание 1
Выборка гомологов белка при помощи программы BLASTP из базы Refseq.
Я запустила BLASTP на сайте NCBI с параметрами по умолчанию для аминокислотной
последовательности своего белка.
Результаты поиска можно скачать.
Число находок: 394. Из них 242 находки, гомологичные моему белку по всей длине
(query cover > 80%). При этом для многих из них E-value >1 (например, для
последовательности с идентификатором WP_014560618.1 E-value = 3,6). Это говорит
том, что оценивать гомологичность белков только по длине выравнивания
неправильно. Для трех находок (лучшей, худшей и любой из середины списка) в
таблице 1 приведена краткая характеристика. Видно, что лучшая и худшая находки
очень сильно различаются по достоверности, в то время как находка из середины
списка примерно такая же по характеристикам, что и худшая. При этом худшая
находка имеет E-value 9.8, что показывает недостоверность выравнивания. Это говорит о том,
что BLAST дает излишне много результатов, и отсеивать неподходящие всегда нужно
самому.
Из находок, приведенных в таблице, можно считать
гомологом целой исходной последовательности только одну последовательность
("лучшая находка"). Из всех 394 находок 90 последовательностей можно считать
гомологом целой исходной последовательности. Условный критерий: E-value
< 1e-3 и Query cover > 70%.
| Название белка | Sequence ID: | Длина выравнивания | bit score | % идентичных остатков | % сходных остатков | E-value | |
| Лучшая находка | carbohydrate kinase | WP_062388230.1 | 273 | 498 | 89 | 94 | 9e-178 |
| Худшая находка | hypothetical protein | WP_055865524.1 | 257 | 37.4 | 34 | 43 | 9.8 |
| Находка из середины списка | ribokinase | WP_007174827.1 | 153 | 43.1 | 24 | 44 | 0.13 |
Выравнивания (построенные blastp)
Лучшая находка
 |

Открыть в формате: FASTA, MSF.
Худшая находка
 |
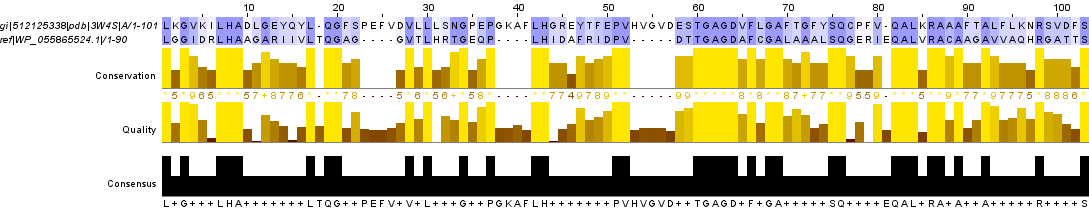
Открыть в формате: FASTA, MSF.
Находка из середины списка
 |

Открыть в формате: FASTA, MSF.
Задание 2
Множественное выравнивание последовательностей из выборки.
Была сохранена выборка из 24 полных последовательностей гомологов моего
белка + его последовательность в формате fasta. Затем было построено
выравнивание в Jalview с помощью программы Muscle (раскраска
BLOSUM62). Для нахождения вертикальных блоков
использовался следующий список групп аминокислотных остатков (на
основании данных о частотах мутаций):
KR ST LIVM FYW DN EQ P G A H С |
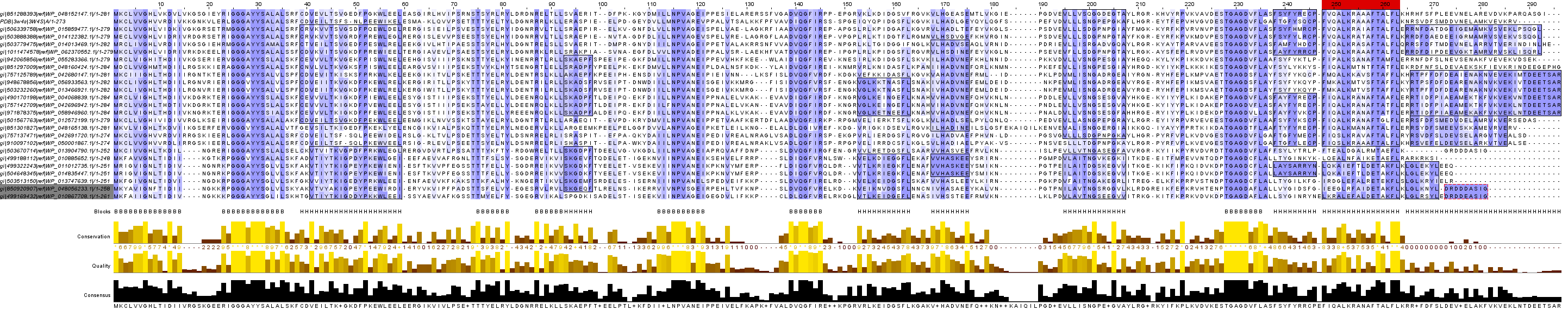
В выравнивании общие для всех последовательностей вертикальные блоки обозначены
"В", а блоки для части последовательностей — "Н" в строке разметки "Blocks".
По выравниванию видно, что блоков для разных групп последовательностей намного
больше, однако и для всех последовательностей их довольно много. Это свидетельствует
о том, что находки гомологичны между собой, а не только исходному белку, что, в
принципе, логично.
На С-конце оказалось много длинных невыровненных участков, различных у разных находок.
Но при более тщательном изучении было найдено множество блоков для групп
последовательностей. Возможно, это обусловлено схожей функцией белков, чьи
последовательности входят в эти группы. На N-конце таких последовательностей
найдено не было. Опять же это можно объяснить тем, что для этих белков консервативность
N-конца важна для выполнения их функций, в то время как С-конец может различаться.
Открыть выравнивание в формате: FASTA,
MSF.
Задания 3, 4
Глобальное и локальное парные выравнивания данного белка и худшей находки из выборки
(ID: WP_010867708.1).
Использовались консольные программы needle и water на kodomo. Для получения парного
выравнивания из построенного множественного были удалены все остальные последовательности.
Также было взято выравнивание, полученное BLAST. Таким образом, четыре полученных
выравнивания:
- глобальное (выданное needle)
- глобальное (полученное из множественного)
- локальное (выданное water)
- локальное (выданное BLAST)
были помещены в одно окно в JalView и объединены в 4 соответствующие группы. Затем было
сделано выравнивание полученных выравниваний между собой. Для этого в JalView смещались
обе последовательности в соответствующих группах. Колонки, которые одинаковы в разных
выравниваниях находятся друг под другом. В некоторые выравнивания были вставлены гэпы.
Группы следуют в вышеуказанной очередности.
Участок, найденный программами BLAST и water (4 и 3 группа соответственно), совпал неполностью.
Выравнивания, полученные этими программами, различаются на участках, где консервативных
нет блоков, т.е. консервативные колонки могут быть получены по-разному, что вполне нормально.
Тем не менее, длина выравнивания и вертикальные блоки совпали достаточно хорошо, что говорит
о взаимозаменяемости этих программ. Такие участки для удобства в проекте Jalview отмечены *
в строке разметки "colons".
В то же время, абсолютно совпали выравнивания, полученные программами needle и water (не считая
того, что программа water не дала на выход недостоверно выравненный С-конец). Хуже всего с
остальными совпадает выравнивание, полученное удалением из множественного выравнивания лишних
последовательностей, что и ожидалось.
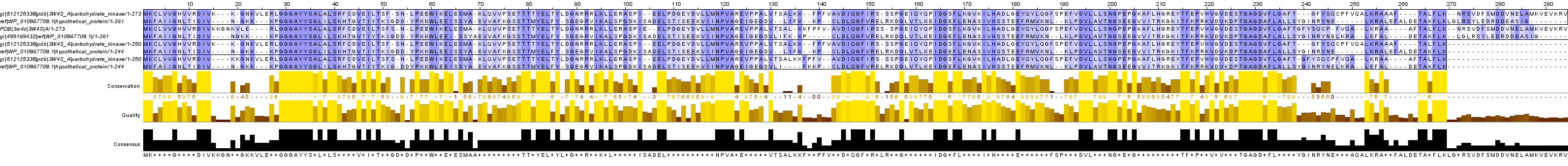
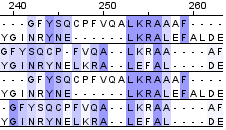 |
Как уже упоминалось, в общем выравнивании были участки, на которых выравнивания различаются. Один из них приведен на рис.1. Его длина составляет 25 а.о. Это связано с большим числом гэпов и маленьким количеством корсервативных колонок, которые могут быть получены при разных выравниваниях. Поэтому такие участки не несут достоверной информации о гомологичности.
Задание 5
Парные выравнивания последовательностей двух заведомо негомологичных белков.
Для этого задания в пару моему белку был взят негомологичный белок. Краткую информацию о
нем можно узнат из таблицы 2.
| Параметр | Значение |
| Идентификатор белка | YP_003134604.1 |
| Идентификатор GI | 257056772 |
| Идентификатор генома | NC_013159 |
| Координаты гена в геноме | 3029789..3030703 |
| Длина гена (в парах нуклеотидов) | 915 |
| Цепь | Прямая |
| Длина белка (в аминокислотных остатках) | 304 |
Для общего выравнивания использовалось два выравнивания, полученных при помощи программ water и needle. Далее вставлялись гэпы для выравнивания их выравниваний относительно друг друга. Ниже приведен участок, содержащий общие колонки (различающиеся N- и C-концы можно посмотреть в проекте). Видно, что общих колонок практически нет, т.е. выравнивания существенно различаются, что говорит об их недостоверности. Из этого можно сделать тот же вывод, что и из различия в выравниваниях на участках с большим числом гэпов (см. предыдущее задание): слабо гомологичные последовательности (участки последовательностей) выравниваются неточно и разными алгоритмами (программами) по-разному. А значит, в этом случае найденные колонки не свидетельствуют ни о чем конкретном. Опять же: все нужно анализировать самому, так как программа на это не способна.

Проект Jalview со всеми заданиями: скачать.
Примечания:
[1] Информацию о белке можно прочитать
здесь.