Информация о белке с идентификатором WP_011251235.1
| Uniprot ID | Uniprot AC | Refseq ID | PDB ID | Длина | Молекулярная масса | Рекомендуемое Unirpot название |
| Q5JDA3_THEKO | Q5JDA3 | WP_011251235.1 | 3W4S, 4XF6, 4XF7 | 273 а.о. | 30309 Да | Carbohydrate/pyrimidine kinase, PfkB family |
Дополнительная информация о белке:
- структура известна для всего белка;
- в нем две цепи: А и В, которые имеют одинаковое строение;
- кодируется геном TK2285 (локус TK_RS11510);
- функции: 1)катализ переноса фосфатной группы на молекулу субстрата
(обычно
с АТФ) и 2)селективное связывание нуклеотидов; - организм: архея Thermococcus kodakarensis ;
- идентификатор нуклеотидной последовательности RefSeq: NC_006624.1;
- PDB: 3W4S, 4XF6, 4XF7;
- последнее редактирование записи: 15.02.2005.
| Кластер | Uniref50 | Uniref90 | Uniref100 |
| Cluster ID | UniRef50_Q5JDA3 | UniRef90_Q5JDA3 | UniRef100_Q5JDA3 |
| Число белков в кластере | 13 | 1 | 1 |
| Идентичность последовательностей | 50% | 90% | 100% |
| Организмы | Thermococcus kodakarensis KOD1 Thermococcus guaymasensis DSM 11113 Thermococcus sp. AM4 Thermococcus sp. EP1 Thermococcus nautili Pyrococcus yayanosii CH1 (JCM 16557) Thermococcus onnurineus NA1 Thermococcus eurythermalis Thermococcus sp. CGMCC 1.5172 / 4557 Thermococcus cleftensis Thermococcus gammatolerans Thermococcus zilligii |
Thermococcus kodakarensis KOD1 | Thermococcus kodakarensis KOD1 |
Анализ результатов. Во втором столбце таблицы 1 приведены результаты поиска
кластеров, включающих последовательности, идентичные на 50%. Как мы видим,
белков, удовлетворяющих этому условию, нашлось немного: всего 13. При этом все кроме
одного принадлежат роду Thermococcus, что говорит о том, что данная карбогидрат-киназа
характерна, возможно, практически только этому роду. Кроме того, род Pyrococcus
относится к тому же семейству Thermococcaceae, что и род Thermococcus, что опять
свидетельствует в пользу характерности данной карбогидрат-киназы только определенным организмам
(в частности, семейству Thermococcaceae).
В столбцах №2 и 3 приведены результаты поиска кластеров при идентичности последовательностей
90% и 100% соответственно. Оказалось, что последовательностей настолько идентичных
нашей исходной в базе данных нет.
| Поиск | Запрос | Нашлось белков всего | Из них в разделе Reviewed |
| 1 поиск по названию | name:carbohydrate | 33.435 | 127 |
| 2 поиск по названию | name:"pyrimidine kinase" | 130 | 0 |
| 3 поиск по названию | name:"pyrimidine kinase, pfkb family" | 11 | 0 |
| Поиск по тому же названию среди белков из организмов того же семейства | name:"pyrimidine kinase" taxonomy:"Thermococcaceae [2259]" | 14 | 0 |
| Поиск по тому же названию среди белков из организмов того же отдела | name:"pyrimidine kinase" taxonomy:"Euryarchaeota [28890]" | 14 | 0 |
| Поиск по названию "лизоцим" | name:lysozyme | 16.824 | 224 |
| Поиск по названию "лизоцим" в таксоне зеленые растения | name:lysozyme taxonomy:"Viridiplantae [33090]" | 16 | 3 |
| Поиск по названию "лизоцим" в таксоне животные | name:lysozyme taxonomy:"Metazoa [33208]" | 1.305 | 148 |
| Поиск по названию "трипсин" | name:trypsin | 11.159 | 301 |
| Поиск позволяющий определить, сколько среди находок предыдущего поиска белков, анонсированных как ингибиторы трипсина | name:"trypsin inhibitor" | 2.558 | 203 |
Анализ результатов. В таблице 2 приведены результаты поиска в UniProt с различными условиями.
Сначала шел поиск для моего белка, ограниченный по семейству, а потом и по отделу.
Затем
был осуществлен поиск для лизоцима. Лизоцим — антибактериальный агент, фермент класса гидролаз,
разрушающий клеточные стенки бактерий путём гидролиза пептидогликана клеточной стенки бактерий муреина.
Содержится в слизистой животных, также найден в растениях, некоторых бактериях и вирусах.
То, что он намного чаще встречается у животных, видно по поиску.
Примечательно, что карбогидрат-киназ было найдено 33.435, а лизоцима намного меньше — 16.824. Однако
127 записей о карбогидрат-киназах находятся в разделе Reviewed (т.е. записи составляются и редактируются вручную, а не компьютером),
в то же время о лизоциме в этом разделе находятся целых 224 записи. Мне кажется, это может быть связано с тем, что лизоцим
лучше изучен или вызывает больший интерес у исследователей, чем карбогидрат-киназы. Интересно, что при запросе в
Google "лизоцим" сразу выходит много ссылок по теме, в то время как по запросу "карбогидрат-киназа" мы получаем
ссылки на страницы, где данный фермент упомянут один-два раза в статье по какой-то другой теме.
Последний поиск показал, что всегда нужно четко формулировать запросы. Для запроса "trypsin" было получено 301 результатов,
203 из которых относились к ингибитору трипсина, а не к нему самому.
4(*) Были найдены две записи, соответствующие моему белку:
RefSeq Protein WP_011251235.1
и UniProt Q5JDA3.
В RefSeq в отличие от UniProt в разы меньше информации о белке
(например, не указана масса, нет никакой информации о PDB,
о кодирующем гене, дисульфидных мостиках). Таким образом,
RefSeq представляет собой сжатое описание белка, содержащее
информацию в основном о сайтах связывания, небольшую характеристику,
последовательность и краткие комментарии.
Вывод: база данных Refseq содержит сжатое описание белка, в целом включающее всю
необходимую для недетального исследования информацию. Это позволяет получить представление
об объекте в целом. Если же нужна более детальная и подробная
информация, лучше использовать базу данных UniProt.
5(*) Была просмотрена история для моего белка, ее можно увидеть перейдя по
этой ссылке.
Можно заметить,
что с 25-й версии (15.06.2010) номер начинают обозначать по-другому
(ср. 15.15/40.15 и 2010_07/2010_07). Кроме того, с 43-й версии (13.11.2013)
название изменяется с Q5JDA3_PYRKO на Q5JDA3_THEKO.
Рабочая версия UniProt является 59-й по счету. Самая первая была опубликована
15.02.2006, а последняя — 16.03.16. Каждый год появляется
обновленная версия (от двух до восьми за весь год). В самой первой
версии информации было совсем немного, затем она постепенно накапливается.
В конце концов, обновляется в основном информация по коду PDB,
добавляются комментарии и т.п.
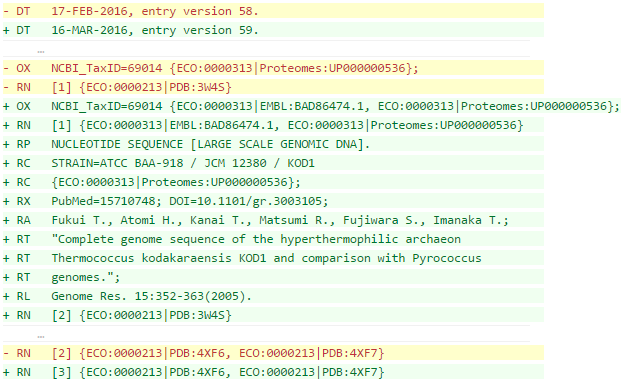
На рис.1 в качестве примера приведено
приведено сравнение версий №58 и №59 (посмотреть на
сайте).
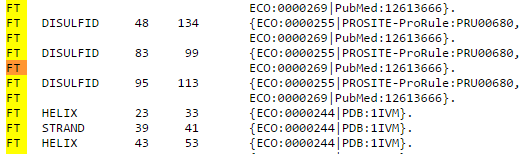
На рис.2 приведена часть от сравнения версий №1 и №59 (полное сравнение можно посмотреть
здесь).
 |
 |
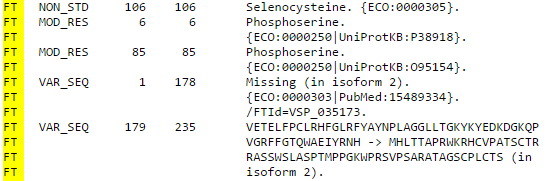
6(*) - Нестандартные аминокислоты указываются в разделе FT (Features), подразделе Amino acid modifications / Non-standard residue. Селеноцистеин
может быть обозначен как 'U', пирролизин — 'O'. При этом селеноцистеин может быть указан еще и в подразделе Sites / Active site,
если он входит в состав активного центра. На рис.2 приведен пример: в формате text в строке FT NON_STD указан Selenocysteine;
- Посттрансляционные модификации указываются в разделе FT (Features) в подразделе Amino acid modifications / Glycosylation и др.
На рис. 2 в строке FT MOD_RES указан Phosphoserine как модификация серина;
- Дисульфидные мостики указываются в разделе FT (Features) в подразделе Amino acid modifications / Disulfide bond.
На рис. 1 приведен пример: в строке FT DISULFID указана дисульфидная связь;
- Альтернативные последовательности в разделе FT (Features) в подразделе Natural variations / Alternative sequence.
На рис.2 приведен пример: в строке FT VAR_SEQ указан вариант последовательности для изоформы 2.
 |
 |
Примечания: