Изображение выравниваний
Описание проделанной работы
Мне было дано шесть аминокислотных последовательностей c UniProt ID:
- GCSH_RHIEC
- B2B5Y8_PODAN
- R5IES6_9FIRM
- GCSH_BURXL
- A7I5E3_METB6
- M3XG19_FELCA
Среди них пять белков гомологичны, а один — не гомологичен остальным.
Для выявления последнего необходимо было построить несколько множественных выравниваний
с помощью редактора Jalview.
Множественное выравнивание последовательностей — выравнивание трёх и
более последовательностей (белков, ДНК или РНК), используя которое, можно оценить
эволюционное происхождение последовательностей. Сходство первичных структур белков
кроме того может отражать их функциональные или структурные взаимосвязи.
Я не стала скачивать
последовательности непосредственно на компьютер,
а воспользовалась прямой загрузкой последовательностей
в редактор из базы данных UniProt (File => Fetch sequences). Для построения
выравнивания использовалась программа Muscle with Defaults.
О гомологичности белков судят по сходству последовательностей.
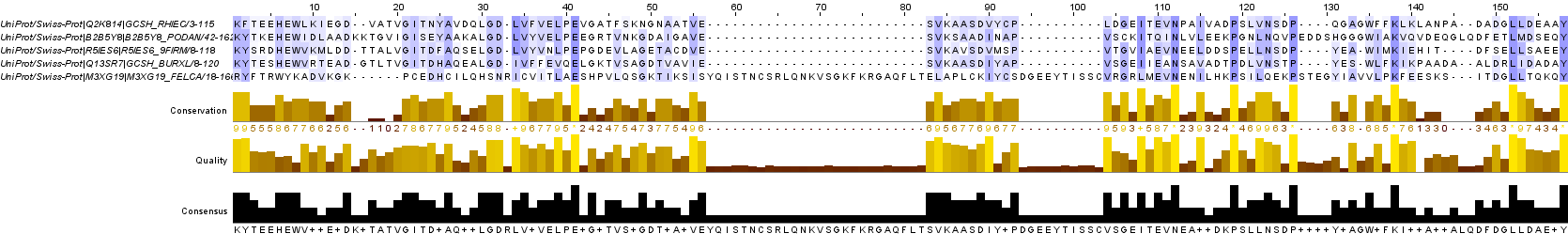
В первом задании выравнивание выполнялось для всех шести последовательностей,
во втором — для пяти гомологичных.
Результаты
Определить лишнюю последовательность оказалось не так-то просто: я сомневалась между A7I5E3_METB6 и M3XG19_FELCA,
так как обе эти последовательности довольно сильно отличаются от четырех остальных. Но, так как удаление A7I5E3_METB6
дало появление большего числа консервативных колонок (ср.
выравнивания без обеих последовательностей по отдельности ), именно эта последовательность была определена как лишняя.
Также были удалены N- и C- концевые участки, которые не были гомологичны.
На рис. 1 изображено полученное выравнивание (раскраска по схеме BLOSUM62 с порогом по
консервативности 30%). Для того, чтобы открыть изображение в большом размере, нажмите на него.
Данные, полученные с помощью выравнивания на сайте
UniProt, подтверждают полученные результаты и неоднозначность
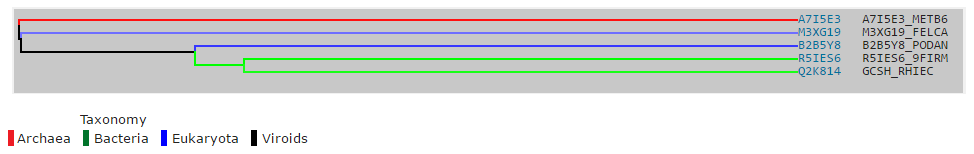
выбора лишней последовательности (см. рис. 2). В то же время
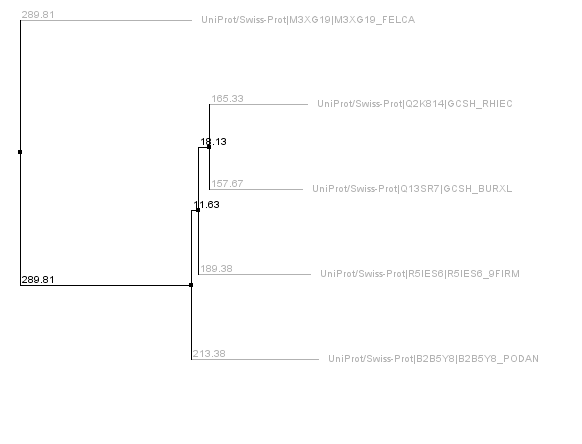
дерево, полученное с помощью Jalview, свидетельствует совершенно
о другом родстве между последовательностями.
Мой выбор лишней последовательности был основан на количестве
консервативных колонок при исключении этой последовательности из выравнивания.
Тем не менее, я бы вообще исключила обе последовательности с ID A7I5E3_METB6 и M3XG19_FELCA,
т.к. четыре остальных явно гомологичны между собой.
 |
 |
Выравнивание в других форматах: FASTA,
MSF.
Проект JalView с выравниванием шести последовательностей и с выравниванием пяти гомологичных последовательностей
в раскрасках BLOSUM62 и ClustalX: скачать.