

 |
 |
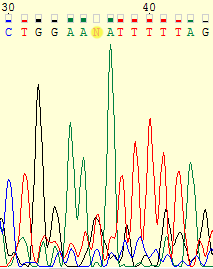
| Рассмотрим самое начало последовательности. На
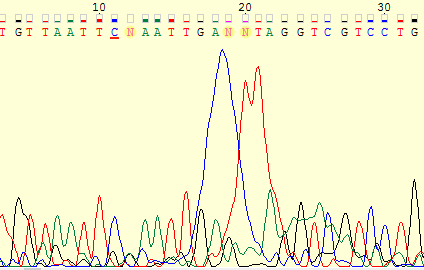
хроматограмме прямой цепи не определены нуклеотиды №12, 19, 20. Для
нуклеотида №12 сигнал настолько слаб, что сливается с шумом, из-за чего он
не может быть определен. В то же время на хроматограмме обратной цепи сигнал
этого нуклеотида достаточно сильный, а качество сигнала, как мне кажется,
достаточно, для того, чтобы считать гуанин верным нуклеотидом. Далее в прямой
цепи не определены нуклеотиды №19, 20 из-за очень сильного постороннего
сигнала (пятна краски?). Однако на хроматограмме обратной цепи вполне
приемлимые два сигнала, показывающие, что на этих позициях находятся гуанин
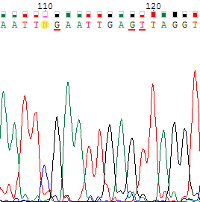
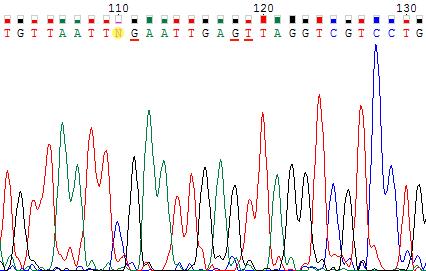
и тимин. И, наконец, для обратной цепи нуклеотид №110 также не был определен
из-за слабого сигнала, сравнимого с шумом. Для прямой цепи программа
определила нуклеотид, как цитозин, хотя сигнал, как и его качество, не особо
хорошие. Таким образом, были заменены в общей сложности четыре нуклеотида. При этом хочется отметить, что хроматограмма для прямой цепи может считаться верной только при сопоставлении с лучшей хроматограммой обратной цепи, т.к. для нее шум слишком силен и качество сигналов слишком низкое, чтобы считать определенную последовательность 100% верной. Например, на участке с 16 по 28 нуклеотид хроматограмма вообще бесполезна для прочтения последовательности. В нашем случае, однако, сопоставление показало, что последовательность определена верно. Хотя плохая хроматограмма в обоих случаях может порождать сомнения. |
|
 |
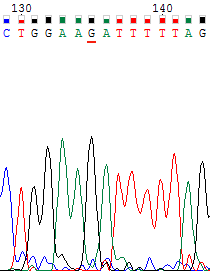 |
| На хроматограмме прямой цепи нуклеотид №35 не определен, т.к. сигнал слился с шумом. В то же время на хроматограмме обратной цепи сигнал достаточно хорошего качества и сам пик довольно четкий и высокий (=> сигнал сильный). Поэтому для прямой цепи была сделана замена на гуанин. Также хочется отметить, что несмотря на относительно низкий уровень шума на хроматограмме обратной цепи, многие пики не разделены до конца. Тем не менее, качество сигнала этих участков хорошее, и для прямой цепи нуклеотиды были определены так же, поэтому делаем вывод, что все ок. | |
 |
 |
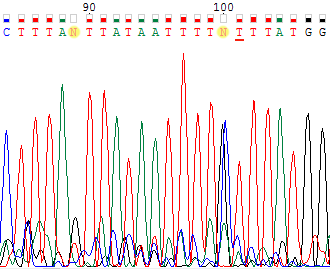
| Для прямой цепи не определены 2 нуклеотида: №89 и №100. И если 89-й нуклеотид вполне верно судя по качеству сигнала определен как гуанин на хроматограмме для обратной цепи судя (поэтому в прямой цепи N было заменено на g), нуклеотид №100 не определен и для обратной цепи. В хроматограмме прямой цепи мы видим практически одинаковые по силе сигналы от цитозина и гуанина, в то время как на хроматограмме для обратной цепи в этой позиции более сильный сигнал от гуанина и более слабый — от тимина. Так как во втором случае сигнал от цитозина сильнее и так как сигнал от цитозина присутствует в обеих хроматограммах, вместо N была поставлена c. Причина плохого качества сигнала в этой позиции в обеих хроматограммах точно не ясна; возможно, это связано с загрязнением или высоким уровнем шума (который достаточно высок и для соседних позиций). Данное событие (одна и та же позиция не определилась одновременно для обеих цепей), по идее, должно происходить достаточно редко. | |
 |
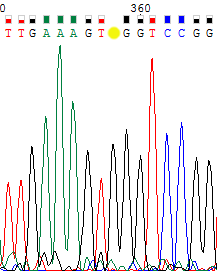 |
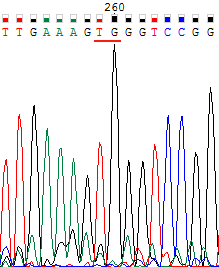
| В этот раз все довольно однозначно. На хроматограмме обратной цепи гуанин № 358 почему-то не был определен программой, хотя пик достаточно высокий при небольшом уровне шума. Для прямой цепи этот нуклеотид был определен как гуанин, поэтому в обратной цепи без сомнений был вставлен g. | |
 |
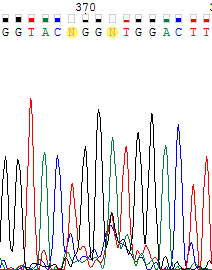 |
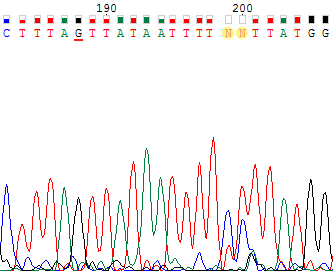
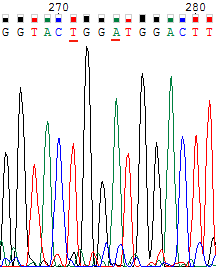
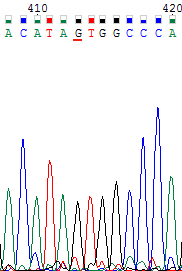
| На хроматограмме справа для обратной цепи на рассматриваемом участке произошло что-то странное. Несмотря на это не определены только два нуклеотида: №369 и 372. Т.к. для прямой цепи оба этих нуклеотида определены с хорошим качеством сигнала как тимин и аденин соответственно, для обратной цепи было сделано две замены. | |
 |
 |
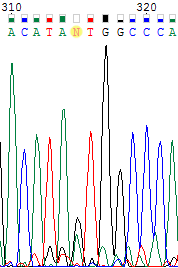
| В этот раз, как мне кажется, все тоже понятно. Несмотря на сравнимые пики для 315-го нуклеотида прямой цепи, на хроматограмме обратной цепи тот же нуклеотид определен как гуанин, поэтому была сделана соответствующая замена. | |
 |
 |
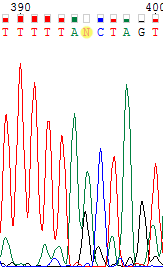
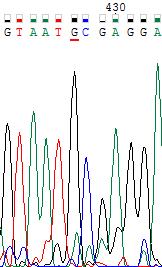
| Такая же ситуация, как в предыдущем случае. 395-й неопределенный нуклеотид прямой цепи был заменен на a. | |
 |
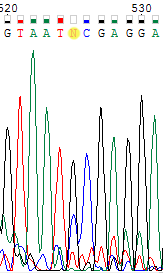 |
| Аналогично: неопределенный 525-й нуклеотид обратной цепи был заменен на g. Хочется тут также отметить, что шум начинает постепенно усиливаться, а сила сигнала падать. | |
 |
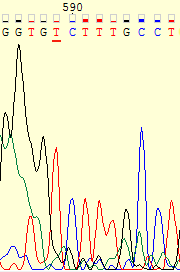 |
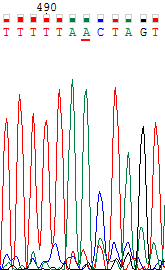
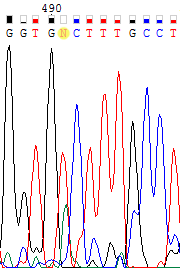
| Можно заметить, что хроматограмма обратной цепи резко ухудшается, и становится все больше неопределенных нуклеотидов. В то же время хроматограмма прямой цепи сохраняет примерно то же качество. Здесь была сделана одна сомнительная замена 491-го нуклеотида прямой цепи на тимин. Сомнительная она потому, что качество сигнала для обратной цепи очень низкое, несмотря на относительно высокий пик. В общем, так как близится конец хроматограммы, качество ее ухудшается, и, соответственно, возможны ошибки в определении нуклеотидов. | |