Нуклеотидные базы данных
 |
| Рис. 1.
Тутовый шелкопряд1 |
Задание 1. Для выполнения задания 1 была выбрана бабочка тутового шелкопряда
(
Bombyx mori) — из семейства настоящие шелкопряды, играющая
важную экономическую роль в получении шелка. Для этого разводятся шелковичные черви
(гусеницы тутового шелкопряда) специальной породы, у которой белый цвет кокона. После
образования кокона, его держат несколько часов при высокой температуре для того,
чтобы убить куколку и облегчить раскручивание кокона. Помимо этого в Китае и Корее
куколки тутового шелкопряда употребляются в пищу, а сушеные гусеницы применяются в
китайской народной медицине
2.
Систематика:
Домен: Эукариоты
Царство: Животные
Тип: Членистоногие
Класс: Насекомые
Отряд: Чешуекрылые
Семейство: Настоящие шелкопряды
Род: Bombyx
Вид: Bombyx mori
На сайте NCBI по ссылке Browse by organism на странице базы данных Genome был выбран
этот вид. Ниже в табл. 1 представлен небольшой отчет.
Таблица 1. Отчет по заданию 1
| Число сборок генома |
4 |
| Число проектов по секвенированию |
4 |
| Число образцов по секвенированию |
4 |
| Характеристика сборки GCA_000151715.1 |
| Описание образца (BioSample ID: SAMN02952907) |
пол: мужской
штамм: Dazao
|
| Описание проекта (BioProject ID: PRJNA13125) |
Геном Bombyx mori секвенировали в Southwest Agricultural
University в Китае с использованием метода WGS (дата регистрации:
18.10.2004). Номер доступа к проекту: ADK00000000; данная версия
проекта имеет номер доступа: ADK01000000 и состоит из
последовательностей
ADK01000001-AADK01066482. Геномные скэффолды представлены контигами
H379587-CH396048. ДНК для секвенирования генома была выделена из
одомашненной породы, Dazao, которую используют для биологических
и генетических исследований во всем мире. ДНК экстрагировали на
третий день из ткани задней шелковичной железы, полученной из 1225
мужских особей. |
| Контиги* |
общее число: 66 490
***N50: 11 516
****L50: 10 102
самый длинный контиг Ctg000001, длина 99 563
самый короткий контиг Ctg079500, длина 174
таблица доступна по ссылке |
| Скэффолды** |
общее число: 23 156
N50: 26 940
L50: 4 598
(т.к. отдельной таблицы нет, данные взяты из общей таблицы; также можно использовать результаты
поиска, но в нем
вместо 23 156 скэффолдов найдено почему-то только 16 462) |
*
Контиг – секвенированный без пропусков фрагмент ДНК;
**
Cкэффолд –
набор контигов, про который доказано, что контиги в ДНК идут по одной цепи строго
друг за другом;
***как найти
N50: упорядочим контиги по длине по убыванию,
найдем первый контиг такой, что он и все более длинные покрывают более половины
генома, длина этого контига и есть N50;
****как найти
L50: посчитаем,
сколько контигов покрывает половину генома; это и есть L50.
Задание 2. В этом задании было необходимо описать десять ключей, используемых
в таблицах особенностей (Feature Key или Feature table). Результаты задания приведены
в таблице 2.
Таблица 2. Некоторые Feature Keys
| Название |
Описание |
Пример |
| ncRNA |
англ. a non-protein-coding gene — не кодирующий
белок ген, результатом
транскрипции которого является особый тип РНК, отличный от рРНК и
тРНК. |
ncRNA 122493..122706
/ncRNA_class="other"
/gene="CR40469"
/locus_tag="Dmel_CR40469"
/gene_synonym="Dmel\CR40469"
/product="CR40469"
/note="putative non-coding RNA;
CR40469-RA; Dmel\CR40469-RA"
/db_xref="FLYBASE:FBgn0058469" |
| precursor_RNA |
англ. precursor RNA — предшественник РНК. Любая
разновидность незрелой РНК; может включать нкРНК, рРНК, тРНК, 5'UTR,
CDS, экзоны, интроны, 3'UTR.
|
precursor_RNA 133385..133502
/gene="mir-4915"
/locus_tag="Dmel_CR43552"
/gene_synonym="CR43552"
/product="mir-4915 precursor RNA"
/note="mir-4915-RM; Dmel\mir-4915-RM; CR43552-RM;
Dmel\CR43552-RM"
/db_xref="miRBase:MI0017697" |
| misc_RNA |
Любые транскрипты, которые не подходят под определения
других feature keys для РНК (prim_transcript, precursor_RNA, mRNA,
5'UTR, 3'UTR, exon, CDS, sig_peptide, transit_peptide, mat_peptide,
intron, polyA_site, ncRNA, rRNA and tRNA)
|
misc_RNA join(539636..540073,540849..540956,541819..543839)
/gene="LOC107971886"
/product="uncharacterized LOC107971886, transcript variant X3"
/note="Derived by automated computational analysis using
gene prediction method: Gnomon. Supporting evidence
includes similarity to: 1 mRNA, 1 EST, and 75% coverage of
the annotated genomic feature by RNAseq alignments"
/transcript_id="XR_001714724.1"
/db_xref="GeneID:107971886"
|
| repeat_region |
Участки генома, содержащие повторы. |
repeat_region 22894..23276
/locus_tag="KLLA0C00308t"
/old_locus_tag="Klla0C.LTR.2"
/rpt_type=long_terminal_repeat
|
| mobile_element |
Участки генома, содержащие подвижные элементы. |
mobile_element 841364..850816
/note="uniprot|Q9C0U1 Yarrowia lipolytica Ylt1 LTR-
retrotransposon"
/mobile_element_type="retrotransposon"
|
| STS |
англ. sequence tagged site —
короткая последовательность ДНК, которая может быть определена с
помощью ПЦР; карта участка генома может быть построена с помощью
определения порядка расположения STS.
|
STS 2050089..2050757
/standard_name="ha2600"
/db_xref="UniSTS:515570"
|
| old_sequence |
Приведенная последовательность корректирует предыдущую версию
последовательности.
|
old_sequence 4086
/citation=[3]
/replace="c"
|
| D-loop |
англ. displacement loop — петля смещения. Область
в митохондриальной ДНК, в которой короткая вытянутая РНК
взаимодействует с одной из цепей ДНК, замещая комплементарную цепь
ДНК в этой области. Также D-loop описывает замещение участка одной
цепи дуплекса ДНК каким-либо другим одноцепочечным фрагментом в
реакции, катализируемой белком RecA.
|
D-loop 15715..16825
/note="control region"
|
| misc_binding |
Сайт в нуклеиновой кислоте, который ковалентно или нековалентно
взаимодействует с другим участком; не может быть описан другими
feature key (primer_bind or protein_bind).
|
misc_binding 111478..111597
/inference="COORDINATES: nucleotide
motif:Rfam:12.0:RF00059"
/inference="COORDINATES: profile:INFERNAL:1.1.1"
/note="TPP riboswitch; Derived by automated computational
analysis using gene prediction method: cmsearch."
/bound_moiety="thiamine pyrophosphate"
/db_xref="RFAM:RF00059"
|
| J_segment
| Сегмент тяжелой и легкой цепи иммуноглобулина и альфа-, бета- и
гамма-цепей Т-клеточного рецептора.
|
J_segment 328..393
/gene="TCR1A" |
Задание 3. В этом задании было необходимо описать состояние дел в одном из
массовых геномных проектов. Ниже представлена информация о выбранном проекте.
- Название проекта: NIH Human Microbiome Project (HMP) Roadmap
Project
- Описание и цель: микробиом человека описывает сообщество
микроорганизмов, которые живут внутри организма человека или на поверхности
его тела.
Это сообщество до последнего времени не было достаточно хорошо изучено,
поэтому был создан
проект, цель которого — обеспечить необходимые данные для полного
описания микробиома человека и анализа его влияния на здоровье и заболевания.
Обычно микроорганизмы изучались при культивировании в лаборатории. При
этом большую часть видов микроорганизмов не удавалось выделить в
основном из-за зависимости развития и
роста микроорганизмов от специфических условий,
которые было невозможно создать в лаборатории. Появление новых технологий
секвенирования ДНК дало начало новой области, метагеномике, которая
сделала возможным
изучение микробных сообществ без необходимости культивации. НМР используют
этот подход в дополнение к генетическому анализу уже доступных штаммов, в
конечном итоге получая совершенно новую информацию о сложном устройстве
этого сообщества микроорганизмов. Таким образом, в центрах HMP исследователи
просеквенируют геномы микроорганизмов, ранее выделенных из организма
человека. Вдобавок, образцы, взятые из ЖКТ, ротовой и носовой полостей,
женского мочеполового тракта и с поверхности кожи у волонтеров, будут
просеквенированы для анализа 16S рРНК и учтены при метагеномном анализе.3,4
- Дата регистрации проекта: 2.01.2010
- Ссылки: официальный сайт проекта
- Организация и страна:
NIH Roadmap for Biomedical Research; США.
- Последняя публикация по проекту: Biogeography and
individuality shape function in the human skin metagenome. Nature,
2014 Oct 2;514(7520):59-64. Всего статей: 52
- Секвенировано геномной ДНК/РНК: 594364
- Примечания: проект делится еще на 4 подпроекта:
1) Human Microbiome Project (HMP) 16S rRNA Gene Diversity (BioProject ID:
PRJNA48489);
2) Project (HMP) Demonstration Projects (BioProject ID:
PRJNA46305);
3) Human Microbiome Project (HMP) Metagenome Projects (BioProject ID:
PRJNA43017);
4) Human Microbiome Project (HMP) Reference Genomes (BioProject ID:
PRJNA28331).
Задание 4. В этом задании необходимо было составить таблицу митохондриальных
генов одного из организмов указаного таксона. Мне был дан таксон Haptophyceae.
Сначала нужно было найти полные митохондриальные геномы представителей таксона.
Всего было найдено 5 полных митохондриальных геномов; из них RefSeq: 1, GenBank: 4.
Для поиска использовался запрос
"Haptophyceae"[Organism] AND mitochondrion[Filter] AND complete[Title] AND (genome[All Fields] OR sequence[All Fields])
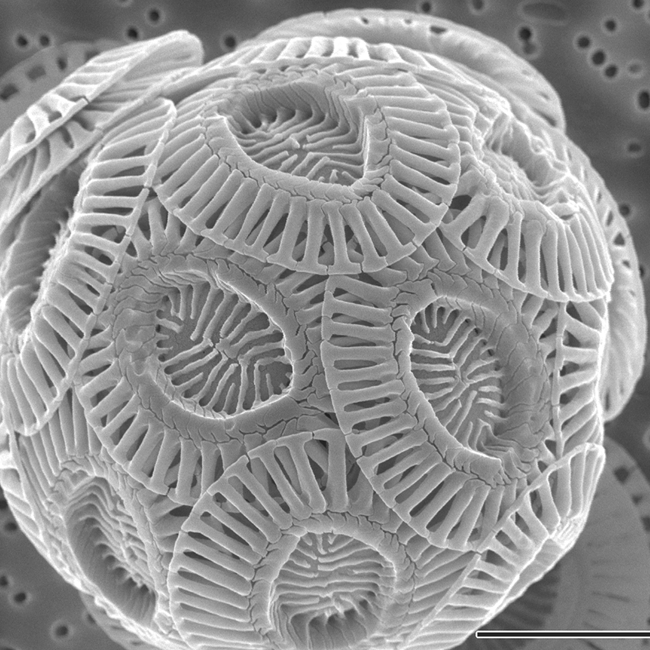 |
| Рис. 1. Электронная фотография
Emiliania huxleyi |
Далее нужно было составить таблицу митохондриальных генов одного из организмов
указанного таксона. Для выполнения этого пункта был выбран организм
Emiliania huxleyi.
Emiliania huxleyi — один из доминирующих
видов, составляющих фитопланктон; распространен от зоны тропиков до зоны субтропиков.
Это одноклеточный организм, покрытый прозрачными и в основном бесцветными дисками из
кальцита (кокколитами), который обеспечивает эффективное преломление света в толще
воды. За счет
этого цветение воды из-за размножения
E.huxleyi хорошо видно даже
из космоса. Такое цветение вызывает увеличение концентрации карбоната кальция и
диметилсульфида, что может оказывать влияние на изменение климата. Кроме того,
кокколиты, а также некоторые части клетки образуют устойчивые к разложению морские
отложения
5.
Систематика:
Домен: Eukaryota
Царство: Chromalveolata
Тип: Haptophyta
Класс: Prymnesiophyceae
Порядок: Isochrysidales
Семейство: Noelaerhabdaceae
Род: Emiliania
Вид: E. huxleyi
Из записи с выбранной последовательностью генома можно перейти на страницу генов по
ссылке gene в разделе Related information. Далее список всех генов был скачан
(send to > File) с сортировкой по порядку в геноме (Sort by chromosome). Скачанный
файл был отредактирован в Excel.
Результат можно скачать:
table.xlsx.
Задание 5. В этом задании необходимо было заполнить таблицу размеров некоторых
геномов из презентации. В качестве размера генома была выбрана длина (total length)
генома представителя (Representative genome), а не median total length. Также в
таблице не указаны "типичные" размеры геномов (для всех, кроме вироидов), так
как их не определить из-за
сильных различий в размерах геномов у разных представителей одного таксона. Еще нужно
отметить, что в случае вирусов сравнивались все группы, в том числе и
вирусы-сателлиты, и вирусы без определенной классификации. В табл. 3
приведены результаты выполнения задания 5.
Прим. При поиске по организму были исключены сборки, содержащие только
контиги и скэффолды. Для
Corbicula fluminea представление генома
оказалось частичным, причем завершение сборки авторами не ожидается. Из таблицы
этот вид я убрала.
Таблица 3. Некоторые геномы из презентации
| Организм |
Размер генома (Mb) |
| Эукариоты |
| Encephalitozoon romaleae [минимальный размер] |
2.18759 |
| Pinus lambertiana (сосна Ламберта) [максимальный
размер] |
27602,7 |
| Yarrowia lipolytica |
20,5509 |
| Octopus bimaculoides (Калифорнийский
двупятнистый осьминог) |
2338,19 |
| Homo sapiens (Человек) |
3238,44 |
| Mus musculus (Домовая мышь) |
2807,72 |
| Intoshia linei |
41,6031 |
| Schizophyllum commune |
38,482 |
| Вироиды |
| Coconut cadang-cadang viroid [минимальный размер] |
0,000246 |
| Persimmon viroid 2 [типичный размер] |
0,000358 |
| Apple hammerhead viroid-like circular RNA [максимальный
размер] |
0,000434 |
| Вирусы |
| Rice yellow mottle virus satellite [минимальный размер] |
0.00022 |
| Pandoravirus salinus [максимальный размер] |
2,47387 |
| Бактерии |
| Cloacimonetes bacterium JGI 0000039-I11 [минимальный
размер] |
0,104827 |
| Mumia flava [максимальный размер] |
16,3772 |
| Археи |
| Candidatus Parvarchaeum acidophilus [минимальный размер] |
0,100212 |
| uncultured marine crenarchaeote 'Gulf of Maine' [максимальный
размер] |
6,4512 |
По данным, приведенным в таблице, видно, что отношение максимального размера генома
к минимальному больше всего у эукариот (max/min=2456630) и поменьше у вирусов
(max/min=11245). Затем идут бактерии (max/min=156) и археи (max/min=64).
Наименьшее отношение у вироидов (max/min=1,76), общее число опубликованных
геномов которых тоже, однако, не очень велико.