Сборка генома de novo
В данном практикуме я работала с проектом по секвенированию бактерии
Buchnera
aphidicola.
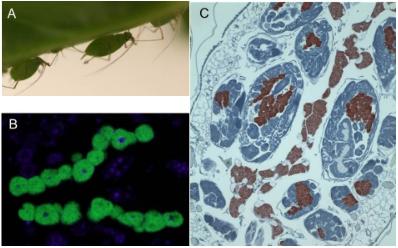
Рис.1.
Бактерия Buchnera в бактериоцитах гороховой тли1.
(A) Внешний вид
гороховой тли; (B) Микрофотография
Buchnera (зеленая) в бактериоцитах; (C)
Локализация
бактериального белка GroEL (красный) в бактериоцитах. |
Бактерия
Buchnera aphidicola относится к gamma-Proteobacteria, которые являются
грам-отрицательными преимущественно патогенными и азотфиксирующими бактериями.
Buchnera
aphidicola — эндосимбионт тлей (насекомых, питающихся растениями). По одной из
версий предки данного вида были свободно живущими бактериями наподобие
E. coli; многие
исследования подтверждают родство данного вида с семейством Enterobacteriaceae.
Для
B. aphidicola характерны практически все особенности, присущие
грам-отрицательным бактериям. Однако у данного вида нет генов, кодирующих ЛПС, в связи с
чем бактерия не патогенна. Геном довольно мал (менее 1 Мб), имеется одна кольцевая хромосома
и несколько плазмид.
Так как
B. aphidicola — симбионт, произошла делеция большого количества
генов (например, генов, кодирующих поверхностные белки).
Бактерия вступила в симбиотические отношения с тлей около 200-150 млн. лет назад. У насекомых
за это время появились специальные клетки — бактериоциты. Тля питается
исключительно растительной пищей, которая богата ЛПС, но почти не содержит соединений азота.
Таким образом, симбиоз выгоден как бактерии, так и насекомому
2.
Мне был дан код доступа SRR4240359. По ссылке http://www.ebi.ac.uk/ena/data/view/SRR4240359
находится архив с короткими ридами, полученными по технологии Illumina.
Подготовка чтений
Сначала был скачан архив с заданными ридами, который затем был распакован в папку
/nfs/srv/databases/ngs/seferbekova командой
gunzip. Затем из файлов в папке с
адаптерами был создан один файл, содержащий все адаптеры, с помощью команды:
cat *.fa > /nfs/srv/databases/ngs/seferbekova/adapters.fasta
Из полученного файла были удалены все адаптеры командой:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359.fastq SRR4240359_out.fastq ILLUMINACLIP:adapters.fasta:2:7:7
После этого были удалены нуклеотиды с низким качеством с концов ридов и риды длиной менее
30 нуклеотидов:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359_out.fastq reads.out TRAILING:20 MINLEN:30
В результате был получен файл
reads.out, содержащий риды после очистки. Для анализа
качества ридов до и после чистки использовалась команда:
fastqc *filename*
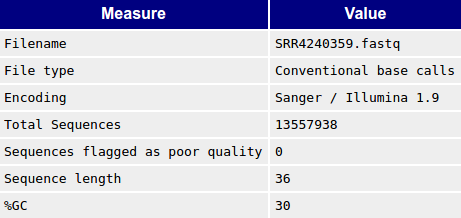
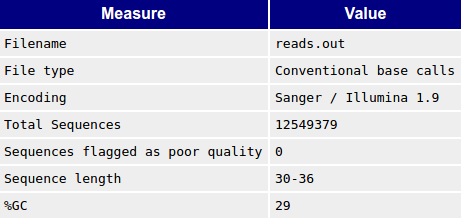
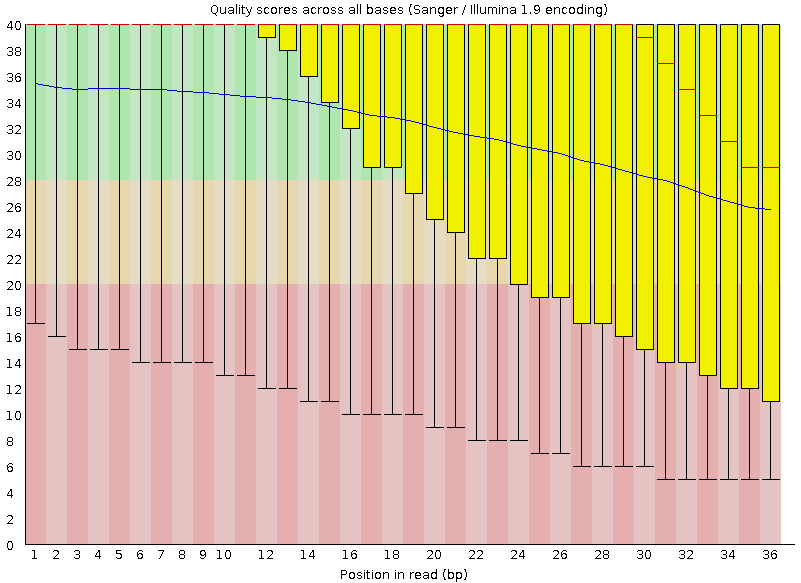
В таблице 1 приведена сравнительная характеристика файлов.
Таблица 1. Сравнительная характеристика ридов до и
после чистки
| |
До чистки (SRR4240359.fastq) |
После чистки (reads.out) |
| Размер файла (кб) |
1408238 |
1286402 |
| html-страницы с характеристиками ридов |
SRR4240359_fastqc.html |
reads.out_fastqc.html |
| Общая статистика |
 |
|
| Качество п.н. в ридах |
 |
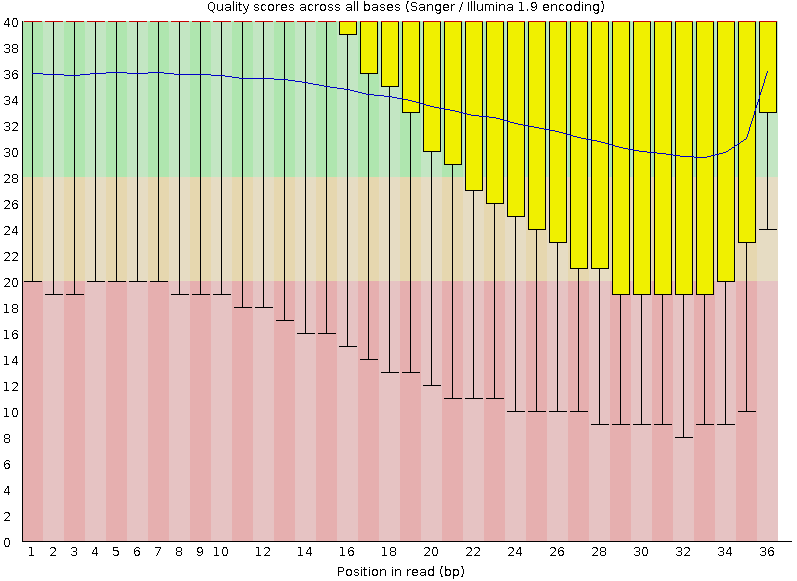 |
Таким образом, после чистки из 13557938 ридов осталось 12549379 (92,56%), т.е. удалено было
1008559 ридов, что, несмотря на страшную цифру, составляет всего 7,44% от исходного числа
ридов. При этом качество отдельных п.н. значительно повысилось. Особенно это заметно
по концевым нуклеотидам. Если посмотреть на характеристики ридов до второго этапа чистки
(
RR4240359_out_fastqc.html), то можно заметить, что,
несмотря на удаление 55902 ридов (0,41%), качество п.н. в ридах осталось прежним. Улучшилось
оно уже только после непосредственного удаления ридов с низким качеством.
Примечательно, что размер файла тоже сильно уменьшился (на 121836 кб).
Использование пакета velvet
Сначала были подготовлены k-меры длины 29, риды короткие и непарные (short):
velveth velveth 29 -fastq -short reads.out
Таким образом, в директории velveth находятся все результаты работы программы. Далее были
собраны контиги на основе полученных k-меров:
velvetg velveth
В итоге было получено 2 файла:
contigs.fa содержит
последовательности контигов и
stats.txt, содержащий статистику.
Таблица 2. Описание трех самых длинных контигов
| ID контига |
Длина |
Покрытие |
Файл с последовательностью |
4 |
91528 |
53.88 |
con4.fa |
| 3 |
83127 |
51.23 |
con3.fa |
| 1 |
78160 |
57.46 |
con1.fa |
Таблица 3. Описание двух контигов с аномальным
покрытием
| ID контига |
Длина |
Покрытие |
| 1704 |
1 |
556838 |
| 1882 |
1 |
2009 |
Всего было найдено 2028 контигов. N50 составляет 47361 п.н. (т.е. ридом с такой длиной и
всеми ридами с большей длиной можно покрыть > половины генома). В таблице 2 описаны 3
самых длинных контига. Среди собранных контигов было 2 контига с
аномальным покрытием (в 5 раз > среднего = 316 ридам). В таблице 3 приведено их описание.
В принципе, такое большое покрытие неудивительно: длина "контигов" всего 1 нуклеотид. Не
знаю, можно ли избежать сборки таких контигов изначально, или можно только потом удалить
все контиги с длиной меньше заданной пороговой.
Контигов с аномально низким покрытием найдено не было.
Таблица 4. Характеристика полученных выравниваний
трех самых длинных контигов с заданной хромосомой
| ID контига |
Total score |
Cover |
Identity |
E-value |
Файл с выравниванием |
| 1 |
24306 |
7% |
75% |
0.0 |
con1_al.txt |
| 3 |
29944 |
9% |
76% |
0.0 |
con3_al.txt |
| 4 |
28895 |
7% |
74% |
0.0 |
con4_al.txt |
Для трех самых длинных контигов был запущен
megablast с хромосомой
Buchnera
aphidicola (GenBank/EMBL AC — CP009253). В таблице 4 приведена характеристика
выравниваний.
Как мне кажется, характеристики у выравниваний неплохие (при этом они схожи для всех трех
контигов). Выравниваний контигов с аномально низкой длиной я не делала, т.к. с учетом их
длины это бессмысленно.
[такое ощущение, что я как-то неправильно использовала velvet; если
на зачете будет найдена какая-то ошибка, переделаю задание]






