Укоренение и бутстрэп
1. Укоренение в среднюю точку
В первом пункте необходимо было дерево, полученное в 4 задании
предыдущего практикума, укоренить в среднюю точку.
Укоренением в среднюю точку называется процесс, при котором на
дереве находится самый длинный путь, соединяющий два листа, и за корень
принимается середина этого пути.
Для выполнения этого задания файл
с деревом в формате Newick был скопирован в файл с названием
intree
и подан на вход программе
retree пакета
PHYLIP (для этого соответствующие программа и файл были помещены в одну
папку). Затем был выбран нужный параметр ("Midpoint root the tree") и построено
укорененное в среднюю точку дерево. Результат представлен на рис.2.
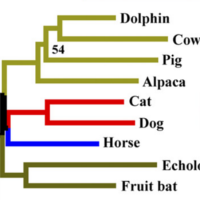
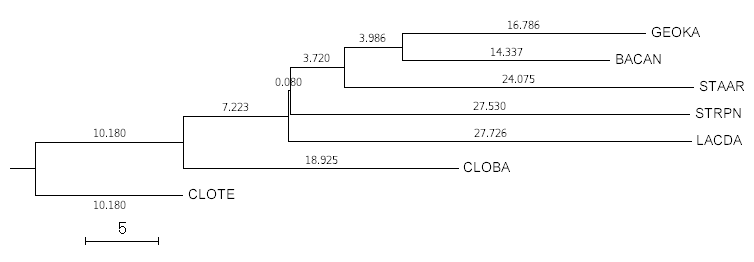 Рис. 1.
Дерево, построенное Jalview (Neighbor-Joining
using % identity) Рис. 1.
Дерево, построенное Jalview (Neighbor-Joining
using % identity) |
 Рис. 2.
Дерево, укорененное в среднюю точку Рис. 2.
Дерево, укорененное в среднюю точку |
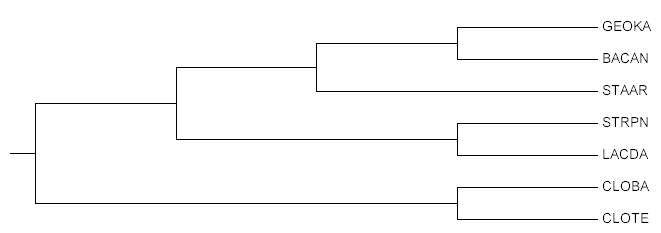 Рис. 3.
Реальное дерево Рис. 3.
Реальное дерево |
Если сравнить все три дерева на рис. 1-3, можно сделать следующие заключения:
- Jalview строит неукорененное дерево (т.к. использует
метод Neighbor-Joining), которое в MEGA укореняется в
случайную(?) (причем еще и тривиальную) ветвь {CLOTE} vs {CLOBA; LACDA,
STRPN, BACAN, GEOKA, STAAR};, очевидно отличающуюся от реальной ветви
{CLOBA, CLOTE} vs {LACDA, STRPN, BACAN, GEOKA, STAAR};, в
которой происходит укоренение. В общем, доверять этому укоренению не стоит;
- если переукоренить дерево в среднюю точку, получится все равно неправильное
из-за неверных исходных данных от Jalview дерево. Оно укоренено в ветвь
{CLOBA, CLOTE, LACDA} vs {STRPN, BACAN, GEOKA, STAAR};, которая вообще отсутствует
в реальном дереве.
Прим. Дерево, укорененное в среднюю точку, MEGA
из-за маленькой длины ветви рисует как
небинарное (в верности можно убедиться, открыв
файл с записью дерева в Newick-формате). Поэтому на рис. 2 представлен
скрин выдачи программы.
Вывод: так как ни один из способов, кроме усовершенствованного метода NJ, реализованного в
MEGA, не дал верное дерево, я бы не стала использовать
Jalview для
построения филогенетических деревьев. Хотя, стоит отметить, что укоренение в среднюю
точку мне не кажется надежным способом; кроме того, как я уже писала, на основе выравнивания
последовательностей одного белка деревья не строятся.
2. Использование внешней группы
В этом пункте необходимо было реконструировать дерево отобранных мною бактерий
(см.
первый практикум) методом максимальной экономии ("Maximum parsimony").
Так как деревья, построенные этим методом, не имеют длину ветвей (а также не
предполагают молекулярных часов), их невозможно укоренить в среднюю точку. Поэтому
было предложено использовать внешнюю группу.
Укоренение с использованием
внешней группы (outgroup) предполагает
укоренение дерева в ветвь, ведущую к аутгруппе. При этом последовательности белков
внешней группы должны иметь более раннего общего предка с исходными, чем общий
предок самих исходных последовательностей. Кроме того, они не должны отличаться
слишком сильно, иначе топология дерева и, следовательно, укоренение могут быть
неверными. В нашем случае в качестве аутгруппы предложили взять пептидил-тРНК-гидролазу
E. coli.
В файл с последовательностями белков фирмикут была добавлена последовательность
белка из кишечной палочки, после чего они были выровнены вместе в
Jalview.
Полученное выравнивание:
task_3_alignment_outgroup.fasta.
Далее с помощью
MEGA из выравнивания было реконструировано дерево (MP).
Затем Subtree>>Root и выбор ветви, ведущей к аутгруппе. После нажатия кнопки
"Show Subtree Separately" было получено дерево, представленное на рис. 4.
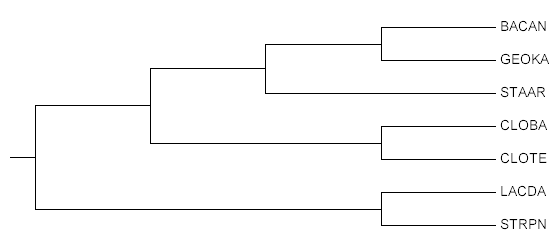 Рис. 4.
Дерево, укорененное в аутгруппу Рис. 4.
Дерево, укорененное в аутгруппу |
Дерево, представленное на рис. 4, укоренено в ветвь {LACDA, STRPN} vs {CLOBA, CLOTE,
BACAN, GEOKA, STAAR}, присутствующую в реальном дереве, которое, тем не менее,
укоренено в другую ветвь. Видимо, что-то пошло не так:
E. coli не является
хорошей аутгруппой в этом случае. Возможно, ее последовательность
пептидил-тРНК-гидролазы не имеет более раннего предка с остальными последовательностями
или же наоборот последовательности слишком далеки друг от друга (что мне кажется
менее вероятным).
3. Бутстрэп
При
бутстрэп-анализе (
bootstrap — «петля на заднике ботинка»)
из исходного выравнивания в результате случайного
удаления половины столбцов и заменой их копиями других (тоже случайно выбранных)
столбцов создается
бутстрэп-реплика. Создается их много (100-500). Далее
для каждой реплики строится дерево, а из них по методу расширенного большинства
(сначала строится дерево из ветвей, встретившихся в большинстве исходных деревьев,
а потом к нему добавляются ветви, не противоречащие уже имеющимся) строится
т.н.
консенсусное дерево. При этом длины ветвей теряются, а ветви
подписываются
бутстрэп-поддержкой — процентом деревьев, в которых
встретилась данная ветвь (нетривиальная).
Для выполнения задания в программе
MEGA было построено дерево с применением
бустрэпа (Test of Phylogeny>>Bootstrap method(100)) методом UPGMA. На рис. 5-6
представлены полученные деревья.
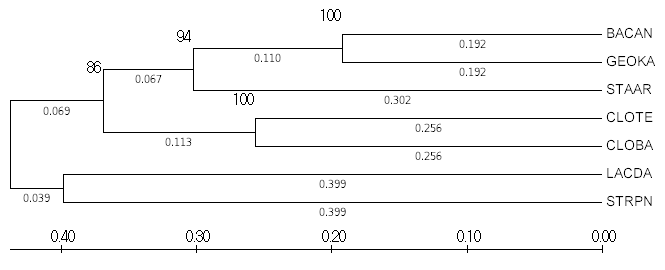 Рис. 5.
Дерево, построенное MEGA методом UGMA (original) Рис. 5.
Дерево, построенное MEGA методом UGMA (original) |
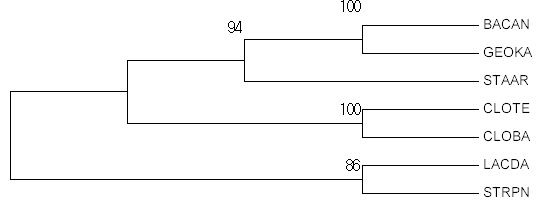 Рис. 6.
Дерево, построенное с помощью бутстрэп-анализа (consensus) Рис. 6.
Дерево, построенное с помощью бутстрэп-анализа (consensus) |
На рисунках видно, что консенсусное и оригинальное деревья имеют одинаковую топологию:
- {BACAN, GEOKA} vs {STAAR, CLOBA, CLOTE, LACDA, STRPN};
- {BACAN, GEOKA, STAAR} vs {CLOBA, CLOTE, LACDA, STRPN};
- {CLOBA, CLOTE} vs {LACDA, STRPN, BACAN, GEOKA, STAAR};
- {LACDA, STRPN} vs {CLOBA, CLOTE, BACAN, GEOKA, STAAR}.
При этом топология соответствует топологии реального дерева. Поддержка ветвей мне
на мой неопытный взгляд кажется довольно хорошей, чем, возможно, и объясняется
верная топология.