Проверка дерева цитохромов b
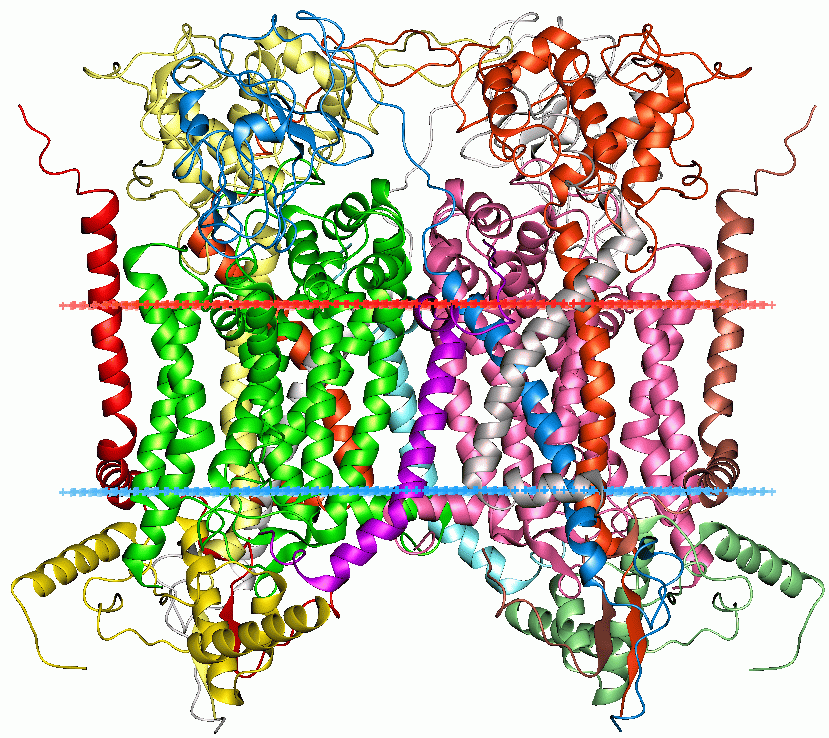 Рис. 1.
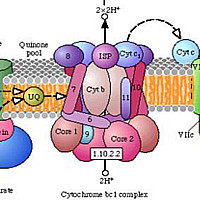
Митохондриальный комплекс цитохромов bc11 Рис. 1.
Митохондриальный комплекс цитохромов bc11 |
Цитохром b — интегральный белок митохондриальных
мембран эукариот, у которых он входит в состав комплекса III, компонента
дыхательной цепи переноса электронов
2. Белок так же
найден у аэробных бактерий и архей. Цитохром b
часто используется для определения филогении организмов из-за
высокой изменчивости его последовательности
3. Лучше
всего подходят для изучения филогенетических связей внутри
семейств и родов.
Мутации в последовательности этого белка у людей вызывают невозможность
выполнения физических упражнений, реже тяжелые мультисистемные
патологии
4.
В этом задании нам было предложено повторить работу, проделанную в
2000 году Michael Schütz и его коллегами
5. На рис.2
представлено дерево цитохромов b, полученное авторами работы с
помощью метода Neighbour-joining (а точнее, "neighbour-joining
method of Saitou and Nei"). Бутстрэп-анализ был проведен с
5000 реплик.
Прим. В презентации сказано: "...авторы работы укоренили дерево
так, чтобы корневая ветвь разделяла археи и эубактерии (что
подтверждало их теорию). Однако такое укоренение ничем обосновано
не было". При этом дерево на рис. 2 неукорененно (тем более метод NJ).
Возможно, я неверно поняла, что имелось в виду. Тем не менее, я сравню
топологию и выберу ветвь, в которую, как по мне, должно быть укоренено
дерево.
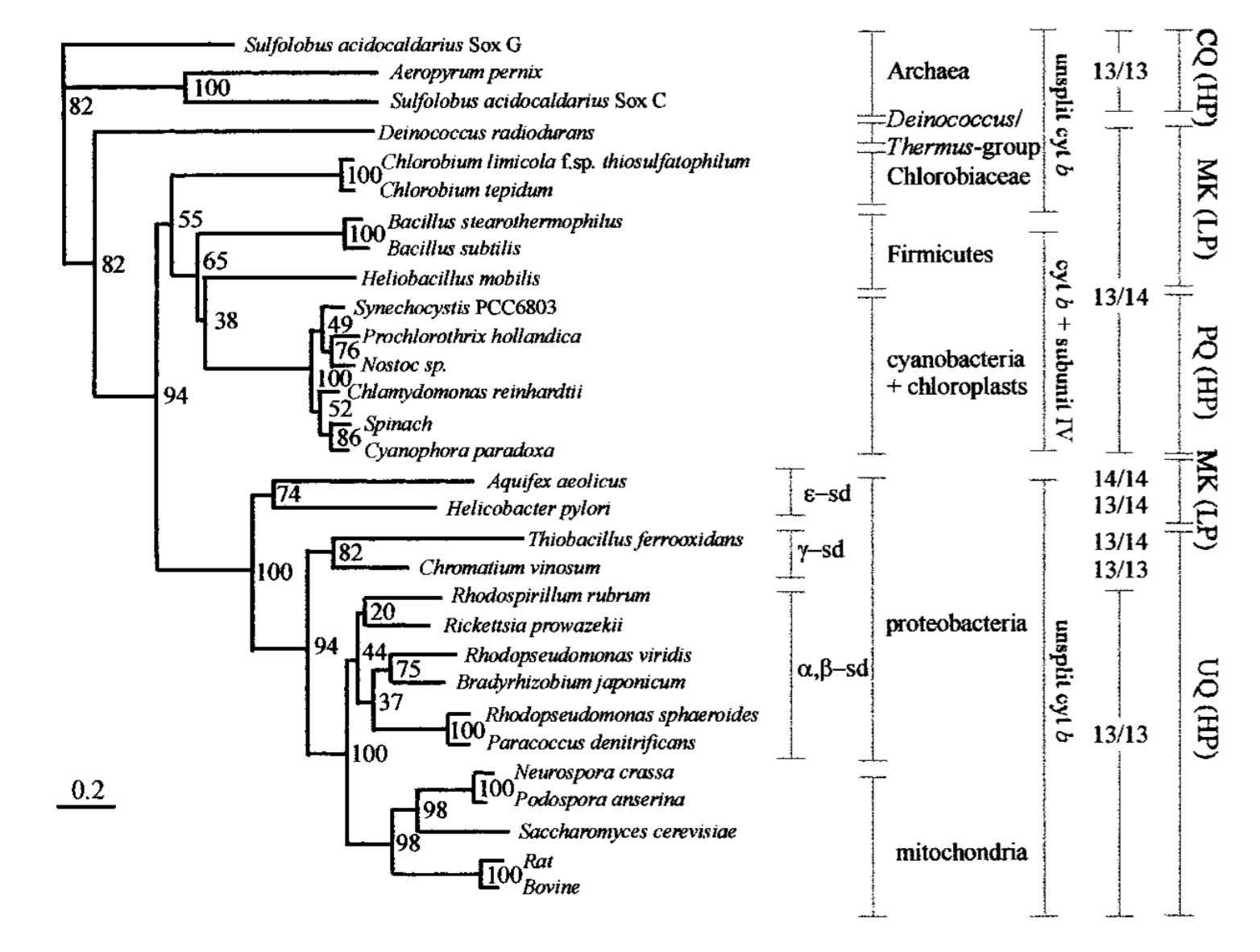 Рис. 2.
Дерево цитохромов b из статьи2 Рис. 2.
Дерево цитохромов b из статьи2 |
С помощью программы
Jalview было построено выравнивание
последовательностей цитохрома b видов, выбранных учеными
для анализа. На рис. 3-6 представлены полученные разными методами
с помощью
MEGA деревья.
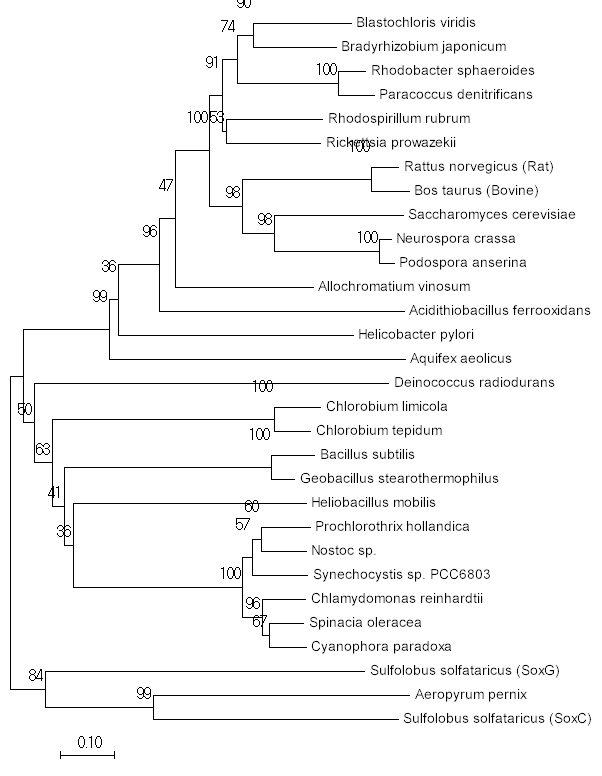 Рис. 3.
Дерево, построенное методом Neighbour-joining Рис. 3.
Дерево, построенное методом Neighbour-joining |
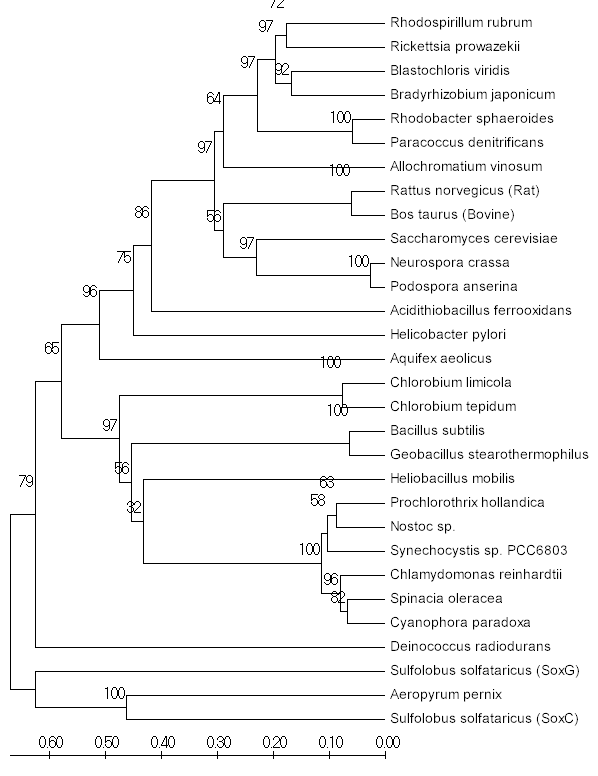 Рис. 4.
Дерево, построенное методом UPGMA Рис. 4.
Дерево, построенное методом UPGMA |
 Рис. 5.
Дерево, построенное методом максимального правдоподобия Рис. 5.
Дерево, построенное методом максимального правдоподобия |
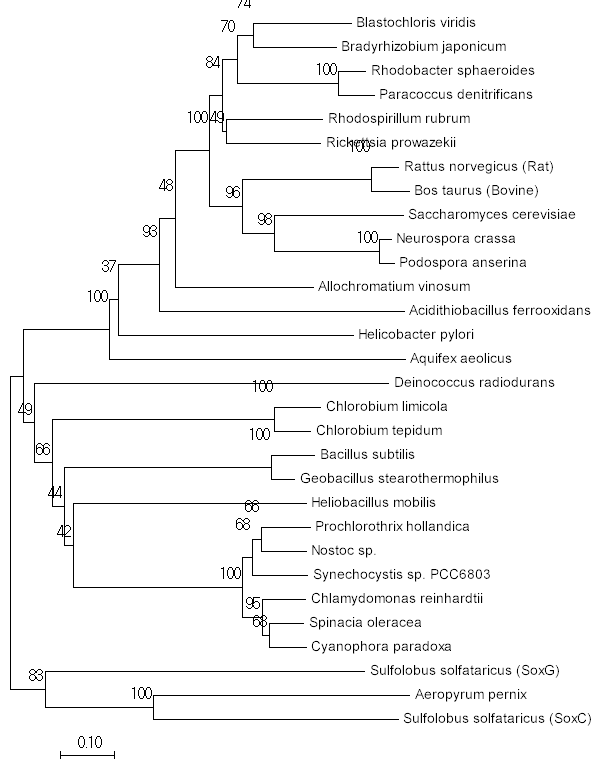 Рис. 6.
Дерево, построенное методом минимальной эволюции Рис. 6.
Дерево, построенное методом минимальной эволюции |
Проанализируем полученные деревья и сравним их с деревом из статьи. Во-первых, хочется
отметить, что дерево в статье является неразрешенным, а все деревья на рис. 3,4,6 —
нет.
Метод Neighbour-joining: Топология дерева на рис. 3 практически не отличается
от топологии дерева из статьи. Небольшая разница заключается в расположении некоторых
тривиальных ветвей, отделяющих
Allochromatium venosum,
Acidithiobacillus
ferrooxidans,
Helicobacter pylori,
Aquifex aeolicus и
Deinococcus
radiodurans.
Метод UPGMA: топология деревьев схожа, но есть некоторые различия:
- нетривиальная ветвь (Rhodobacter sphaeroides, Paracoccus
denitrificants) на рис. 4 является внешней по отношению к кладе
((Rhodospirillum rubrum, Rickettsia prowazekii),(Blastochloris
viridis, Bradyrhyzobium japonicum)), в то время как на дереве в
статье ветвь (Rhodospirillum rubrum, Rickettsia prowazekii) является
внешней для клады ((Rhodobacter sphaeroides, Paracoccus
denitrificants), (Blastochloris viridis, Bradyrhyzobium japonicum));
- опять ветви Allochromatium vinosum, Acidithiobacillus ferrooxidans,
Helicobacter pylori, Aquifex aeolicus расположены по-другому.
Метод максимального правдоподобия: все еще не совсем то дерево, которое
представлено в статье. Отличия:
- как и в предыдущем случае, нетривиальная ветвь (Rhodobacter sphaeroides,
Paracoccus denitrificants) на рис. 5 является внешней по отношению к кладе
((Rhodospirillum rubrum, Rickettsia prowazekii),(Blastochloris viridis,
Bradyrhyzobium japonicum)), в то время как на дереве в статье ветвь (
Rhodospirillum rubrum, Rickettsia prowazekii) является внешней для клады ((
Rhodobacter sphaeroides, Paracoccus denitrificants), (Blastochloris
viridis, Bradyrhyzobium japonicum));
- ветви Helicobacter pylori и Aquifex aeolicus, Deinococcus
radiodurans расположены по-другому;
- ветвь Heliobacillus mobilis на рис. 5 вынесена в отдельную кладу (
Heliobacillus mobilis,(Bacillus subtilis, Geobacillus stearothermophilus)),
что кажется вполне логичным;
- присутствует неразрешенное поддерево.
Метод минимальной эволюции: в этом случае отличается расположение ветвей
Allochromatium vinosum, Acidithiobacillus ferrooxidans, Helicobacter pylori,
Aquifex aeolicus и
Deinococcus radiodurans.
Таким образом, сильнее всего от дерева в статье отличается дерево, построенное методом
максимального правдоподобия. Деревья, построенные методами минимальной эволюции
и Neighbour-joining не отличаются друг от друга по топологии.
Выводы:
- так как на всех полученных деревьях нетривиальная ветвь, отделяющая архей,
является внешней к кладе бактерий, мне кажется, что укоренение в эту ветвь вполне обосновано.
Вероятно, последовательности белков архей в этом случае можно использовать в качестве
аутгруппы для бактерий в дереве;
- разное расположение ветвей Allochromatium vinosum, Acidithiobacillus ferrooxidans,
Helicobacter pylori, Aquifex aeolicus можно объяснить невысокой
поддержкой нетривиальных ветвей. Согласно таксономии NCBI упомянутые бактерии не являются
членами каких-либо общих таксонов (ниже протеобактерий или бактерий), так что
если опираться на NCBI, я бы не стала объединять эти бактерии в отдельные клады
(как на деревьях на рис. 2, 4). Хотя отсутствие общих низких таксонов для этих
организмов может быть следствием неизученности их эволюционных взаимоотношений;
- ветвь бактерии Deinococcus radiodurans я бы все-таки сделала внешней
для остальных бактерий и расположила "поближе" к археям, как на рис.2,4, т.к. эта
грамположительная бактерия является экстремофилом, известным благодаря высокой
устойчивости к радиции6. Хотя эти характеристики могут быть следствием
не близкородственности этой бактерии и архей, а результатом конвергентной эволюции
в сходных условиях обитания;
- исходя из всего вышесказанного, самым правдоподобным мне кажется дерево,
построенное по методу UPGMA.
Ссылки:
[1] Cytochrome b // Wikipedia: the free encyclopedia
[URL].
[2] Blankenship, Robert (2009). Molecular Mechanisms of Photosynthesis.
Blackwell Publishing. pp. 124–132.
[3] Castresana, J. (2001). Cytochrome b Phylogeny and the Taxonomy of
Great Apes and Mammals. Molecular Biology and Evolution. 18 (4): 465–471.
[4] Blakely E.L., Mitchell A.L., Fisher N., Meunier B., Nijtmans L.G., Schaefer A.M.,
Jackson M.J., Turnbull D.M., Taylor R.W. (2005). A mitochondrial cytochrome
b mutation causing severe respiratory chain enzyme deficiency in humans and yeast.
FEBS J. 272 (14): 3583–92.
[5] Schütz M., Brugna M., Lebrun E., Baymann F., Huber R., Stetter K., Hauska G., Toci R.,
Lemesle-Meunier D., Tron P., Schmidt C., Nitschke W. (2000). Early evolution of
cytochrome bc complexes. Journal of Molecular Biology. 300 (4): 663-75.
[6] Rainey F. A., Ray K., Ferreira M., Gatz B. Z., Nobre M. F., Bagaley D., Rash B. A.,
Park M. J., Earl A. M., Shank N. C., Small A. M., Henk M. C., Battista J. R., Kämpfer P.,
da Costa M. S. (2005). Extensive diversity of ionizing-radiation-resistant bacteria
recovered from Sonoran Desert soil and description of nine new species of the genus
Deinococcus obtained from a single soil sample. Applied and environmental
microbiology. 71(9: 5225—5235.