Дерево по нуклеотидным последовательностям. Паралоги
1. Построение дерева по нуклеотидным последовательностям
Последовательности 16S рибосомальной РНК каждой из бактерий из
практикума
были получены из файлов
.frn (было выбрано по одной из копий гена) из
базы полных
геномов NCBI. Все они были записаны в общий файл
rnas.fasta
с соответствующими мнемониками для последовательности каждой бактерии. Затем
полученный файл с выравниваниями был открыт в
Jalview и было построено
выравнивание алгоритмом
Muscle с умолчательными настройками. На рис. 1 представлено
полученое выраванивание. Выравнивание в формате
.fasta:
alignment.fa.
Рис. 1. Выравнивание
последовательностей 16S-рРНК в Jalview (раскраска
Nucleotide)
Полученное выравнивание было импортировано в
MEGA и методом Neighbour-joinong
было построено дерево, представленное на рис. 2.
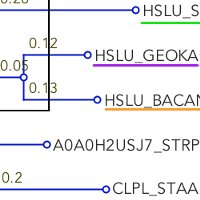
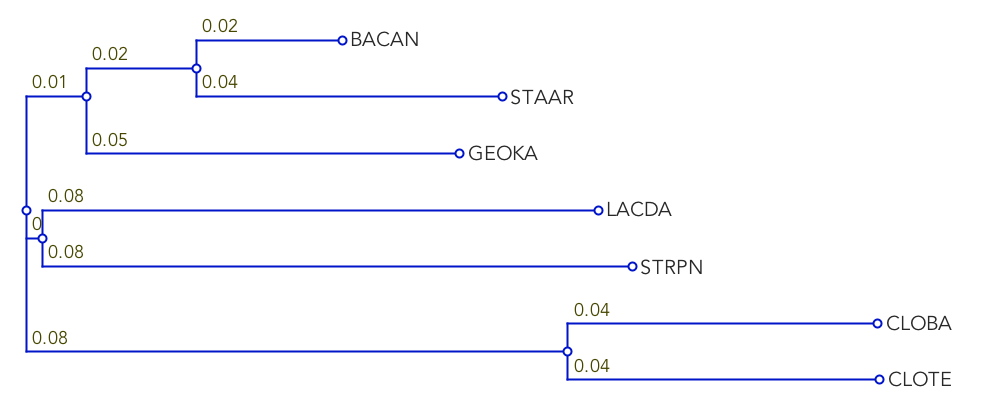 Рис. 2. Построенное
дерево (метод NJ) Рис. 2. Построенное
дерево (метод NJ) |
Полученное дерево отличается от истинного взаимным расположением видов с
мнемониками STAAR и GEOKA. Интересно отметить, что в случае построения
дерева тем же методом, но по последовательностям белка, а не 16S-рРНК,
получилось правильное дерево. Мне стало интересно, является ли это
следствием того, что деревья, построенные по выравниваниям белковых
последовательностей точнее отражают эволюционные взаимоотношения, чем
деревья, построенные по выравниваниям нуклеотидных последовательностей и,
в частности, 16S-рРНК. Поэтому я решила воспользоваться силами Google.
Ген 16S-рРНК присутствует во всех клетках; у бактерий в геноме может быть до
10 копий этого гена. Они часто используются для построения деревьев, т.к.:
- присутствуют во всех клетках;
- функция продукта одинакова во всех клетках;
- содержат как быстро, так и медленно эволюционирующие участки.
Первые подходят для определения эволюционных взаимоотношений близких видов,
а вторые — дальних;
- горизонтальный перенос генов рРНК если и происходит, то очень редко1.
Таким образом, не совсем понятно, почему в этом случае дерево отличается от
правильного. Более того, два других метода (минимальной эволюции и максимального
правдоподобия) построили деревья с такой же топологией, как NJ (деревья не
выкладываю). Такие результаты могут быть связаны с тем, что выравнивание
нуклеотидных последовательностей менее достоверно, чем выравнивание белковых
последовательностей, так как i) не учитывается, является ли замена нуклеотида
синонимичной и ii) в выравниваниях аминокислотных последовательностей есть
учет замены аминокислоты на аминокислоту со сходными свойствами и замены
на аминокислоту с отличающимися свойствами, что делает выравнивание более
точным; в выравниваниях нуклеотидных последовательностей такого, очевидно,
нет.
Вывод: в общем, несмотря на приведенную цитату о возможности
использования генов 16S-рРНК для построения деревьев, в моем случае этот
способ сработал неидеально. Хотя, с другой стороны, строить дерево
по последовательностям одной рРНК/одного белка — не самая лучшая идея.
2. Построение и анализ дерева, содержащего паралоги
Паралоги — гомологичные белки из одного организма.
Ортологи
— гомологичные белки из разных организмов или гомологичные белки, для
которых разделение их общего предка на линии, ведущей к ним, произошло в
результате видообразования. В данном задании предлагалось построить дерево
гомологичных белку
CLPX_BACSU белков моих бактерий. Затем, считая, что дерево построено
верно, определить ортологов и паралогов, а также указать некоторые
эволюционные события.
Сначала был создан файл с протеомами моих бактерий:
proteomes.fasta, который был подан на вход
blastp. Таким образом
были найдены предполагаемые
гомологи
(порог e-value=0.001). В таблице 1 перечислены найденные гомологи.
Табл. 1. Найденные гомологи
| Мнемоника |
Продукт |
Score |
E-value |
| CLPX_BACAN |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
733 |
0.0 |
| CLPX_GEOKA |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
712 |
0.0 |
| CLPX_STAAR |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
607 |
0.0 |
| CLPX_CLOTE |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
596 |
0.0 |
| CLPX_CLOBA |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
593 |
0.0 |
| CLPX_STRPN |
ATP-dependent Clp protease ATP-binding subunit
ClpX |
541 |
0.0 |
| HSLU_GEOKA |
ATP-dependent protease ATPase subunit HslU |
101 |
6e-24 |
| HSLU_BACAN |
ATP-dependent protease ATPase subunit HslU |
97.8 |
1e-22 |
| HSLU_STAAR |
ATP-dependent protease ATPase subunit HslU |
97.8 |
2e-22 |
| Q5L436_GEOKA |
ATP-dependent Clp protease ATPase subunit |
65.9 |
6e-12 |
| Q81VV9_BACAN |
ATP-dependent Clp protease ATP-binding
subunit ClpC |
60.5 |
3e-10 |
| A0A0H2USJ7_STRPN |
ATP-dependent Clp protease, ATP-binding
subunit |
59.3 |
7e-10 |
| Q890L5_CLOTE |
Negative regulator of genetic competence
mecB/clpC |
57.4 |
3e-09 |
| CLPL_STAAR |
ATP-dependent Clp protease ATP-binding subunit
ClpL |
55.1 |
2e-08 |
| CLPC_STAAR |
ATP-dependent Clp protease ATP-binding subunit
ClpC |
53.9 |
4e-08 |
| Q899V4_CLOTE |
Negative regulator of genetic competence
mecB |
52.4 |
1e-07 |
| A0A0H2UNL3_STRPN |
Putative ATP-dependent Clp protease,
ATP-binding subunit |
47.0 |
5e-06 |
| Q899H3_CLOTE |
ATP-dependent zinc metalloprotease FtsH |
45.1 |
2e-05 |
| FTSH_STRPN |
ATP-dependent zinc metalloprotease FtsH |
45.1 |
2e-05 |
| RUVB_CLOBA |
Holliday junction ATP-dependent DNA helicase
RuvB |
42.7 |
1e-04 |
| Q891B9_CLOTE |
ATP-dependent zinc metalloprotease FtsH |
42.7 |
1e-04 |
| Y1421_STAAR |
Uncharacterized protein SAR1421 |
42.0 |
1e-04 |
| Q5L3T1_GEOKA |
ATP-dependent zinc metalloprotease FtsH |
42.0 |
2e-04 |
| Q81VX5_BACAN |
ATP-dependent zinc metalloprotease FtsH |
41.6 |
2e-04 |
| Q898D1_CLOTE |
ATP-dependent zinc metallopeptidase |
40.8 |
4e-04 |
| CLPB_CLOTE |
Chaperone protein ClpB |
40.8 |
5e-04 |
Jalview почему-то не отбросил нижние семь находок (выделены курсивом),
хотя их e-value выше порогового (0.001). Поэтому при построении выравнивания
они не учитывались. Последовательности 19 найденных белков были загружены в
Jalview и выровнены (
Muscle with defaults). На рис.3 представлено
полученное выравнивание (
.fasta:
alignment2.fasta). Выравнивание всех 26 находок можно посмотреть
здесь.
По приведенному выше выравниванию в
MEGA было построено дерево (метод
Neighbour-joining). Оно изображено на рис. 4.
Рис. 3. Выравнивание
последовательностей исходного белка и гомологов
в Jalview (раскраска Clustalx)
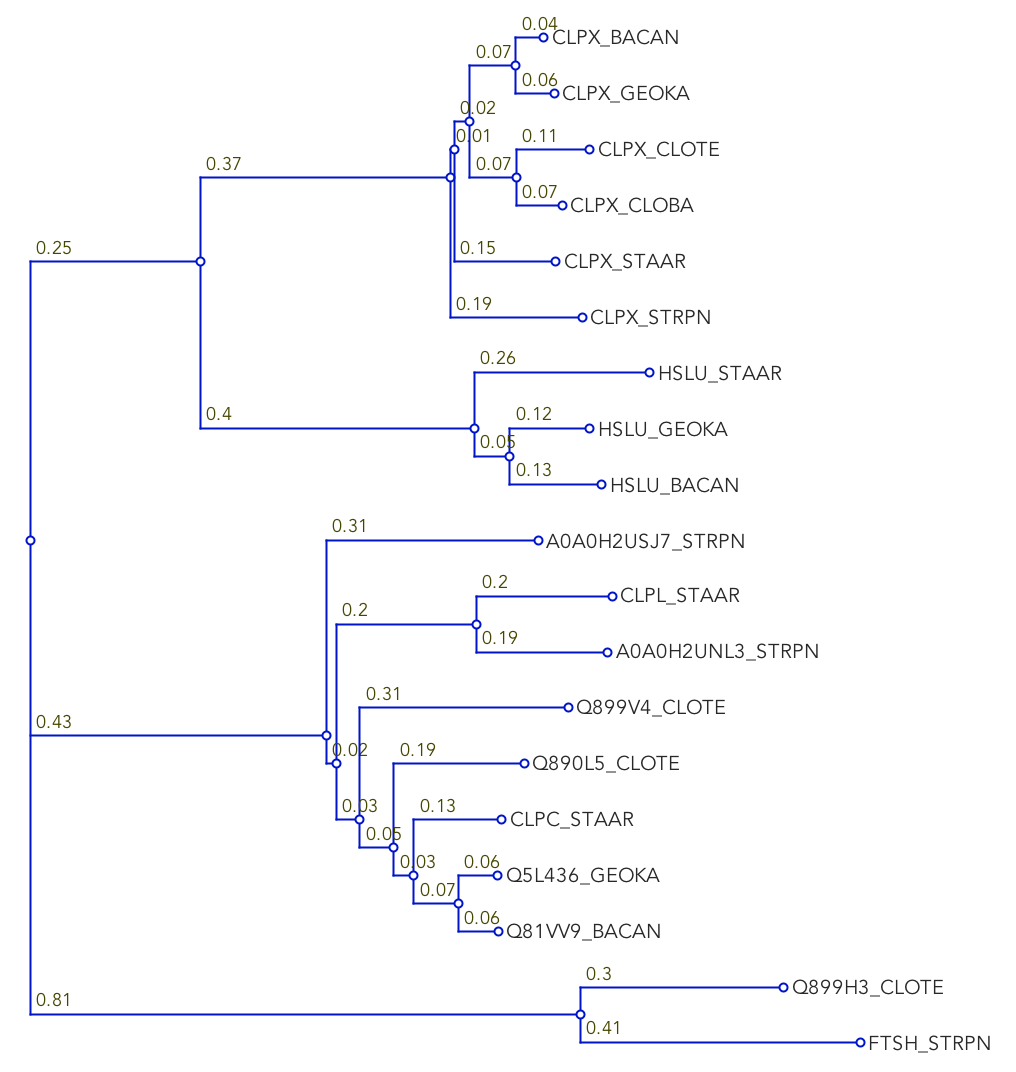 Рис. 4.
Построенное дерево (метод NJ) Рис. 4.
Построенное дерево (метод NJ) |
Clp — эндопептидаза, катализирующая гидролиз белков в
присутствии АТФ
2. Состоит из нескольких субъединиц: ClpP/ClpQ (обладают протеазной
активностью) и ClpX/ClpC/ClpE/и др. (связывают АТФ). Этот белковый комплекс найден
в бактериях, а также в митохондриях и хлоропластах эукариот
3.
HlsVU — АТФ-зависимая протеаза, присутствующая у многих бактерий.
Состоит из субъединиц: HslU (АТФ-зависимый шаперон и активатор второй субъединицы) и HslV
(обладает протеазной активностью)
4.
FtsH — АТФ-зависимая металлопептидаза (кофактор — ион цинка)
5.
Таким образом, некоторые группы ортологов, представленные на дереве:
- CLPX_BACAN, CLPX_GEOKA, CLPX_STAAR, CLPX_CLOTE, CLPX_CLOBA, CLPX_STRPN:
одна и та же субъединица ClpX;
- HSLU_GEOKA, HSLU_BACAN, HSLU_STAAR: одна и та же субъединица HslU;
- CLPC_STAAR, Q81VV9_BACAN: одна и та же субъединица ClpC;
- Q899H3_CLOTE, FTSH_STRPN: одна и та же субъединица FtsH
Все они кодируют определенную субъединицу, выполняющую одну и ту же роль в разных
бактериях.
Принимая во внимание тот факт, что HslU и СlpX имеют схожее строение и функцию
6,
некоторые паралоги, представленные на дереве:
- CLPX_BACAN и HSLU_BACAN;
- HSLU_GEOKA и CLPX_GEOKA;
- CLPX_STAAR и HSLU_STAAR.
На рис. 5 отмечены все упомянутые группы гомологов, а также дупликации генов и
разделение путей эволюции белков в результате видообразования.
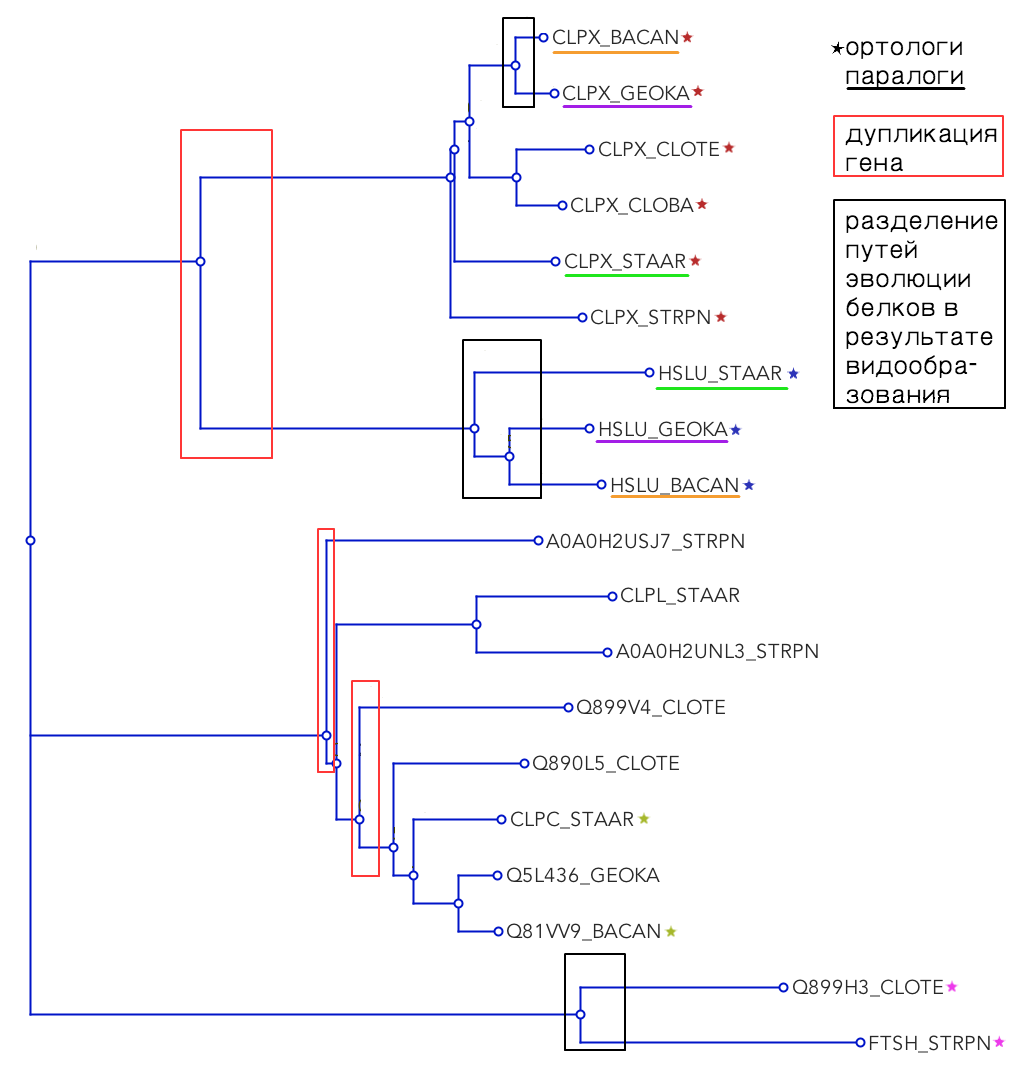 Рис. 5.
То же самое дерево + группы гомологов + эволюционные события Рис. 5.
То же самое дерево + группы гомологов + эволюционные события
|