Работа с KEGG ORTHOLOGY
1. Выбор пары ортологических рядов для дальнейшей работы
Таблица 1. Выбранные ряды
| Идентификатор ряда |
K13920 |
K13919 |
| Число генов |
1211 |
1353 |
Число белковых последовательностей
(из них в UniProt) |
129 (128) |
106 (105) |
В базе данных
KEGG был
выбран метаболический путь
Propanoate metabolism (метаболизм пропаноата). В этом метаболическом
пути была выбрана реакция (EC:
4.2.1.28):
Propane-1,2-diol => Propanal
Данную реакцию катализирует три ряда ортологичных белков:
K01699,
K13919,
K13920.
Из них было выбрано два ортологических ряда, краткая информация о которых
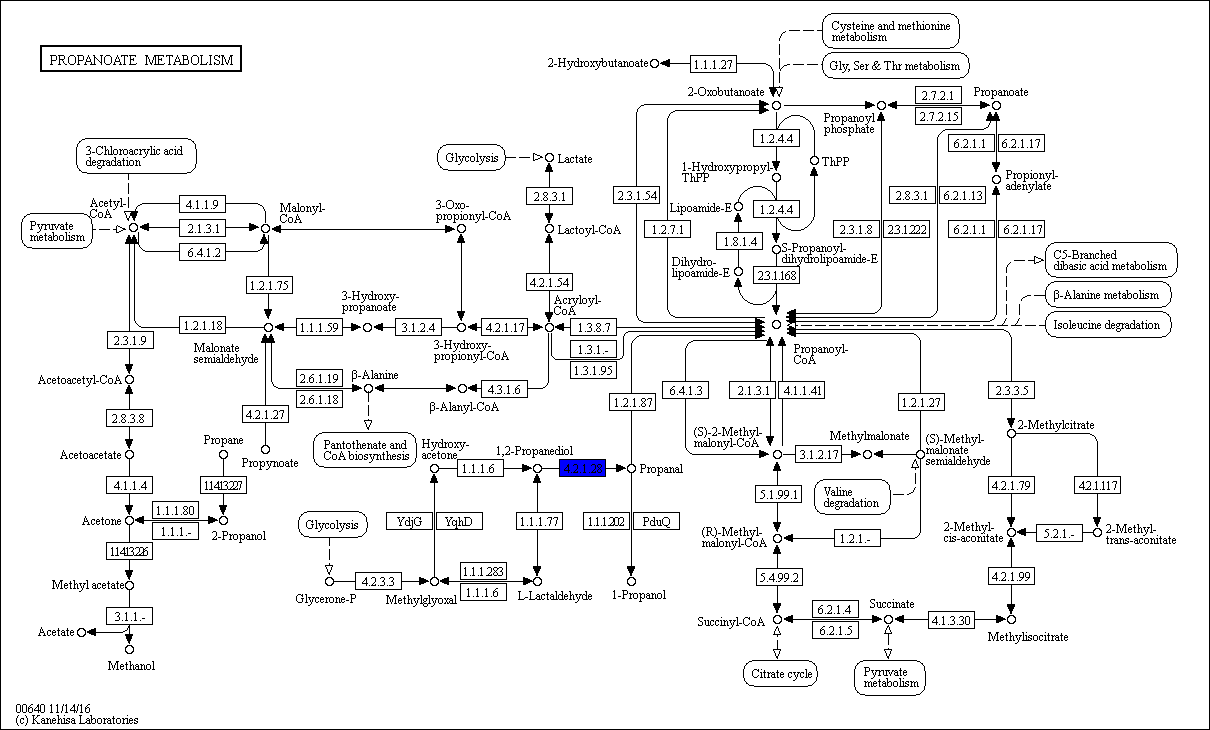
приведена в таблице 1. На рис. 1 представлено изображение метаболического
пути с окрашенной выбранной реакцией.
Рис. 1. Метаболизм пропаноата. Выбранная
реакция окрашена синим.
2, 3. Получение совместного множественного выравнивания.
Проверка гомологичности белков в выравнивании
Для получения белковых последовательностей для каждого из двух
ортологичных рядов я щелкнула на 'UniProt' и получила таблицы
с идентификаторами соответствующих белков. Затем полученные
идентификаторы были поданы сервису
Retrive/ID mapping UniProt и
таким образом были получены последовательности всех белков для
обоих рядов. Далее в файлах с последовательностями с помощью
скрипта к каждому идентификатору белка был приписан идентификатор
соответствующего ряда. Так, были получены итоговые файлы с
последовательностями белков:
K13920.fasta
и
K13919.fasta. Все последовательности
были объединены в один файл и выровнены в
MEGA (
Muscle).
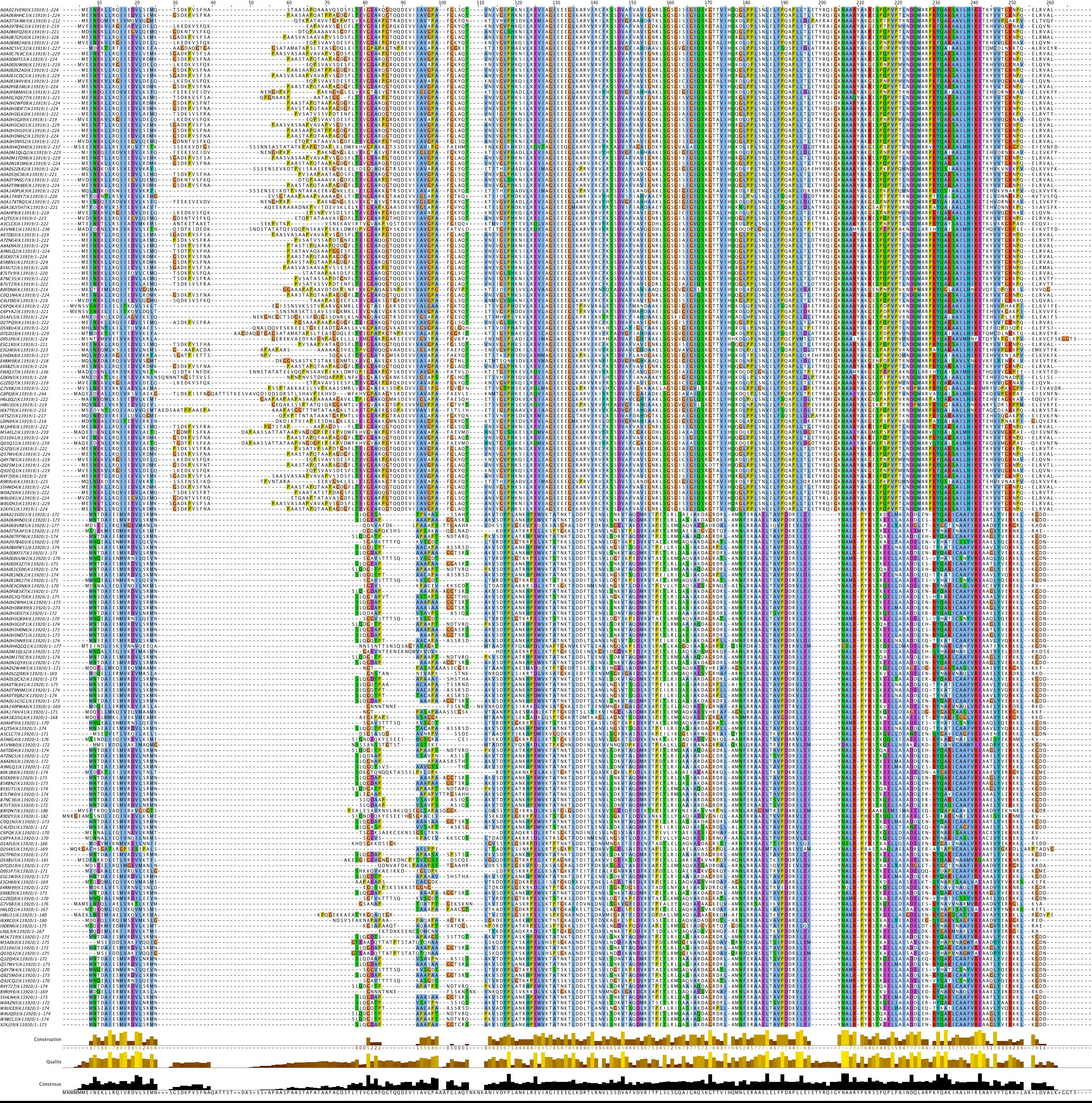
На рис. 2 представлено построенное выравнивание до "чистки", а на рис. 3
— после.
Соответствующие выравнивания в
.fasta и проект
Jalview:
Проверка выравнивания. Так как мое выравнивание содержало аж 233
последовательности, из него без зазрения совести были удалены 48
последовательностей, в которых в консервативных позициях выравнивания
стояли гэпы (
список). При этом 35
последовательностей принадлежали ряду
K13920. В целом,
последовательности этого ортологического ряда выровнялись хуже
последовательностей ряда
K13919. Таким образом, было получено
выравнивание, изображенное на рис. 3, содержащее уже 185
последовательностей.
Рис. 2. Выравнивание до чистки
(раскраска Clustalx).
Рис. 3. Выравнивание после
чистки (раскраска Clustalx).
Гомологичность белков в выравнивании. Мне сложно судить, гомологичны
ли белки в полученном множественном выравнивании и можно ли вообще здесь
говорить о множественном выравнивании как таковом, так как у меня мало
опыта построения выравниваний и оценки фиогении. Тем не менее, очевидна
гомология белков, принадлежащих одному ряду, а вот гомологии белков из разных
рядов я не вижу, так как вертикальных блоков с большим числом консервативных
позиций для обоих рядов нет. Если внутри рядов блоки наблюдаются на протяжении
всей длины последовательностей,то для обоих рядов сразу консервативные позиции
наблюдаются только в конце последовательностей, и то о каких-либо блоках говорить
нельзя. В общем, я бы сказала, что множественное выравнивание отсутствует, а белки
из разных рядов не являются гомологами в моем случае.
4. Построение филогенетического дерева
Так как в предыдущем пункте был сделан вывод, что белки из разных рядов
негомологичны и множественного выравнивания нет, строить дерево не
имеет смысла.
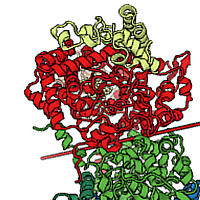
Результат, в принципе, неудивительный. Белки из исследуемых
рядов являются двумя разными субъединицами pduD (
K13919)
и pduE (
K13920) дегидратазы пропандиола. Очевидно, субъединицы
сложного белка могут быть как идентичными, так и гомологичными или
же вовсе различными, в зависимости от выполняемых ими функций.
К сожалению, толковых статей по этому белку и субъединицам я не
нашла, так что вывод основываю только на приведенных здесь данных.