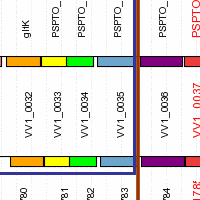
Геномное окружение. База данных GO
1. Получение информации о КОГе
Для работы использовался белок, выданный в I семестре. Это карбогидрат-киназа
из генома археи
Thermococcus kodakarensis KOD1 Краткая
информация о нем доступна по
ссылке. Последовательность этого белка в
fasta-формате
была вставлена в поле поиска сервиса
CDD. Затем для результатов был выбран режим
View>Full Results.
Всего было найдено два КОГа, информация о которых приведена в
таблице 1. Общая длина белка — 273 а.о.
Таблица 1. Краткая информация о найденных КОГах
| COG ID |
Название |
e-value |
Остатки белка, соответствующие КОГу |
Функциональная категория |
| COG0524 |
Sugar or nucleoside kinase, ribokinase family
(Карбогидрат- или нуклеозид-киназа, семейство
рибокиназ) |
1.76e-05 |
2-247 |
Carbohydrate transport and metabolism
(транспорт и метаболизм сахаров) |
| COG1105 |
Fructose-1-phosphate kinase or kinase (PfkB)
(фруктоза-1-фосфат-киназа или киназа) |
3.77e-05 |
181-273 |
Carbohydrate transport and metabolism
(транспорт и метаболизм сахаров) |
2. Визуализация геномного окружения
Для КОГа
COG0524 с помощью сервиса
STRING
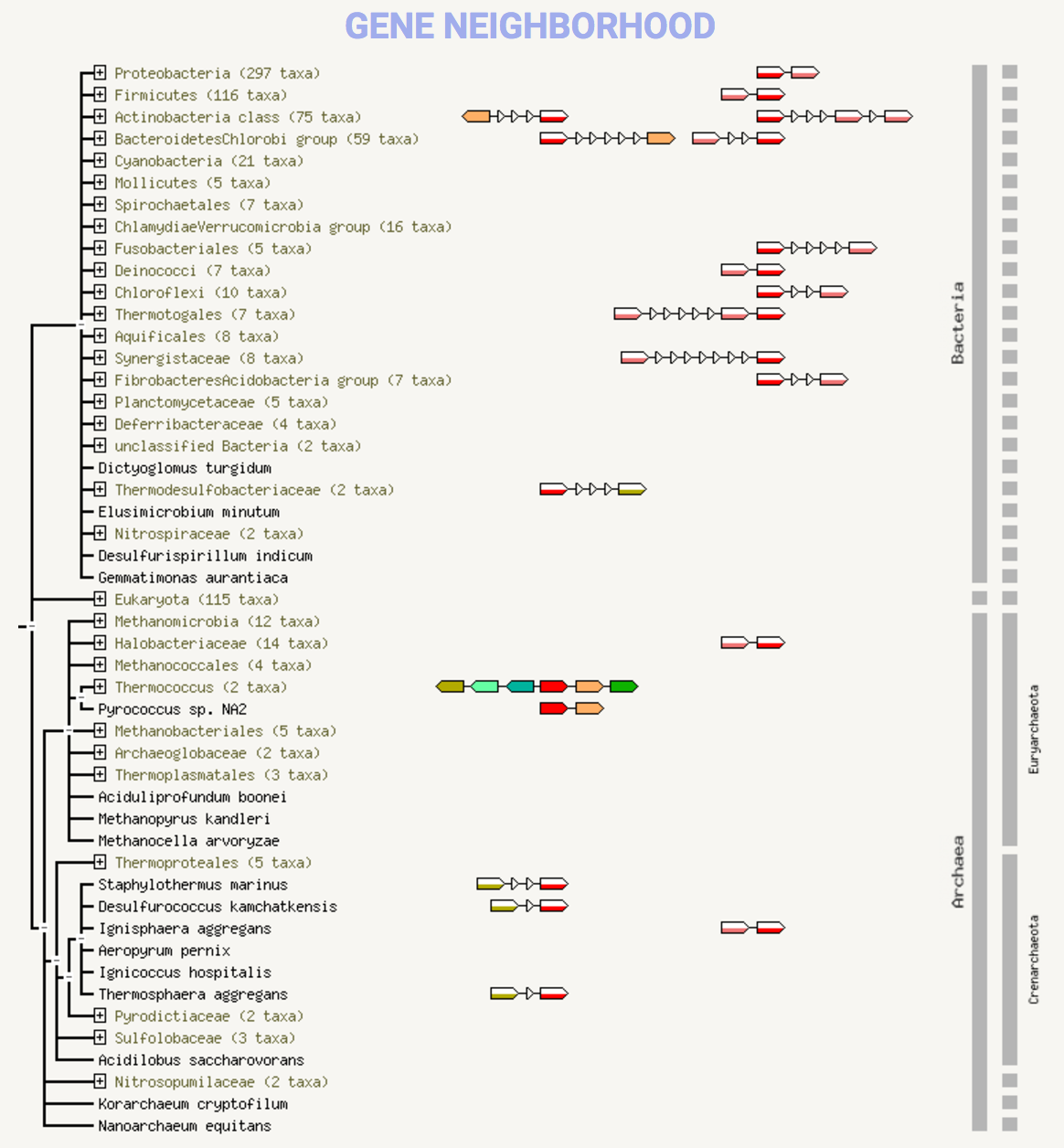
было получено геномное окружение, представленное на рис. 1. На рис. 2
представлены все найденные сервисом белки (для поиска были выбраны параметры по
умолчанию).
 Рис. 1. Геномное
окружение КОГа COG0524 Рис. 1. Геномное
окружение КОГа COG0524 |
 Рис. 2. Найденные
КОГи и их взаимодействия Рис. 2. Найденные
КОГи и их взаимодействия |
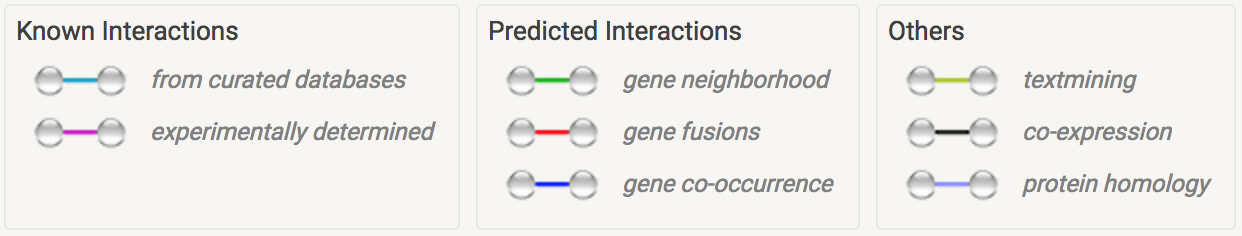 Рис. 3. Расшифровка
взаимодействий Рис. 3. Расшифровка
взаимодействий |
На рис. 1 представлен граф, в котором узлы (изоформы белка, транскрибирующиеся с
одного и того же гена) соединены различными отношениями (см. рис. 3). Красный узел
представляет собой наш белок. Как видно по графу, для трех находок предсказана 3D
структура для (крупные узлы со структурой внутри). В соответствии
с цветами можно выделить следующие типы отношений:
- для известных отношений голубые ребра обозначают отношения,
представленные в курируемых базах данных, а малиновые —
это экспериментально определенные отношения;
- для предсказанных отношений зеленые ребра обозначают
соседство генов в геноме, красные — слияние генов, а синие
— их совместную встречаемость;
- для остальных типов салатовым окрашены ребра, соединяющие
совместно упоминаемые в PubMed белки, черным —
ко-экспрессируемые белки, а фиолетовым — гомологичные белки.
В нашем случае большинство узлов соединены зелеными или синими ребрами. При этом ни
экспериментально доказаных, ни аннотированных в базах данных взаимосвязей нет. Более
того, часть находок являются плохо изученными белками, а остальные явно выполняют
другие функции (см. рис. 2). На рис. 4 представлено соседство генов. Видно, что
никаких определенных паттернов нет. На рис. 5 показана совместная встречаемость.
Видно, что здесь тоже нет какой-то закономерности. Таким
образом, я бы сделала вывод, что в данном случае консервативного окружения
нет.
Рис. 4. Соседство генов
Рис. 5. Совместная встречаемость генов
Отнесение белка с UniProt ID UCRI_RAT из крысы к терминам GO
Так как для моего белка БД
GO не нашлось белка, сходного с моим настолько,
чтобы можно было переносить термины GO с одного на другой, мне был выдан другой
белок с UniProt ID
UCRI_RAT из
Rattus norvegicus (Серая крыса).
Это субьединица Риске из комплекса цитохромов bc1 (или комплекса III), располагающегося
во внутренней мембране митохондрий
1.
С помощью инструмента
AmiGO
поиском BLAST в БД
GO я попыталась найти белок, наиболее похожий на упомянутый
выше. Оказалось, что сам этот белок есть в БД
GO, так что для дальнейшей
работы был взят сам белок Риске из крысы. В таблице 1 описаны все GO, отнесенные
к исследуемому белку (
Term Associations).
Таблица 2. Термины GO, отнесенные к белку с идентификатором
Uniprot P20788 (UCRI_RAT)
| Аспект |
Идентификатор GO |
Название термина |
Перевод названия термина |
Код типа достоверности |
| биологический процесс |
GO:1902600 |
hydrogen ion transmembrane transport |
трансмембранный транспорт протонов |
IEA |
| биологический процесс |
GO:0055114 |
oxidation-reduction process |
окислительно-восстановительный процесс |
IEA |
| биологический процесс |
GO:0046677 |
response to antibiotic |
реакция на антибиотик |
IEP |
| биологический процесс |
GO:0042493 |
response to drug |
реакция на наркотик |
IEP |
| биологический процесс |
GO:0009725 |
response to hormone |
реакция на гормон |
IEP |
| клеточный компонент |
GO:0005743 |
mitochondrial inner membrane |
внутренняя митохондриальная мембрана |
ISO |
| клеточный компонент |
GO:0031966 |
mitochondrial membrane |
митохондриальная мембрана |
IDA |
| клеточный компонент |
GO:0005750 |
mitochondrial respiratory chain complex III |
комплекс III митохондриальной дыхательной цепи |
IDA |
| клеточный компонент |
GO:0005739 |
mitochondrion |
митохондрия |
ISO |
| клеточный компонент |
GO:0043209 |
myelin sheath |
миелиновая оболочка |
IEA |
| клеточный компонент |
GO:0045275 |
respiratory chain complex III |
комплекс III дыхательной цепи |
TAS |
| молекулярная функция |
GO:0051537 |
2 iron, 2 sulfur cluster binding |
связывание кластера [2Fe-2S] |
IEA |
| молекулярная функция |
GO:0046872 |
metal ion binding |
связывание ионов металла |
IEA |
| молекулярная функция |
GO:0032403 |
protein complex binding |
связывание белкового комплекса |
IDA |
| молекулярная функция |
GO:0008121 |
ubiquinol-cytochrome-c reductase |
убихинол-цитохром-с редуктаза |
TAS |
Как видно из таблицы 1, всего с белком соотносится 15 GO с разными кодами
типа достоверности: 5 IEA, 3 IEP, 2 ISO, 3 IDA и 2 TAS. Более подробное
описание этих кодов приведено в таблице 3.
Таблица 3. Описание кодов достоверности2,
использованных в Таблице 1
| Код типа достоверности |
Расшифровка кода типа достоверности |
Объяснение |
Достоверность |
| IEA |
Inferred from Electronic Annotation (аннотация получена
путем электронной обработки) |
Используется для аннотаций, полученных автоматически из
из существующей базы данных и не проверяемых куратором. В частности,
используется для "внутреннего" поиска, когда публикация результата
не предполагается. |
Низкая |
| IEP |
Inferred from Expression Pattern (аннотация основана на паттерне
экспрессии) |
Используется для аннотаций, основанных на сравнении паттерна
экспрессии неописанного гена с паттерном экспрессии гена, для
которого уже известна его роль в том или ином процессе. При этом
аннотация может относиться как к самому гену, так и к РНК или
белку. Этот тип достоверности рекомендуется использовать только
для биологических процессов. |
Высокая |
| ISO |
Inferred from Sequence Orthology (аннотация основана на ортологии
последовательности) |
Присваевается, если аннотация основывается на анализе
ортологов продуктов гена, причем аннотация должна быть
подтверждена экспериментально. Является одной из подкатегорий
кода ISS. |
Средняя |
| IDA |
Inferred from Direct Assay (аннотация основана на прямом
анализе) |
Используется, когда для определения аннотации GO проводился
специальный анализ. Присваевается, когда анализ проводился с
целью изучения естественной функции продукта (иначе используется
код IMP). |
Высокая |
| TAS |
Traceable Author Statement (аннотация основана на отчете
автора, который можно отследить) |
Любое заявление в статье, в которой доказательства (результаты
эксперимента, анализ последовательностей и т. д.) не приведены, но
упоминаются и поэтому их можно отследить (прочитать в другом
источнике). Обычно это связано со статьями-обзорами. |
Высокая |
Если теперь проанализировать данные таблицы 1 с учетом характеристик кодов,
приведенных в таблице 2, можно сказать, что судить о функциях продукта того
или иного гена следует в первую очередь по
GO с достоверными кодами
(в нашем случае это
IEP, IDA, TAS), внимательно нужно анализировать
функции, описанные
GO со средне достоверными кодами (в нашем случае
это
ISO) и только на основе дополнительной информации принимать или
отвергать аннотацию с малодостоверными кодами (в нашем случае это
IEA).