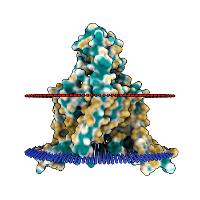
Мембранные белки
Описание белка
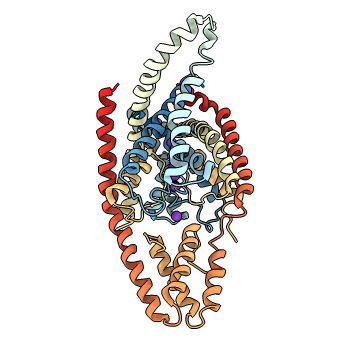 Рис. 1. Структура
исследуемого белка Рис. 1. Структура
исследуемого белка |
В этом практикуме нужно было описать расположение белка в мембране,
а также попробовать предсказать его функции с помощью нескольких
баз данных и сервисов.
Для работы мне был выдан белок с PDB ID
4RUF, образующий калиевые ионные каналы в клетках человека.
Каливые каналы — самый распространенный тип ионных каналов,
найденный практически во всех живых организмах
1. Эти
каналы участвуют и контролируют большое число клеточных
процессов
2,3. Механическую и термическую активацию
этих каналов связывают, к примеру, с термочувствительностью,
чувствами осязания и боли.
Калиевые каналы из семейства
TRAAK/TREK K(2P) (к которому и относится наш белок), участвуют
в изменении возбудимости нейронов в ответ на давление, температуру,
сигнальные липиды и анестетики
4. Механизм все еще неизвестен.
На рис. 1 изображен описанный белок.
1. База данных OPM
База данных
OPM
(Orientation of Proteins in the Membrane) содержит
предсказания о положении белков относительно мембраны. С помощью
этой базы в первом задании необходимо было описать расположение
выданного мне белка в мембране. Затем нужно было точно так же
описать белок, в трансмембранной части которого не α-спирали,
а β-листы. Мною был выбран белок с PDB ID
2JK4 — потенциалзависимый анионный канал во внешней
митохондриальной мембране человека.
В таблице 1 приведена характеристика для двух исследуемых
белков, а на рис. 2-3 их изображения по отношению к мембране
(на этих изображениях внешняя мембрана окрашена красным, а
внутренняя — синим).
Табл. 1. Информация о трансмембранных белках
| Белок |
4RUF (цепь А) |
2JK4 |
| Классификация |
Тип: 1. Трансмембранные белки
Класс: 1.1. Альфа-спиральные политопные белки
Суперсемейство: 1.1.012. Ионные каналы (VIC)
Семейство: 1.1.12.06. Двухпоровые каналы |
Тип: 1. Трансмембранные белки
Класс: 1.3. Бета-бочонковые трансмембранные белки
Суперсемейств: 1.3.022. Митохондриальные и пластидные
порины
Семейство: 1.3.22.01. Потенциал-зависимые анионные
каналы (VDAC) |
| Толщина гидрофобной части мембраны |
31.8 Å |
23.4 Å |
| Трансмембранные спирали/β-тяжи (а.о.) |
1(30-53), 2(142-176), 3(196-218), 4(255-280) |
1(29-36), 2(42-51), 3(58-65), 4(72-79), 5(84-90),
6(98-105), 7(115-122), 8(126-134), 9(141-148),
10(152-160), 11(170-176), 12(182-187), 13(193-199),
14(206-213), 15(222-228), 16(235-241), 17(246-252),
18(258-266), 19(277-285) |
Среднее количество остатков в одной трансмембранной
спирали (или одном β-тяже) белка |
~ 15 а.о. |
~ 8 а.о. |
| В какой мембране находится белок |
плазматическая мембрана эукариот |
наружняя митохондриальная мембрана |
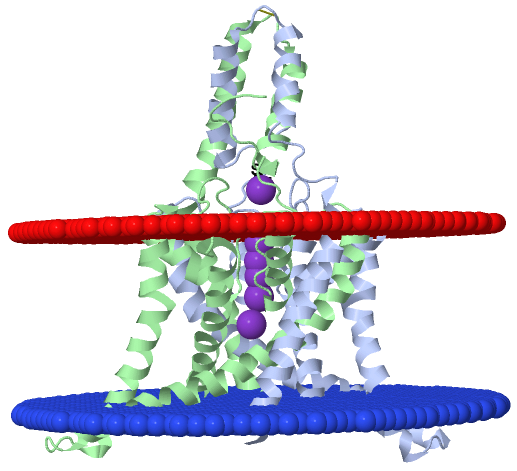 Рис. 2. Расположение
белка 4RUF в эукариотической мембране Рис. 2. Расположение
белка 4RUF в эукариотической мембране |
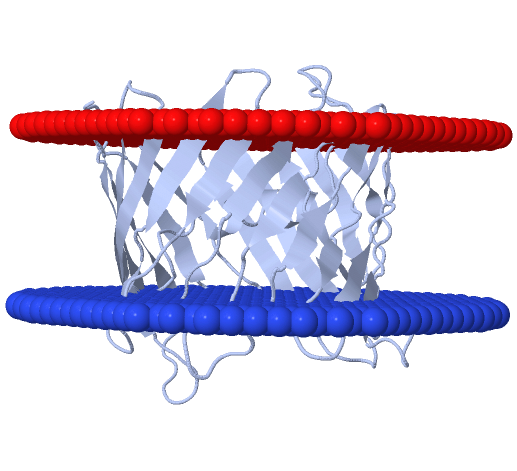 Рис. 3. Расположение
белка 2JK4 в митохондриальной мембране Рис. 3. Расположение
белка 2JK4 в митохондриальной мембране |
2. Анализ предсказания трансмембранных спиралей
В этом задании необходимо было оценить корректность результатов работы
сервисов
TMHMM и
Phobius,
которые используются для предсказания трансмембранных участков белков.
Опишу сначала кратко принципы работы двух сервисов.
TMHMM (TransMembrane Hidden Markov Model) — метод определения
трансмембранных спиралей белка на основе модели Маркова
6. Он
учитывает 7 возможных для а.о. положений относительно мембраны (рис. 4):
- в "ядре" трансмембранной спирали (transmembrane helix core),
т.е. в средней части мембраны в области гидрофобных хвостов липидов;
- по две позиции в "кэпе" трансмембранной спирали (transmembrane
helix caps), т.е. в периферийной части мембраны в области гидрофильных
головок липидов (по одной для каждой поверхности мембраны);
- 3 позиции для петлей: по одной позиции для петли на цитоплазматической
поверхности мембраны (cytoplasmic loop), две для соответственно
короткой (short non-cytoplasmic loop) и длинной петли (long
non-cytoplasmic loop) на наружной поверхности мембраны;
- одна позиция для глобулярного домена (globular) в центре
каждой петли.
Для каждого а.о. рассчитывается постериорная вероятность нахождения в той
или иной позиции, при этом все вероятности для всех а.о. связаны друг
с другом. При расчетах используется метод
максимального правдоподобия.
При расчетах также приняты следующие положения:
- в кэп входит большое число полярных а.о., а общая длина кэпа
составляет 5 а.о.;
- в ядре находится 5-25 по большей части гидрофобных а.о.;
- в петлях на цитоплазматической поверхности мембраны большое
число положительно заряженных а.о.;
- для короткой и длинной петлей на внешней поверхности мембраны
используются разные подходы, так как в отличие от коротких петлей
длинные петли не всегда содержат маленькое число положительно
заряженных а.о.
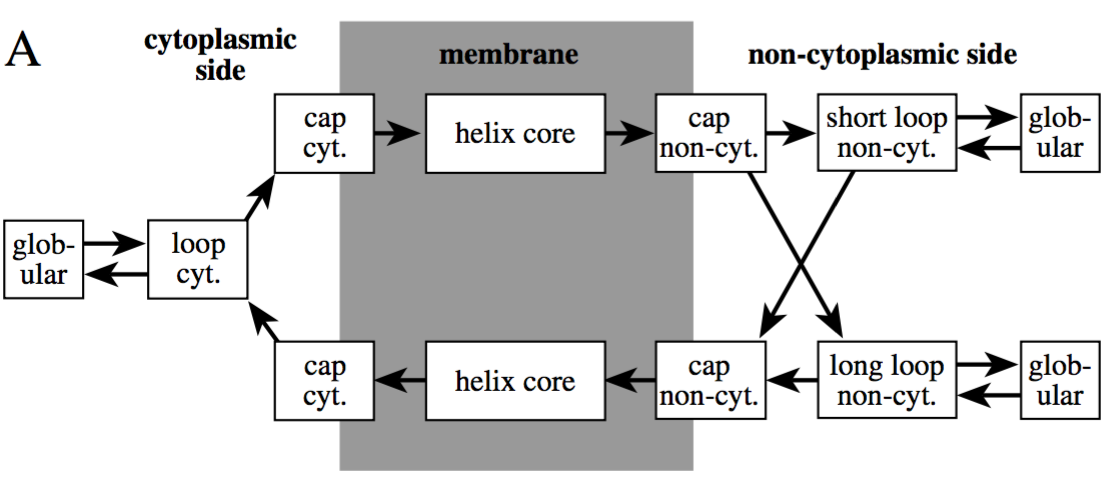 Рис. 4.
Схема модели, используемой в THMHH6 Рис. 4.
Схема модели, используемой в THMHH6
|
Кроме того, эта программа в небольшой части случаев способна отличать
сигнальные пептиды (которые тоже содержат большое количество
гидрофобных а.о.) от трансмембранных участков.
TMHMM также отличает
белки-порины от трансмембранных спиралей
7.
Phobius во многом похожа на
TMHMM, однако эта программа
отличается способностью определять наличие сигнального пептида в белке.
На рис. 5 показана схема модели, используемой в
Phobius'е. Она
отличается от модели
TMHMM присутствием субмодели сигнального
пептида, включающей
n-, h-, c-участки и участок "
cut".
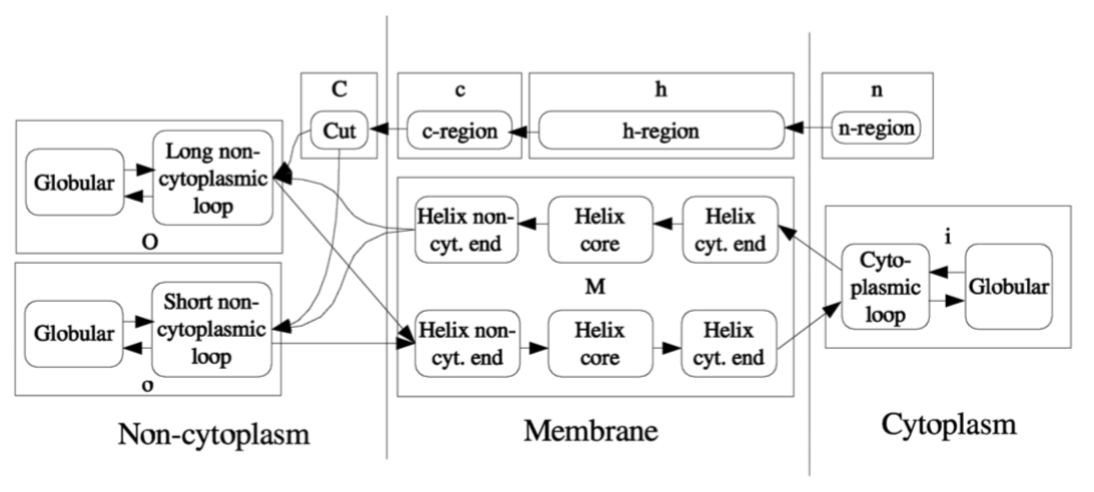 Рис. 5.
Схема модели, используемой в Phobius8 Рис. 5.
Схема модели, используемой в Phobius8
|
Такое усовершенствование модели делает
Phobius более точным
инструментом, чем
TMHMM8.
Перейдем теперь к самому заданию. Для его выполнения последовательность А
цепи белка была подана на вход этим сервисам и результаты были сохранены в
графическом и текстовом видах.
Обсудим сначала выдачу сервиса
Phobius. На рис. 6, 7 приведены
результаты работы программы в текстовом и графическом видах соответственно.
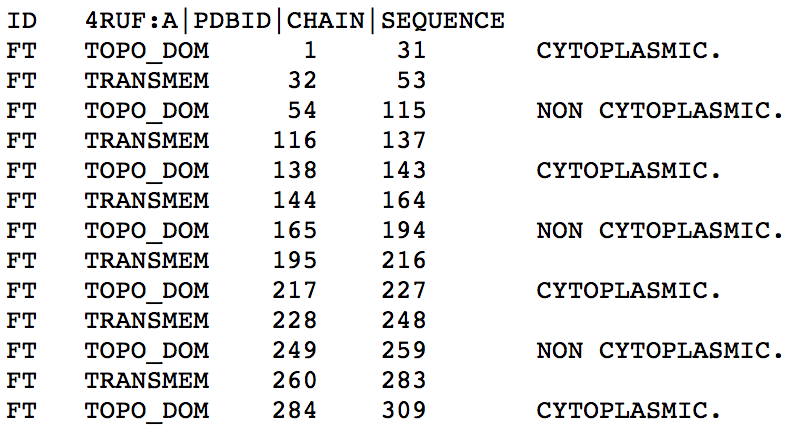 Рис. 6.
Текстовая выдача программы Phobius. Рис. 6.
Текстовая выдача программы Phobius.
TRANSMEM -
трансмембранная часть;
CYTOPLASMIC - часть белка,
обращенная в цитоплазму;
NON CYTOPLASMIC - часть белка,
обращенная наружу
|
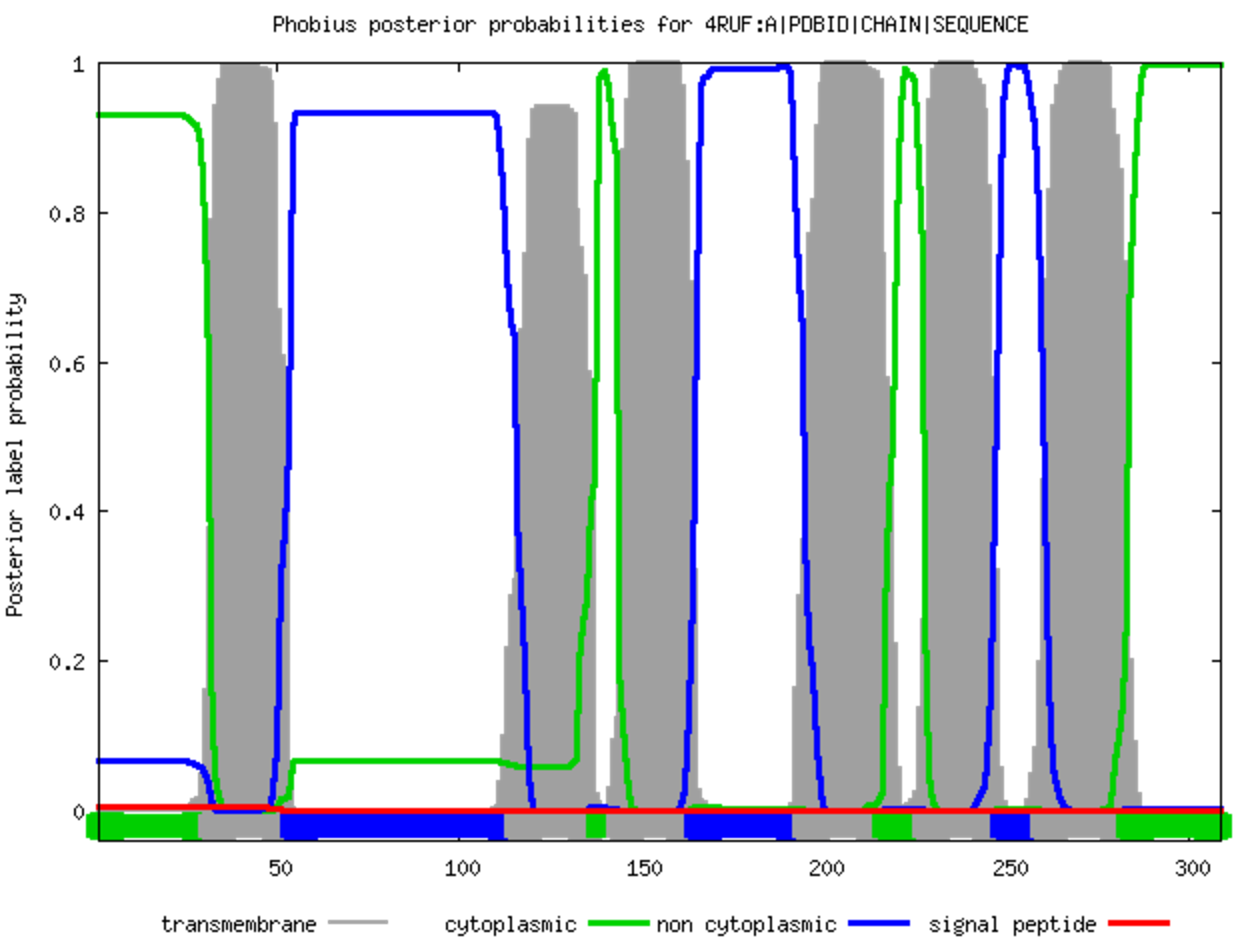 Рис. 7.
Графическая выдача программы Phobius. Рис. 7.
Графическая выдача программы Phobius.
Серый - трансмембранная часть белка;
зеленый - часть белка, обращенная в цитоплазму;
синий - часть белка, обращенная наружу.
По горизонтали - а.о.; по вертикали - вероятность
|
Как видно из рис. 6 и 7, сервис нашел 6 трансмембранных спиралей, в то время
как в реальности их всего 4. Таким образом, было найдено 2 лишних спирали:
116-137 а.о. и 228-248 а.о. Кроме того, для верно найденных спиралей а.о.
найдены неточно: сервис ошибся во всех четырех случаях как минимум на 2 а.о.
Ну и персональная оценка: графический результат неудобен для восприятия,
линии слишком жирные, а "пики" для трансмембранных спиралей какие-то кривые.
В общем, ужасно некрасивый график (чем вообще часто грешат биоинформатические
программы). С другой стороны, таблица мне тут кажется удобнее для чтения, чем
в следующем сервисе.
Теперь обсудим выдачу сервиса
TMHMM. На рис. 8, 9 приведены
результаты работы программы в текстовом и графическом видах соответственно.
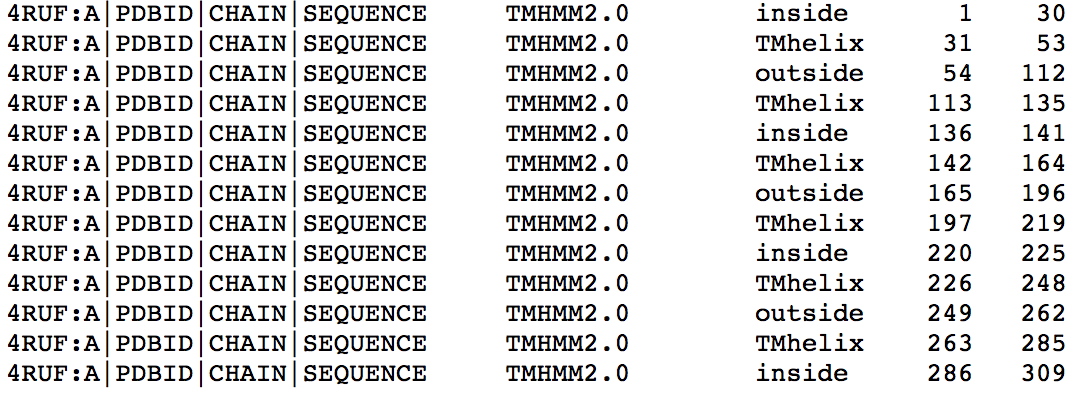 Рис. 8.
Текстовая выдача программы TMHMM. Рис. 8.
Текстовая выдача программы TMHMM.
TMhelix -
трансмембранная часть;
inside - часть белка,
обращенная в цитоплазму;
outside - часть белка,
обращенная наружу
|
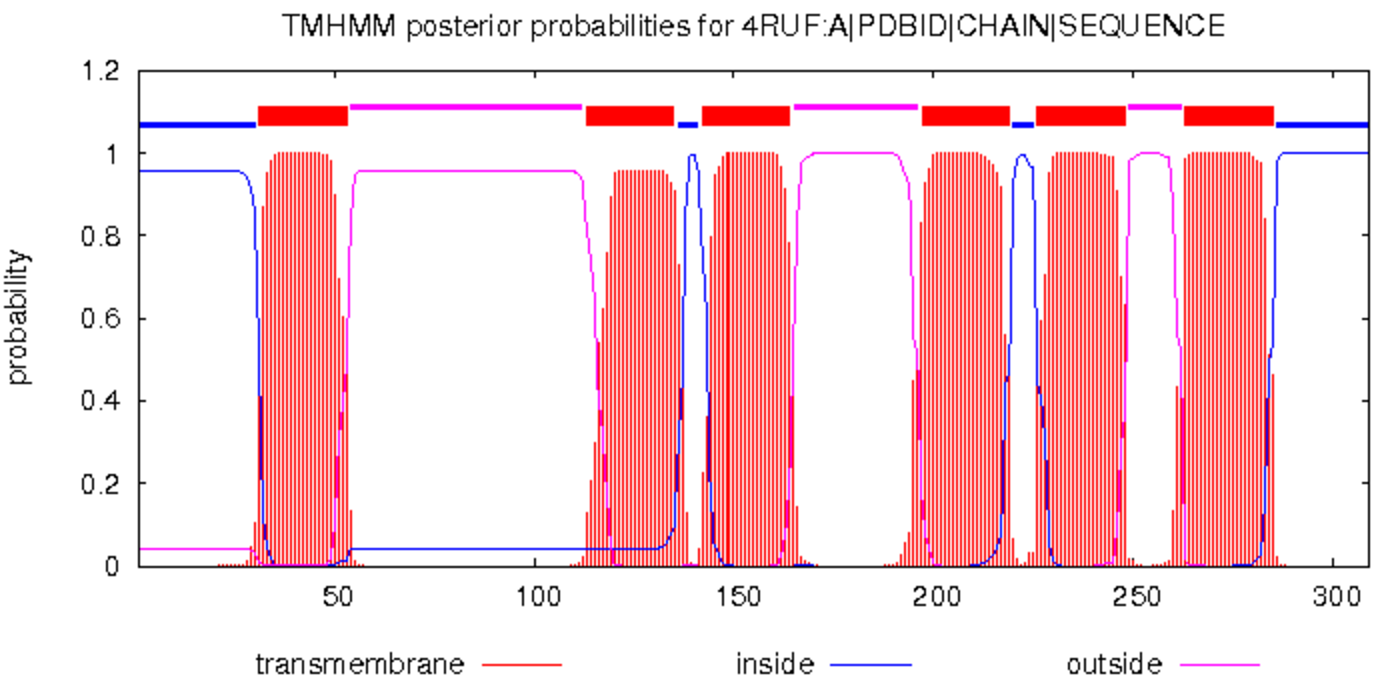 Рис. 9.
Графическая выдача программы TMHMM. Рис. 9.
Графическая выдача программы TMHMM.
Красный - трансмембранная часть белка;
синий - часть белка, обращенная в цитоплазму;
розовый - часть белка, обращенная наружу.
По горизонтали - а.о.; по вертикали - вероятность
|
Как видно из рис. 8, 9 этот сервис также нашел 6 трансмембранных спиралей,
т.е. 2 лишних: 113-135 а.о. и 226-248 а.о. В координатах остальных спиралей
погрешности сравнимы с погрешностями предыдущего сервиса. Что касается
личной оценки: график, на мой взгляд, в этом случае намного приятнее
визуально, зато в таблица сложно воспринимается из-за включения названия
входной последовательности в каждую строчку.
Выводы. В целом, результаты обоих сервисов примерно похожи. В обоих
случаях координаты последних и начальных а.о. для верно найденных
трансмембранных спиралей были определены неточно, а также было найдено
2 лишние трансмембранные спирали и там, и там. Это, скорее всего, реальные
спирали 225-237 а.о. и 119-128 а.о., которые погружены в мембрану, но не
пересекают ее насквозь и, видимо, поэтому в БД
OPM они не аннотированы
как
трансмембранные спирали.
В общем, я бы не стала стопроцентно
полагаться на результаты этих двух программ, если бы мне были нужны
точные координаты трансмембранных спиралей/тяжей для некоторого
неописанного белка, но для получения примерного представления его
расположения в мембране я бы вполне охотно использовала эти сервисы
(особенно второй, от графика первого болят глаза!).
3. База данных TCBD
В этом задании нужно было найти информацию об описанных в задании 2 белках в
БД
TCBD.
В
базе данных транспортеров TCDB (Transport Classification DataBase)
можно найти белки сходные с заданным в базе, почитать про них (со ссылками),
посмотреть комплекс белков по одной субъединице, а также просто узнать
классификацию некоторого белка и его возможные функции.
К сожалению, оба моих белка не представлены в этой БД, поэтому описывать
мне нечего. Если будет нужно, опишу выданный белок с другим идентификатором.
Описание белков (доп. задание)
В ПРОЦЕССЕ
Для белка
4RUF в разных базах данных было найдено описание:
Ссылки:
[1] Littleton J.T., Ganetzky B. (2000). Ion channels and synaptic
organization: analysis of the Drosophila genome.
Neuron,
26 (1): 35–43.
[2] Hille, Bertil (2001). Chapter 5: Potassium Channels and Chloride
Channels.
Ion channels of excitable membranes. pp. 131–168.
[3] Jessell T.M., Kandel E.R., Schwartz J.H. (2000). Chapter 6: Ion Channels.
Principles of Neural Science (4th ed.). pp. 105–124.
[4] Lolicato M., Riegelhaupt P. M., Arrigoni C., Clark K. A., Minor D. L.
(2014). Transmembrane Helix Straightening and Buckling Underlies Activation
of Mechanosensitive and Thermosensitive K2P Channels.
Neuron,
84 (6):
1198–1212.
[5] Human K2P4.1 (TRAAAK) potassium channel, W262S mutant // RCSB PDB.
[URL].
[6] Erik L.L. Sonnhammer, Gunnar von Heijne, Anders Krogh. (1998). A hidden
Markov model for predicting transmembrane helices in protein sequences.
Proc.Sixth Int.Conf. on Intelligent Systems for
Molecular Biology,175-182.
[7] Anders Krogh, Bjorn Larsson, Gunnarvon Heijne, Erik L. L. Sonnhammer. (2001).
Predicting Transmembrane Protein Topology with a Hidden Markov Model: Application to
Complete Genomes.
J. Mol. Biol.,
305: 567-580.
[8] Lukas Ka ̈ll, Anders Krogh, Erik L. L. Sonnhammer. (2004). A Combined Transmembrane
Topology and Signal Peptide Prediction Method.
J. Mol. Biol. ,
338: 1027–1036.