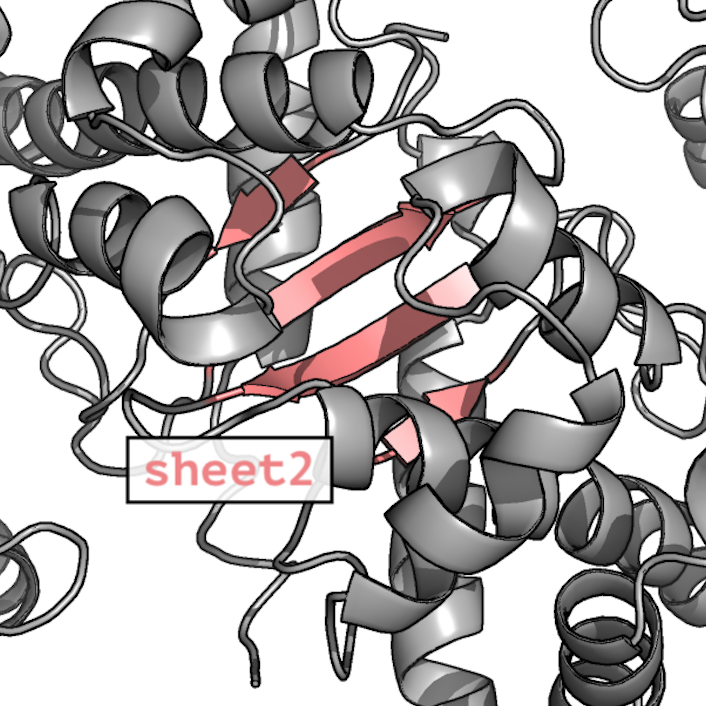
Определение вторичной структуры
Для анализа был взят белок, который используется во всех
практикумах этого семестра — высокостабильная глицерол
киназа 2ZF5. Исследуемый димер показан на рис.1 и покрашен
в соответствие со вторичными структурами.
Вторичные структуры определялись с помощью программ
DSSP и Stride, установленных на kodomo. Полученные файлы с
результатами:
stride.out,
dssp.out. Эти файлы сравнивались с
файлом pdb:
2zf5.pdb.
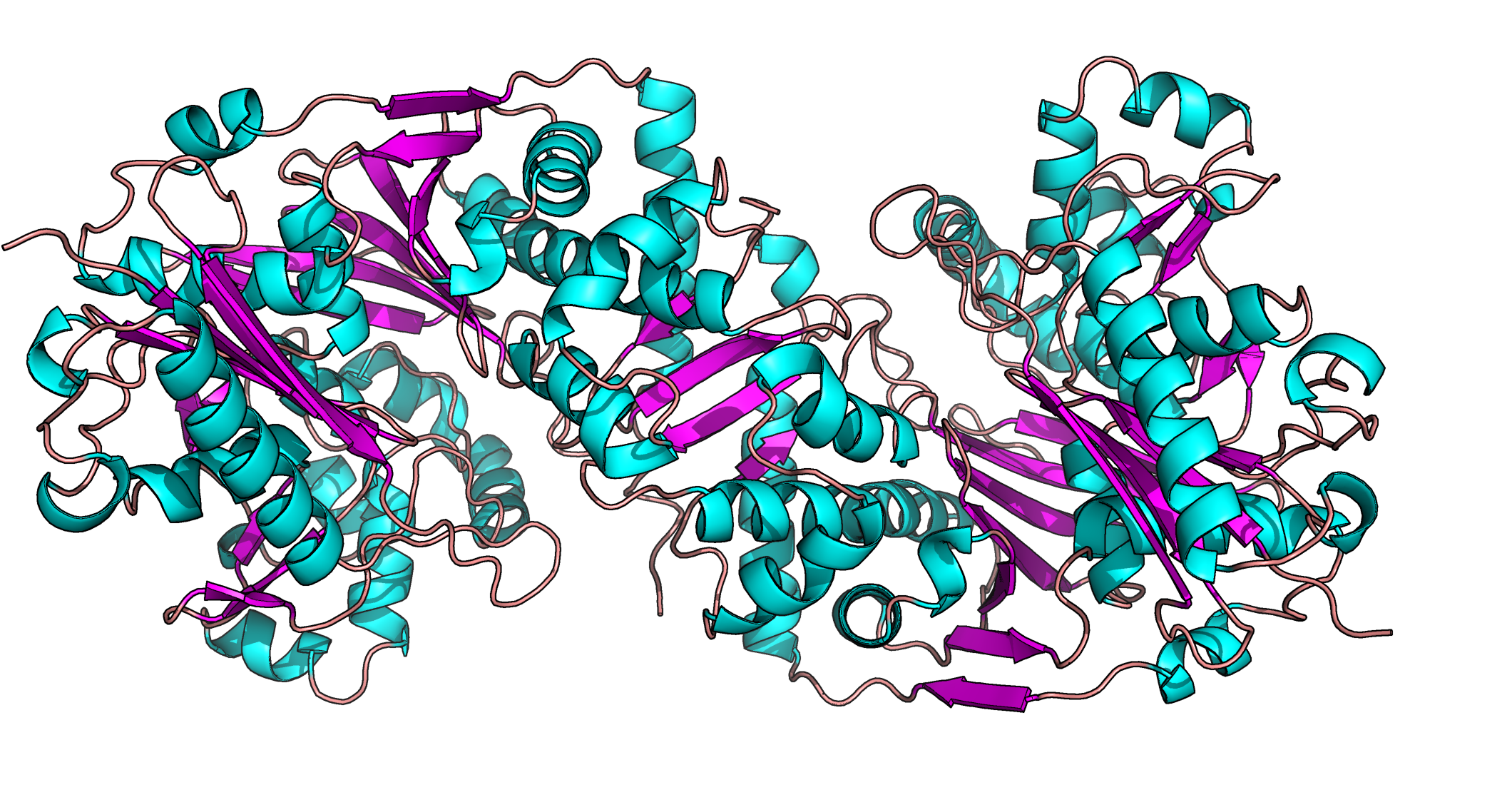
Рис.1. Вторичные структуры в составе глицерол киназы
2ZF5
Для анализа я решила выбрать 4 структуры: 2 альфа-спирали и
2 бета-листа. При этом, в качестве одной из спиралей была взята
очень короткая альфа-спираль, близкая к поверхности (helix2), и
симметричные альфа-спирали из разных цепей для сравнения
предсказания одинаковых участков в разных цепях. В качестве
одного из бета-листов был взят бета-лист, образованный участками
обеих цепей сразу (helix2). На рис. 2 показано расположение
выбранных структур в молекуле глицерол киназы, а в таблице
1 приведено сравнение границ данных структур для pdb файла и
предсказаний упомянутыми выше программами.
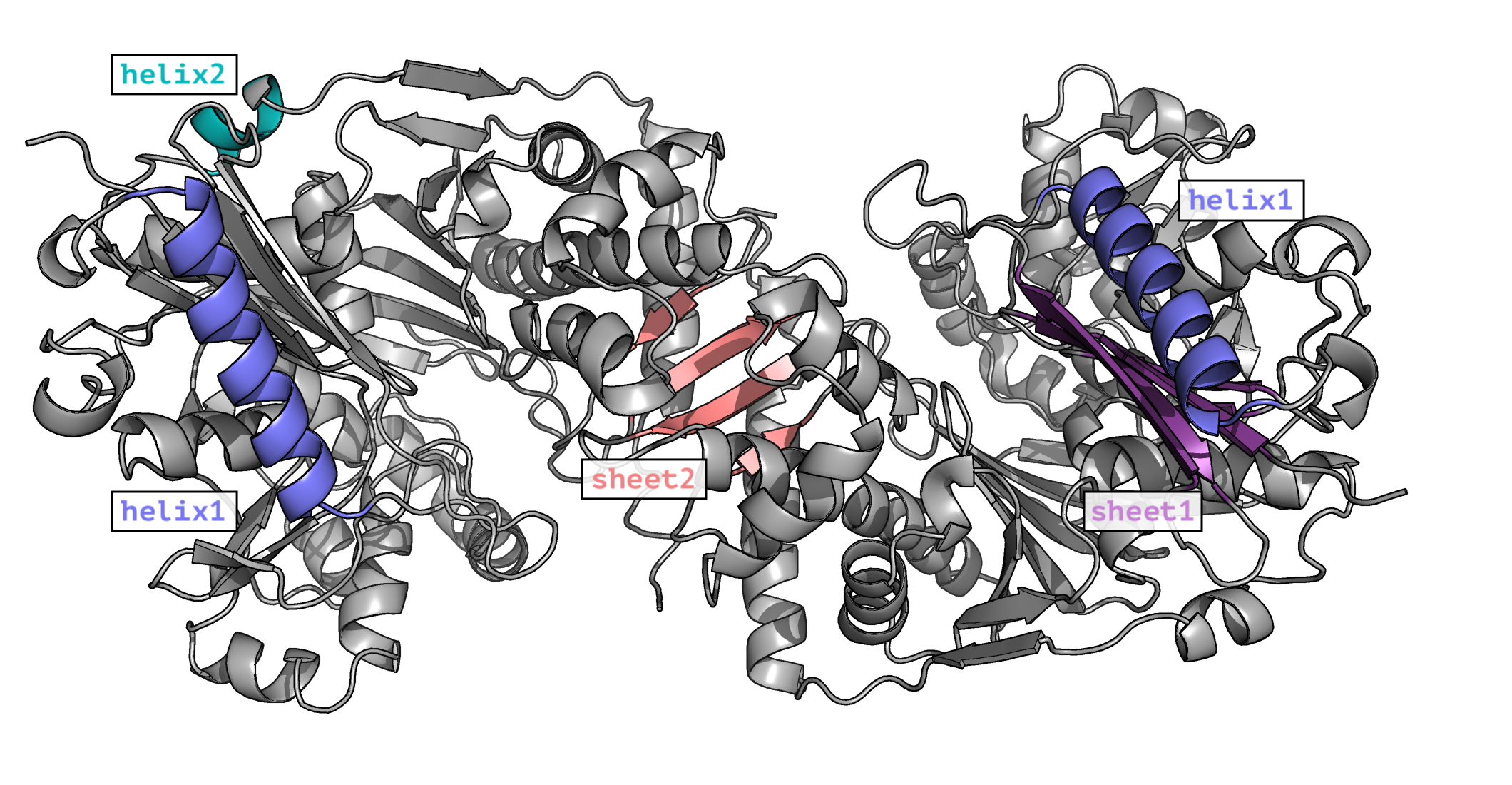
Рис.2. Положение выбранных элементов вторичной
структуры, окрашенных в разные цвета.
Таблица 1. Сравнение границ
выбранных структур в исходном pdb-файле и в файлах
с предсказаниями программ DSSP и Stride
| Структура |
Границы в PDB |
Границы в DSSP |
Границы в Stride |
| helix1 |
ASN47-ARG67 (O, Y) |
PRO48-ALA66 (O, Y) |
PRO48-ALA66 (O, Y) |
| helix2 |
ASP447-GLU453 (Y) |
THR448-ALA452 (Y) |
THR448-LEU454 (Y) |
| sheet1 |
ILE26-GLU33 (O)
SER14-PHE20 (O)
PHE4-GLU10 (O)
ILE73-ASN80 (O)
PRO232-GLY238 (O)
SER216-TYR221 (O)
|
ILE26-GLU33 (O)
SER14-PHE20 (O)
PHE4-GLU10 (O)
ILE73-ASN80 (O)
PRO232-GLY238 (O)
SER216-TYR221 (O)
|
ASN25-GLU33 (O)
SER14-ASP21 (O)
PHE4-GLU10 (O)
ILE73-ASN80 (O)
ILE231-GLY238 (O)
SER216-THR222 (O)
|
| sheet2 |
TYR336-VAL338 (O)
GLY355-ILE360 (O)
GLY355-ILE360 (Y)
TYR336-VAL338 (Y)
|
TYR336-VAL338 (O)
GLY355-ILE360 (O)
GLY355-ILE360 (Y)
TYR336-VAL338 (Y)
|
TYR336-VAL338 (O)
GLY355-ILE360 (O)
GLY355-ILE360 (Y)
TYR336-VAL338 (Y)
|
Исходя из данных, указанных в таблице, в целом, бета-листы определяются более
похожим способом, нежели альфа-спирали. Я склонна верить разметке бета-листов
в pdb и DSSP.
В случае альфа-спиралей разметка отличается на 1-2 а.о., что, как мне кажется,
скорее вопрос принятых договоренностей: включать малоструктурированные концы
или нет (см. рис. 3). Стоит также отметить, что разметки, полученные программами DSSP
и Stride совпадают для helix1 и ограничивают более короткую спираль, которая
мне кажется более корректной.
Для helix2 мне больше нравится разметка Stride, так как она добавляет
неокрашенный альфа-спиральный участо. Стоит, однако, отметить, что в случае DSSP
невошедшие в альфа-спираль остатки 453-454 отмечены как элементы hydrogen bonded
turn. Вообще, то, что разметка этой вторичной структуры самая разная, неудивительно,
так как спираль короткая и выходит на поверхность белка.
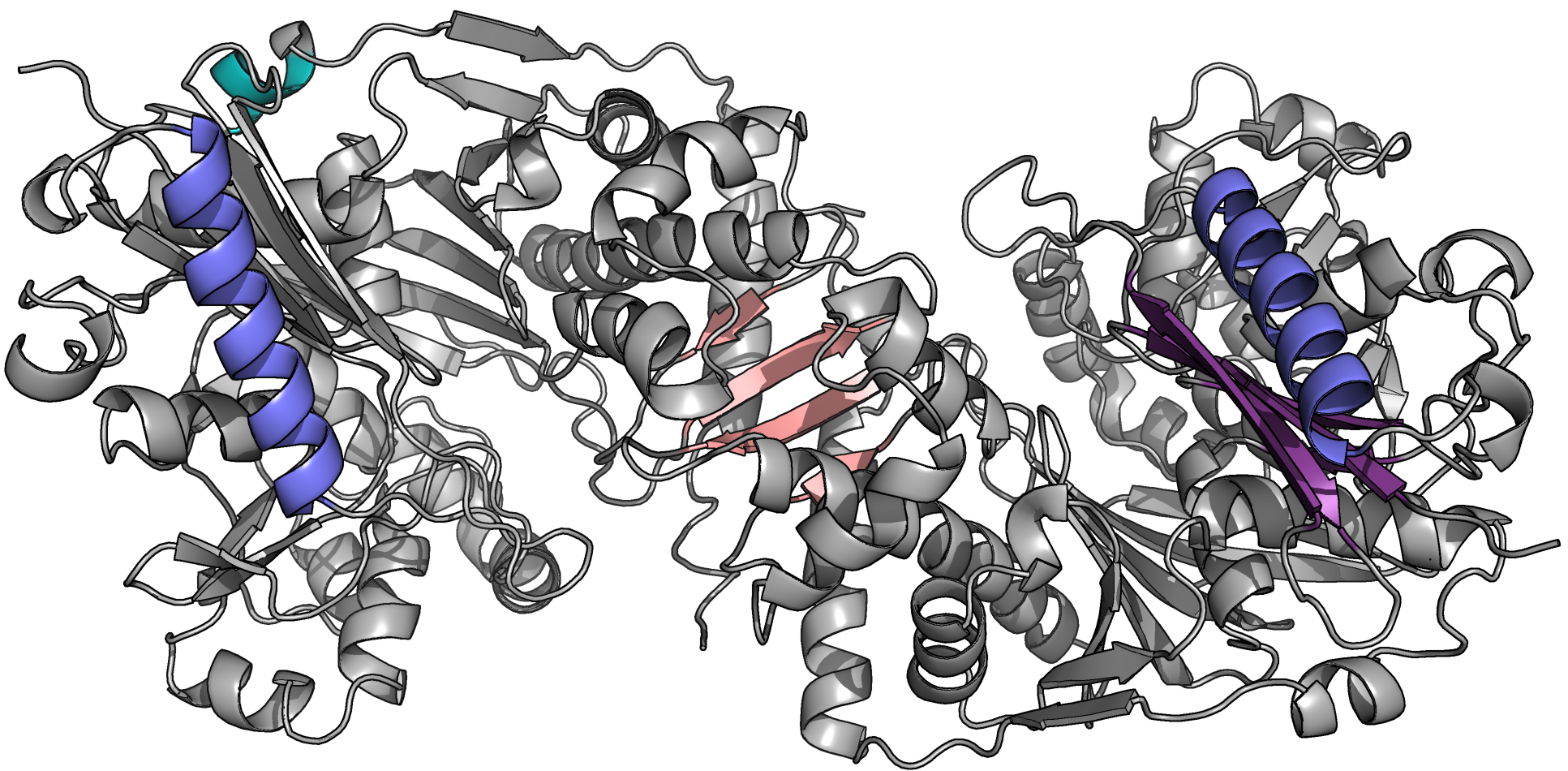
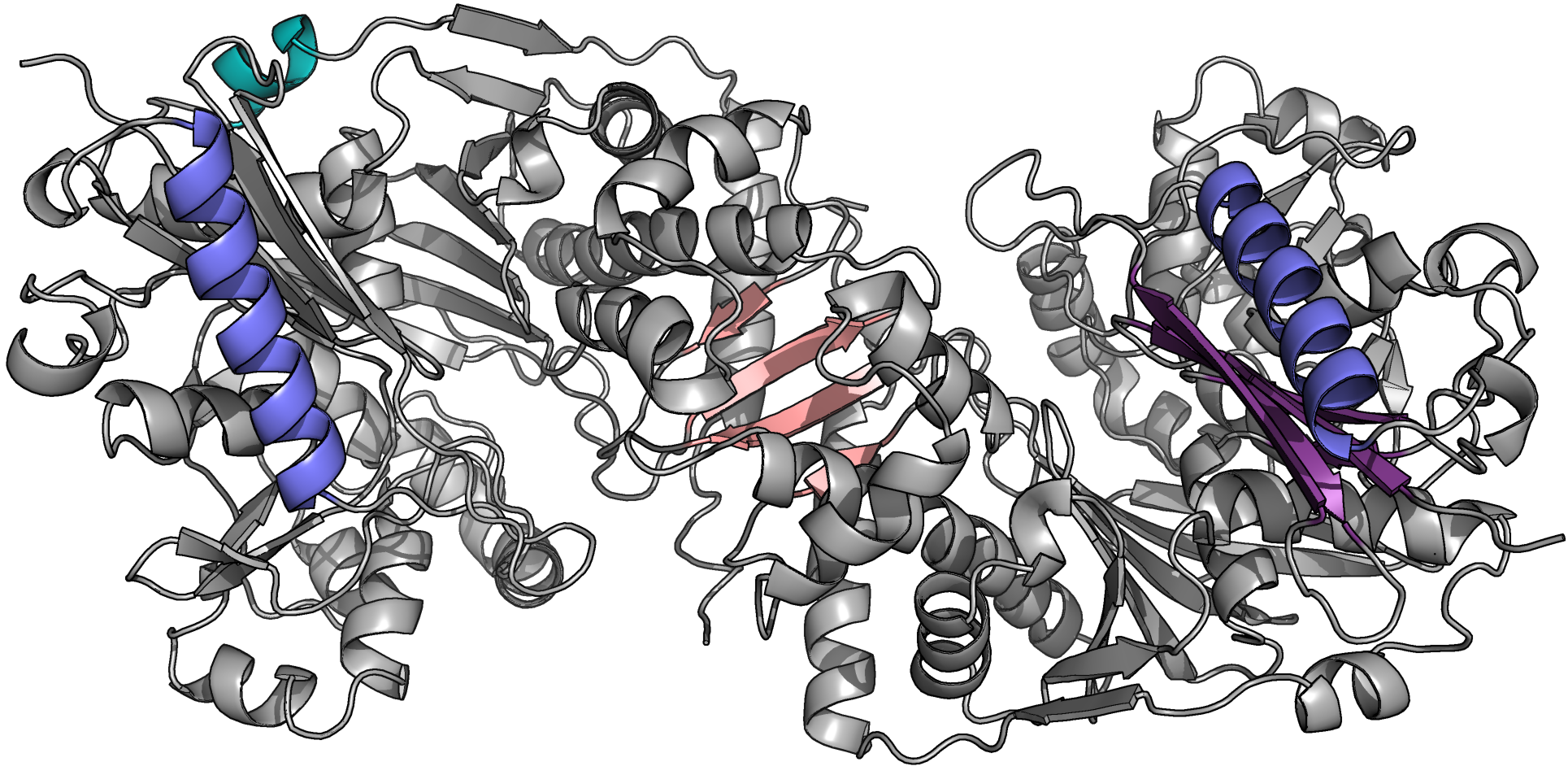
Рис.3. Положение выбранных элементов вторичной
структуры, окрашенных в разные цвета, на основании предсказаний
DSSP (слева) и Stride (справа)