Полезные ссылки
Программа BLASTP.
I. Поиск гипотетических гомологов белка RISA_ECOLI в разных БД.
| Поиск по БД Swiss-Prot | Поиск по БД PDB | Поиск по БД "nr" | |
| 1. Лучшая находка (в принципе должна соответствовать заданному белку) | |||
| Идентификатор БД | RISA_ECOLI | 1I8D | NP_416179.1 |
| E-value | 2e-123 | 2e-124 | 3e-122 |
| Вес (в битах) | 440 | 440 | 440 |
| % идентичности | 100% | 100% | 100% |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? Если найдены, то укажите общее число и приведите один идентификатор (любой, но желательно Swiss-Prot ID) |
нет | нет | нет |
| 2. Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний, Descriptions, с E-value < 1E-10) | 21 | 4 | 1156 |
| 2. "Худшая" находка (последняя в выдаче с E-value < 1) | |||
| Номер находки в списке описаний (Descriptions) | 21 | 5 | 1238 |
| Идентификатор БД | LUXP_PHOLE | 2JER | ZP_03225981.1 |
| E-value | 7e-16 | 0.80 | 0.022 |
| Вес (в битах) | 83.6 | 29.6 | 42,7 |
| % идентичности | 31% | 30% | 39% |
| % сходства | 50% | 47% | 58% |
| Длина выравнивания | 184 | 70 | 58 |
| Координаты выравнивания (номера первых и последних а.о.) | 1 - 184 RISA_ECOLI и 1 - 182 LUXP_PHOLE | 77 - 138 RISA_ECOLI и 193 - 262 2JER | 130 - 185 RISA_ECOLI и 15 - 72 ZP_03225981.1 |
| % гэпов | 1% | 11% | 3% |
Данный белок удалось найти во всех трех банках данных. Параметры выравнивания изучаемого белка с самим собой при поиске по разным БД никак не отличаются (кроме Evalue - оно различно для разных БД, так как зависит от количества информации, содержащейся в них). Самый большой по объему банк данных - nr (некурируемый), затем SwissProt (курируемый), и самый маленький - PDB (3D-структура). Число потенциальных гомологов уменьшается в этом ряду. Худшие находки не совпадают, так как БД по которым производился поиск совершенно необязательно должны содержать в себе одну и ту же последовательность. То есть не каждую последовательность аннотируют и тем более находят для нее третичную структуру.
Из этих "худших" находок лучшей, несомненно является LUXP_PHOLE, так как Evalue = 7e-16 позволяет нам сделать вывод о его гомологии с RISA_ECOLI.
Не могу не упомянуть одну очень интересную находку, а именно LUXY_VIBFI - Желтый флуоресцентный белок бактерии Vibrio fischeri. Evalue = 2e-17 (!). Поэтому вывод такой: гомологи не всегда выполняют сходные функции.
II. Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам.
Первый полученный гомолог (с Evalue < 0.001) - RISA_MYCTU был найден среди организмов таксона Actinobacteria.| Номер находки в списке описаний (Descriptions) | 1 |
| Идентификатор БД | RISA_MYCTU |
| E-value | 8e-26 |
| Вес (в битах) | 112 |
| % идентичности | 36% |
| % сходства | 54% |
| Длина выравнивания | 203 |
| Координаты выравнивания (номера первых и последних а.о.) | 1 - 199 RISA_ECOLI и 1 - 197 RISA_MYCTU. |
| % гэпов | 4% |
III. Поиск белка по его фрагменту.
>seq2 GHLLSGHVFGTAEIFLIKENRДанный фрагмент относится к белку RISA_CHLPN.
| Поиск по фрагменту | Поиск по полной последовательности |
|
| АС лучшей находки | Q9Z820 | Q9Z820 |
| E-value | 2e-12 |
6e-112 |
| Вес (в битах) | 69.4 | 402 |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? |
нет | нет |
Выравнивание, построенное программой BLASTP (Query = RISA_CHLPN, Sbjct = RISA_ECOLI):
Фрагмент выровненный в пробном выравнивании помечен (*)
Score = 164 bits (414), Expect = 3e-40, Method: Compositional matrix adjust.
Identities = 93/201 (46%), Positives = 128/201 (63%), Gaps = 5/201 (2%)
Query 1 MFSGIIQELGEVCFFEAQGNGLSLGIKSTPLFVTPLVTGDSVAVDGVCLTLTSCNESKIF 60
MF+GI+Q ++ + + N + ++ + L TG SVA +G CLT+T N + +
Sbjct 1 MFTGIVQGTAKLVSIDEKPNFRTHVVELPDHMLDGLETGASVAHNGCCLTVTEINGNHVS 60
************** ****** *
Query 61 FDVIPETLACTTLGEKRCSDQVNLEAALKMGDSIGGHLLSGHVFGTAEI--FLIKEN--R 116
FD++ ETL T LG+ + D VN+E A K D IGGHL+SGH+ TAE+ L EN +
Sbjct 61 FDLMKETLRITNLGDLKVGDWVNVERAAKFSDEIGGHLMSGHIMTTAEVAKILTSENNRQ 120
Query 117 YYFR-GSKELSQYLFEKGFIAIDGISLTLVSVDSDTFSVGLIPETLQRTTLGKKREGERV 175
+F+ +L +Y+ KGFI IDGISLT+ V F V LIPETL+RTTLGKK+ G RV
Sbjct 121 IWFKVQDSQLMKYILYKGFIGIDGISLTVGEVTPTRFCVHLIPETLERTTLGKKKLGARV 180
Query 176 NIEIDMSTKIQVDTVKRILAS 196
NIEID T+ VDTV+R+LA+
Sbjct 181 NIEIDPQTQAVVDTVERVLAA 201
Сравним его с полученными вручную выравниваниями:
Query 95 GHLLSGHVFGTAEI--FLIKEN--R 116
GHL+SGH+ TAE+ L EN +
Sbjct 95 GHLMSGHIMTTAEVAKILTSENNRQ 120
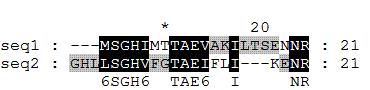
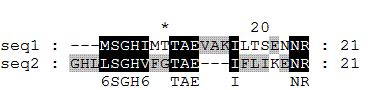
IV. Сравнение выравниваний, выданных программой BLASTP, с оптимальными глобальным и локальным выравниваниями.
В построенных выравниваниях штраф за создание гэпа был 11, за удлинение - 1.1) Оптимальное глобальное выравнивание последовательностей белков RISA_CHLPN и RISA_ECOLI.
#=======================================
#
# Aligned_sequences: 2
# 1: RISA_CHLPN
# 2: RISA_ECOLI
# Matrix: EBLOSUM62
# Gap_penalty: 11.0
# Extend_penalty: 1.0
#
# Length: 213
# Identity: 93/213 (43.7%)
# Similarity: 128/213 (60.1%)
# Gaps: 13/213 ( 6.1%)
# Score: 407.0
#
#
#=======================================
RISA_CHLPN 1 MFSGIIQELGEVCFFEAQGNGLSLGIKSTPLFVTPLVTGDSVAVDGVCLT 50
||:||:|...::...:.:.|..:..::.....:..|.||.|||.:|.|||
RISA_ECOLI 1 MFTGIVQGTAKLVSIDEKPNFRTHVVELPDHMLDGLETGASVAHNGCCLT 50
RISA_CHLPN 51 LTSCNESKIFFDVIPETLACTTLGEKRCSDQVNLEAALKMGDSIGGHLLS 100
:|..|.:.:.||::.|||..|.||:.:..|.||:|.|.|..|.|||||:|
RISA_ECOLI 51 VTEINGNHVSFDLMKETLRITNLGDLKVGDWVNVERAAKFSDEIGGHLMS 100
RISA_CHLPN 101 GHVFGTAEI--FLIKEN--RYYFR-GSKELSQYLFEKGFIAIDGISLTLV 145
||:..|||: .|..|| :.:|: ...:|.:|:..||||.|||||||:.
RISA_ECOLI 101 GHIMTTAEVAKILTSENNRQIWFKVQDSQLMKYILYKGFIGIDGISLTVG 150
RISA_CHLPN 146 SVDSDTFSVGLIPETLQRTTLGKKREGERVNIEIDMSTKIQVDTVKRILA 195
.|....|.|.||||||:|||||||:.|.|||||||..|:..||||:|:||
RISA_ECOLI 151 EVTPTRFCVHLIPETLERTTLGKKKLGARVNIEIDPQTQAVVDTVERVLA 200
RISA_CHLPN 196 SSGKD-------- 200
:....
RISA_ECOLI 201 ARENAMNQPGTEA 213
2) Оптимальное локальное выравнивание последовательностей белков RISA_CHLPN и RISA_ECOLI.
#=======================================
#
# Aligned_sequences: 2
# 1: RISA_CHLPN
# 2: RISA_ECOLI
# Matrix: EBLOSUM62
# Gap_penalty: 11.0
# Extend_penalty: 1.0
#
# Length: 201
# Identity: 93/201 (46.3%)
# Similarity: 128/201 (63.7%)
# Gaps: 5/201 ( 2.5%)
# Score: 412.0
#
#
#=======================================
RISA_CHLPN 1 MFSGIIQELGEVCFFEAQGNGLSLGIKSTPLFVTPLVTGDSVAVDGVCLT 50
||:||:|...::...:.:.|..:..::.....:..|.||.|||.:|.|||
RISA_ECOLI 1 MFTGIVQGTAKLVSIDEKPNFRTHVVELPDHMLDGLETGASVAHNGCCLT 50
RISA_CHLPN 51 LTSCNESKIFFDVIPETLACTTLGEKRCSDQVNLEAALKMGDSIGGHLLS 100
:|..|.:.:.||::.|||..|.||:.:..|.||:|.|.|..|.|||||:|
RISA_ECOLI 51 VTEINGNHVSFDLMKETLRITNLGDLKVGDWVNVERAAKFSDEIGGHLMS 100
RISA_CHLPN 101 GHVFGTAEI--FLIKEN--RYYFR-GSKELSQYLFEKGFIAIDGISLTLV 145
||:..|||: .|..|| :.:|: ...:|.:|:..||||.|||||||:.
RISA_ECOLI 101 GHIMTTAEVAKILTSENNRQIWFKVQDSQLMKYILYKGFIGIDGISLTVG 150
RISA_CHLPN 146 SVDSDTFSVGLIPETLQRTTLGKKREGERVNIEIDMSTKIQVDTVKRILA 195
.|....|.|.||||||:|||||||:.|.|||||||..|:..||||:|:||
RISA_ECOLI 151 EVTPTRFCVHLIPETLERTTLGKKKLGARVNIEIDPQTQAVVDTVERVLA 200
RISA_CHLPN 196 S 196
:
RISA_ECOLI 201 A 201
3) Выравнивание, полученное программой BLASTP:
Score = 164 bits (414), Expect = 3e-40, Method: Compositional matrix adjust.
Identities = 93/201 (46%), Positives = 128/201 (63%), Gaps = 5/201 (2%)
Query 1 MFSGIIQELGEVCFFEAQGNGLSLGIKSTPLFVTPLVTGDSVAVDGVCLTLTSCNESKIF 60
MF+GI+Q ++ + + N + ++ + L TG SVA +G CLT+T N + +
Sbjct 1 MFTGIVQGTAKLVSIDEKPNFRTHVVELPDHMLDGLETGASVAHNGCCLTVTEINGNHVS 60
************** ****** *
Query 61 FDVIPETLACTTLGEKRCSDQVNLEAALKMGDSIGGHLLSGHVFGTAEI--FLIKEN--R 116
FD++ ETL T LG+ + D VN+E A K D IGGHL+SGH+ TAE+ L EN +
Sbjct 61 FDLMKETLRITNLGDLKVGDWVNVERAAKFSDEIGGHLMSGHIMTTAEVAKILTSENNRQ 120
Query 117 YYFR-GSKELSQYLFEKGFIAIDGISLTLVSVDSDTFSVGLIPETLQRTTLGKKREGERV 175
+F+ +L +Y+ KGFI IDGISLT+ V F V LIPETL+RTTLGKK+ G RV
Sbjct 121 IWFKVQDSQLMKYILYKGFIGIDGISLTVGEVTPTRFCVHLIPETLERTTLGKKKLGARV 180
Query 176 NIEIDMSTKIQVDTVKRILAS 196
NIEID T+ VDTV+R+LA+
Sbjct 181 NIEIDPQTQAVVDTVERVLAA 201
| Оптимальное глобальное | Оптимальное локальное | Построенное BLASTP | |
| Score | 407 | 412 | 414 |
| Длина выравнивания | 213 | 201 | 201 |
| Процент идентичности | 93/213 (43.7%) | 93/201 (46.3%) | 93/201 (46%) |
| Процент сходства | 128/213 (60.1%) | 128/201 (63.7%) | 128/201 (63%) |
Локальное выравнивание абсолютно точно совпадает с построенным BLASTP, а глобальное содержит его в себе без каких либо изменений. Замечу, что для идентичных выравниваний, полученных с помощью BLASTP и water получились разные значения веса выравнивания, хотя штрафы за гэпы были одинаковыми. Вероятно это связано с отличиями в алгоритмах этих программ относительно учета этих штрафов.