Полезные ссылки
SCOP и CATH.
1. Классификация доменов согласно SCOP.В записи 1PKV содержится два идентичных домена:
- Координаты: цепь A: 1-87, цепь B: 1-87.
- Классификация по SCOP:
- класс - бета белки (All beta proteins)
- укладка - простой домен фактора редуктазы/изомеразы/элонгации (Reductase/isomerase/elongation factor common domain)
- суперсемейство - подобные доменам рибофлавинсинтазы (Riboflavin synthase domain-like)
- семейство - рибофлавинсинтаза (Riboflavin synthase)
- В данном суперсемействе содержится 3 семейства.
- Данную укладку имеют 4 суперсемейства.
- Координаты: цепь A, 1 - 67.
- Классификация по SCOP:
- класс - бета белки (All beta proteins)
- укладка - OB-укладка (OB-fold)
- суперсемейство - белки, связывающиеся с нуклеиновыми кислотами (Nucleic acid-binding proteins)
- семейство - подобные ДНК-связывающим холодового шока (Cold shock DNA-binding domain-like)
- В данном суперсемействе содержится 16 семейств.
- Данную укладку имеют 16 суперсемейств.
- Координаты: цепь A, 68-298.
- Классификация по SCOP:
- класс - альфа и бета белки (Alpha and beta proteins (a/b))
- укладка - содержащие P-петлю нуклеозидтрифосфатгидролазы (P-loop containing nucleoside triphosphate hydrolases)
- суперсемейство - содержащие P-петлю нуклеозидтрифосфатгидролазы (P-loop containing nucleoside triphosphate hydrolases)
- семейство - G-белки (G proteins)
- В данном суперсемействе содержится 24 семейства.
- Данную укладку имеют 24 суперсемейства.
для доменов записи 1PKV:
- Координаты: цепь A: 1-87, цепь B: 1-87.
- Классификация по CATH - 2.40.30.20.4.1.1.1.5
- Классификация по CATH:
- класс - большей частью бета(Mainly Beta)
- архитектура - бета баррель (Beta Barrel)
- топология - фактор элонгации Tu; домен 3 (Elongation Factor Tu (Ef-tu); domain 3)
- В данном суперсемействе содержится 5 семейств.
- Данную топологию имеют 8 суперсемейств.
- Координаты: цепь A, - (-7) - 65.
- Классификация по CATH - 2.40.50.140.28.1.1.1.1
- Классификация по CATH:
- класс - большей частью бета(Mainly Beta)
- архитектура - бета баррель (Beta Barrel)
- топология - OB - укладка (дигидролипидоамид-ацетилтрансфераза) (OB fold (Dihydrolipoamide Acetyltransferase, E2P))
- суперсемейство - белки, связывающиеся с нуклеиновыми кислотами (Nucleic acid-binding proteins)
- В данном суперсемействе содержится 89 семейств.
- Данную топологию имеют 17 суперсемейств.
- Координаты: цепь A, - 66 - 230.
- Классификация по CATH - 3.40.50.300.106.1.1.1.1
- Классификация по CATH:
- класс - альфа - бета (Alpha Beta)
- архитектура - 3-х слойный (аба) сэндвич(3-Layer(aba) Sandwich)
- топология - Укладка Россмаанна (Rossmann fold)
- суперсемейство - содержащие P-петлю нуклеотидтрифосфатгидролазы (P-loop containing nucleotide triphosphate hydrolases)
- В данном суперсемействе содержится 208 семейств.
- Данную топологию имеют 120 суперсемейств.
- Координаты: цепь A, - 231 - 298.
- Классификация по CATH - 1.10.40.50.1.1.1.1.1
- Классификация по CATH:
- класс - большей частью альфа (Mainly Alpha)
- архитектура - Ортогональная связка (Orthogonal Bundle)
- топология - Рибонуклеотидредуктаза белок R1; домен 1 (Ribonucleotide Reductase Protein R1; domain 1)
- суперсемейство - Предположительно ГТФ-аза; домен 3 (Probable gtpase engc; domain 3)
- В данном суперсемействе содержится 2 семейства.
- Данную топологию имеют 4 суперсемейства.
Прежде всего в классификации СATH для записи 1T9H было три домена, против двух по классификации SCOP (2 домена здесь объединены в один)
Кроме того суперсемйства CATH судя по этим примерам содержат в себе больше семейств, и вообще - классификация здесь подробней.
Те домены которые совпали по координатам, несмотря на разность в терминологии, имеют одинаковую классификацию.
Еще удивили координаты для N-концевого домена в записи 1T9H - они начинаются с (-7).
4. Выравнивание двух доменов имеющих одну укладку, но из разных суперсемейств:
Была взята хорошо знакомая запись 1PKV и 1EFU - в ней домен (цепь А 205-296) имеет ту же укладку (Reductase/isomerase/elongation factor common domain), но принадлежит суперсемейству Translation proteins.
Выравнивание было получено с помощью команды pair_fit в PyMOL:
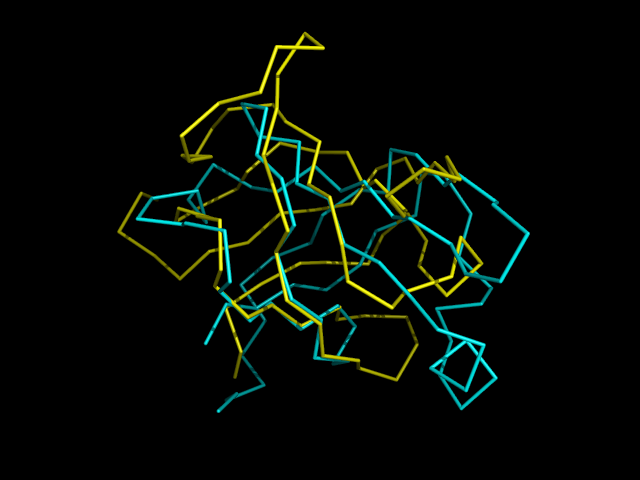
Хотя структуры визуально не очень хорошо совпадают, все же видны общие особенности структуры (например специфическое расположение бета тяжей). Таким образом даже принадлежность доменов к одной топологии свидетельствует о значительном сходстве их структур.