Практикум 4
Паралоги, визуализация.
Здание 1.
1) Необходимо было найти в своих выбранных бактериях достоверные
гомологи белка CLPX_ECOLI и построить по ним дерево. Полные семи бактерий были скопированы из папки
"/P/y17/term4/Proteomes" и объединены в один файл командой: "cat *.fasta > proteones.fa."
2) База данных для локального blast: "makeblastdb -dbtype prot -in proteones.fa -out proteones."
3) Из файла с полным протеомом E. coli я вырезала последовательность белка CLPX_ECOLI и сохранила в отдельный файл clpx_ecoli.fasta:
"seqret sw:clpx_ecoli -auto"
4)При помощи программы blastp был произведен поиск гомологов, с порогом на E-value = 001, по протеомам отобранных
бактерий: "blastp -query clpx_ecoli.fasta -db proteones -evalue 0.001 -out clpx.blastp"
Список находок:
Файл
C помощью Uniprot я получила последовательности белков и оставила только идентификаторы.
С помощью MEGA(MUSCLE) были выровнены последовательности белков и построено дерево по методу Neighbor-joining. Скобочная формула:
Скобки
Ортологи: CLPX_BACSU - CLPX_BACAN, CLPY_BACSU - CLPY_GEOKA, CLPE_BACSU-Q5L436_GEOKA.
Паралоги: CLPY_BACSU - CLPX_BACSU, CLPX_MOOTA - Q2RJP5_MPOOTA, Q5L3T1_GEOKA - HSLU_GEOKA.
На рисунке номер 1 4мя разными цветами раскрашены ортологичные группы, на рисунке 2 - то же самое изображение, но ортологичные группы
схлопнуты. Дерево белков только местами схоже с древом бактерий.
 рисунок 1
рисунок 1
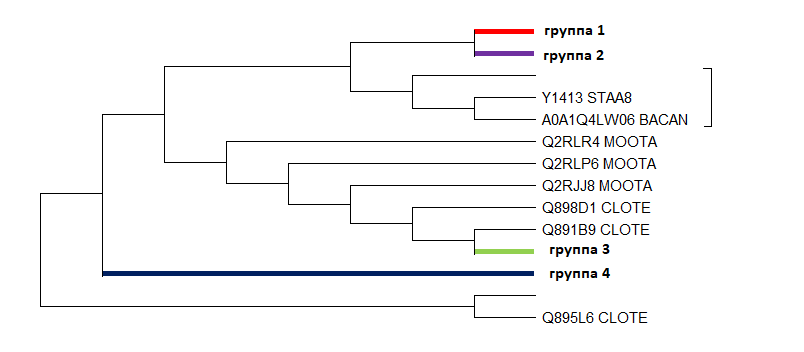 рисунок 2
рисунок 2
 рисунок 1
рисунок 1 рисунок 1
рисунок 1 рисунок 2
рисунок 2