Занятие 8. Структура тРНК
-
PDB - 1QTQ;
Название структуры - GLUTAMINYL-TRNA SYNTHETASE COMPLEXED WITH TRNA AND AN AMINO Организм - ESCHERICHIA COLI Идентификатор цепи РНК – B Дополнительные макромолекулы – Белок - GLUTAMINYL-TRNA SYNTHETASE
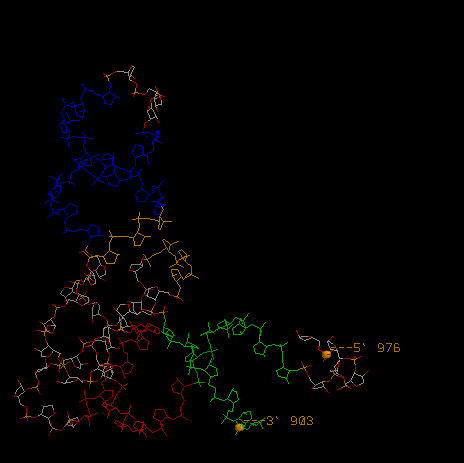
Последовательность РНК U G G G G U A U C G C C A A G C G G U A A G G C A C C G G A U U C U G A U U C C G G C A U U C C G A G G U U C G A A U C C U C G U A C C C C A G C C A Модифицированные основания - SO4 SULFATE ION - QSI 5'-O-[N-(L-GLUTAMINYL)-SULFAMOYL] ADENOSINE Последовательность РНК начинается с 902 номера и заканчивается 976. Однако в PDB файле пропущены нуклеотиды U G находящиеся в начале цепи. Номер 917 пропущен.U GGGGUA UC GCC AAGCGGUAA GGC ACCGGAUU CUG AUUCCGGC AUUC CGAGG UUCG CCUCG AAU UACCCC AGCCA Найдено 4 спирали. Длина первой – 6 пар. Длина второй - 5 пар. Длина третьей – 8 пар. Длина четвертой – 3 пары.По результатам программы find_pair выявлено 4 спирали.
Strand I Strand II Helix --1 (0.005) B:.902_:[..G]G-----C[..C]:.971_:B (0.004) | --2 (0.007) B:.903_:[..G]G-----C[..C]:.970_:B (0.005) | --3 (0.009) B:.904_:[..G]G-----C[..C]:.969_:B (0.003) | --4 (0.003) B:.905_:[..G]G-----C[..C]:.968_:B (0.005) | --5 (0.006) B:.906_:[..U]U-----A[..A]:.967_:B (0.004) | --6 (0.004) B:.907_:[..A]Ax----U[..U]:.966_:B (0.002) | --7 (0.002) B:.949_:[..C]C-----G[..G]:.965_:B (0.003) | --8 (0.003) B:.950_:[..G]G-----C[..C]:.964_:B (0.003) | --9 (0.003) B:.951_:[..A]A-----U[..U]:.963_:B (0.002) | --10 (0.008) B:.952_:[..G]G-----C[..C]:.962_:B (0.003) | --11 (0.004) B:.953_:[..G]G----xC[..C]:.961_:B (0.004) | 12 (0.002) B:.954_:[..U]U-**-xA[..A]:.958_:B (0.004) | 13 (0.004) B:.955_:[..U]Ux**+xG[..G]:.918_:B (0.013) x --14 (0.002) B:.937_:[..A]A-*---U[..U]:.933_:B (0.004) | --15 (0.006) B:.938_:[..U]U-*---U[..U]:.932_:B (0.004) | --16 (0.003) B:.939_:[..U]U-----A[..A]:.931_:B (0.004) | --17 (0.006) B:.940_:[..C]C-*---G[..G]:.930_:B (0.010) | --18 (0.006) B:.941_:[..C]C-----G[..G]:.929_:B (0.003) | --19 (0.007) B:.942_:[..G]G-----C[..C]:.928_:B (0.003) | --20 (0.006) B:.943_:[..G]G-----C[..C]:.927_:B (0.004) | --21 (0.005) B:.944_:[..C]Cx*---A[..A]:.926_:B (0.009) | --22 (0.005) B:.910_:[..G]G-----C[..C]:.925_:B (0.003) | --23 (0.006) B:.911_:[..C]C-----G[..G]:.924_:B (0.008) | --24 (0.008) B:.912_:[..C]C----xG[..G]:.923_:B (0.005) | 25 (0.003) B:.913_:[..A]A-**+xA[..A]:.945_:B (0.005) | 26 (0.004) B:.914_:[..A]A-**-xU[..U]:.908_:B (0.009) | 27 (0.020) B:.915_:[..G]Gx**+xC[..C]:.948_:B (0.003) x 28 (0.008) B:.919_:[..G]G-----C[..C]:.956_:B (0.003) +
Их координаты даны в скрипте.
restrict rna and backbone define h1 902-907:B, 966-971:B define h2 949-953:B, 961-965:B define h3 937-944:B, 926-933:B define h4 910-912:B, 923-925:B select h1 color green select h2 color red select h3 color blue select h4 color orange zoom 150 select 903:B and *.P spacefill 200 label ---3` %r color orange select 976:B and *.P spacefill 200 label ---5` %r
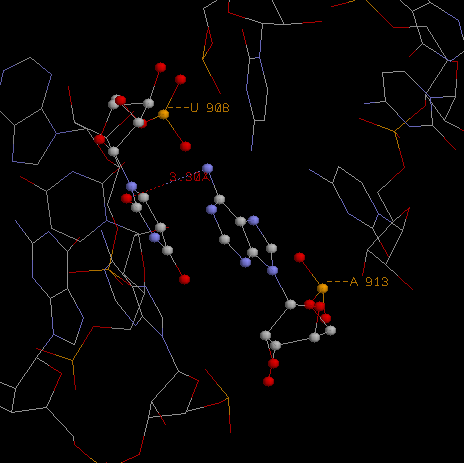
Пример неспирального стекинг взаимодействия U908-----A913:
 Информацию о не Уотсон-Криковских взаимодействиях и водородных связях можно узнать из результатов программы
analyze(1QTQ.out). Pick-distance в RasMol`е подтверждает данные.
Note: This structure contains 9[5] non-Watson-Crick base-pairs.
**************************************************************************
Detailed H-bond information: atom-name pair and length [ON]
1 G-----C [3] O6 - N4 2.64 N1 - N3 2.98 N2 - O2 3.19
2 G-----C [3] O6 - N4 2.93 N1 - N3 2.81 N2 - O2 2.56
3 G-----C [3] O6 - N4 2.96 N1 - N3 2.84 N2 - O2 2.71
4 G-----C [3] O6 - N4 2.95 N1 - N3 2.92 N2 - O2 2.85
5 U-----A [2] N3 - N1 2.92 O4 - N6 3.23
6 A-----U [2] N6 - O4 3.01 N1 - N3 2.86
7 C-----G [3] O2 - N2 2.65 N3 - N1 2.73 N4 - O6 2.71
8 G-----C [3] O6 - N4 2.97 N1 - N3 2.83 N2 - O2 2.64
9 A-----U [2] N6 - O4 3.07 N1 - N3 2.73
10 G-----C [2] O6 * N3 3.09 N2 - O2 3.00
11 G-----C [3] O6 - N4 3.20 N1 - N3 2.98 N2 - O2 2.62
12 U-**--A [2] O2 - N6 3.17 N3 - N7 3.05
13 U-**+-G [2] O4'* O6 3.64 O2 - N2 2.96
14 A-*---U [1] N6 - O2 2.76
15 U-*---U [1] O4 - N3 2.96
16 U-----A [2] N3 - N1 3.34 O4 - N6 3.21
17 C-*---G [3] O2'- N2 3.99 O2 - N1 2.87 N3 * O6 3.98
18 C-----G [3] O2 - N2 2.89 N3 - N1 2.89 N4 - O6 2.74
19 G-----C [3] O6 - N4 3.55 N1 - N3 3.21 N2 - O2 2.77
20 G-----C [3] O6 - N4 3.02 N1 - N3 2.90 N2 - O2 2.69
21 C-*---A [2] O2 * N1 3.27 N3 - N6 3.13
22 G-----C [3] O6 - N4 3.10 N1 - N3 2.94 N2 - O2 2.67
23 C-----G [3] O2 - N2 2.56 N3 - N1 2.82 N4 - O6 2.94
24 C-----G [3] O2 - N2 2.98 N3 - N1 3.00 N4 - O6 2.91
25 A-**+-A [1] N6 - N1 3.85
26 A-**--U [3] OP2* O4 3.62 N7 - N3 2.82 N6 - O2 2.75
27 G-**+-C [2] N1 - O2 3.02 N2 - N3 2.74
28 G-----C [3] O6 - N4 2.92 N1 - N3 2.90 N2 - O2 2.83
**************************************************************************
Примеры не Уотсон-Криковских взаимодействий:
Информацию о не Уотсон-Криковских взаимодействиях и водородных связях можно узнать из результатов программы
analyze(1QTQ.out). Pick-distance в RasMol`е подтверждает данные.
Note: This structure contains 9[5] non-Watson-Crick base-pairs.
**************************************************************************
Detailed H-bond information: atom-name pair and length [ON]
1 G-----C [3] O6 - N4 2.64 N1 - N3 2.98 N2 - O2 3.19
2 G-----C [3] O6 - N4 2.93 N1 - N3 2.81 N2 - O2 2.56
3 G-----C [3] O6 - N4 2.96 N1 - N3 2.84 N2 - O2 2.71
4 G-----C [3] O6 - N4 2.95 N1 - N3 2.92 N2 - O2 2.85
5 U-----A [2] N3 - N1 2.92 O4 - N6 3.23
6 A-----U [2] N6 - O4 3.01 N1 - N3 2.86
7 C-----G [3] O2 - N2 2.65 N3 - N1 2.73 N4 - O6 2.71
8 G-----C [3] O6 - N4 2.97 N1 - N3 2.83 N2 - O2 2.64
9 A-----U [2] N6 - O4 3.07 N1 - N3 2.73
10 G-----C [2] O6 * N3 3.09 N2 - O2 3.00
11 G-----C [3] O6 - N4 3.20 N1 - N3 2.98 N2 - O2 2.62
12 U-**--A [2] O2 - N6 3.17 N3 - N7 3.05
13 U-**+-G [2] O4'* O6 3.64 O2 - N2 2.96
14 A-*---U [1] N6 - O2 2.76
15 U-*---U [1] O4 - N3 2.96
16 U-----A [2] N3 - N1 3.34 O4 - N6 3.21
17 C-*---G [3] O2'- N2 3.99 O2 - N1 2.87 N3 * O6 3.98
18 C-----G [3] O2 - N2 2.89 N3 - N1 2.89 N4 - O6 2.74
19 G-----C [3] O6 - N4 3.55 N1 - N3 3.21 N2 - O2 2.77
20 G-----C [3] O6 - N4 3.02 N1 - N3 2.90 N2 - O2 2.69
21 C-*---A [2] O2 * N1 3.27 N3 - N6 3.13
22 G-----C [3] O6 - N4 3.10 N1 - N3 2.94 N2 - O2 2.67
23 C-----G [3] O2 - N2 2.56 N3 - N1 2.82 N4 - O6 2.94
24 C-----G [3] O2 - N2 2.98 N3 - N1 3.00 N4 - O6 2.91
25 A-**+-A [1] N6 - N1 3.85
26 A-**--U [3] OP2* O4 3.62 N7 - N3 2.82 N6 - O2 2.75
27 G-**+-C [2] N1 - O2 3.02 N2 - N3 2.74
28 G-----C [3] O6 - N4 2.92 N1 - N3 2.90 N2 - O2 2.83
**************************************************************************
Примеры не Уотсон-Криковских взаимодействий:
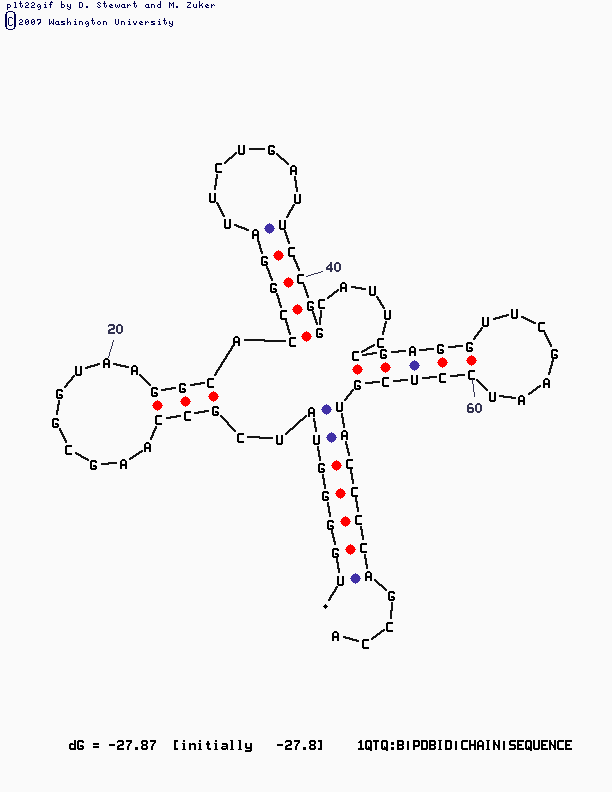
По результатам программы analyze данная структура тРНК является А-формой. Это подтверждается также визуально - при просмотре структуры с торца в RasMol видна полость, соответствующая структуре ДНК А-формы.Программа einverted выдает при настройках нижнего порога 21 один участок: SEQUENCE: Score 21: 7/7 (100%) matches, 0 gaps 1 tggggta 7 ||||||| 71 accccat 65 Уменьшая параметры gap match dismatch threshold почти до 0 получаем: SEQUENCE: Score 30: 25/25 (100%) matches, 20 gaps 1 tggggt------a-tcgccaag---cggtaag-gcaccgg-attc 33 |||||| | ||| || ||| || | |||| | || 71 accccatgctcctaagc--tt-ggagcc-tt-ac--ggcctt-ag 35
В первом случае участки соответствуют кусочкам первой спирали. Во втором случае с довольно плохой точностью программа указала правильное расположение спиралей. Результат не соответствует 3D структуре, вероятно, программа работает лучше, если структура тРНК имеет более длинную последовательность и меньшее искривление (более длинные спирали). На результат, возможно, влияют также не Уотсон-Криковские взаимодействия.- На мой взгляд, эта структура, полученная mfold с параметром P=15(3-ий рисунок), наиболее близка к 3D структуре
и данным анализа. Напоминает трилистник тРНК.
вернуться к 3 семестру