Занятие 2. Моделирование и реконструкция эволюции гена
Скобочная формула исходного дерева эволюции гена PHR_ECOLI следущая: (((А:90,B:90):5,C:70):30,((D:90,E:90):4,F:80):20);
Графическое изображение дерева приведено ниже:
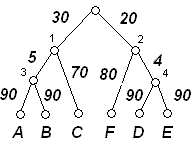
Также приведена таблица описывающая ветви исходного дерева как разбиение по листьям:
A B C D E F . . * * * * . . . * * * . . . * * .
С помощью программы msbar пакета EMBOSS были получены искуственные мутантные последовательности, соответствующие листьям и узлам исходного дерева.
Длина гена PHR_ECOLI = 1418 нп.
N = L*C/100, где
N - число мутаций;
L - длина гена;
С - число замен(длина ветви).
Значение N нужно для параметра -count пргораммы msbar. Ниже приведен текст скрипта, использовавшийся для получения мутантных последовательностей:
msbar 12.fasta 3C.fasta -point 4 -count 425 -auto msbar 3C.fasta AB.fasta -point 4 -count 71 -auto msbar 3C.fasta C.fasta -point 4 -count 993 -auto msbar AB.fasta A.fasta -point 4 -count 1276 -auto msbar AB.fasta B.fasta -point 4 -count 1276 -auto msbar 12.fasta 4F.fasta -point 4 -count 284 -auto msbar 4F.fasta DE.fasta -point 4 -count 57 -auto msbar 4F.fasta F.fasta -point 4 -count 1134 -auto msbar DE.fasta D.fasta -point 4 -count 1276 -auto msbar DE.fasta E.fasta -point 4 -count 1276 -autoЗдесь находятся полученные файлы. Все последовательности листьев были собраны в один файл. Файл был запущен на реконструкцию дерева разными алгоритмами.
Дерево полученное алгоритмом максимального правдоподобия
fdnaml ali.fasta -ttratio 1 -auto Выходной файл+--------------------B | | +----------------F | +-----------4 | | | +-------------------E 1--2 +---3 | | +-----------------D | | | +---------------C | +-------------------A
Дерево полученное алгоритмом Neighbor-joining
fdnadist ali.fasta -ttratio 1 -auto # высчитывает попарные расстояния между последовательностямиfneighbor ali.fdnadist -auto Выходной файл
+--------------------B ! ! +--------------C ! ! 3---4 +----------------D ! ! +-----1 ! +-------------2 +---------------------E ! ! ! +---------------F ! +-------------------A
Дерево полученное алгоритмом UPGMA
fdnadist ali.fasta -ttratio 1 -autofneighbor ali.fdnadist -treetype u -auto Выходной файл
+--------------------------------------A
+-2
+----------------3 +--------------------------------------C
! !
! +---------------------------------------B
--5
! +--------------------------------------D
! +-1
+---------------4 +--------------------------------------E
!
+----------------------------------------F
Сравнение эволюционных древ
A B C D E F Исходное Макс.пр. N-J UPGMA . . * * * * + + + - . . . * * * + + + + . . . * * . + + + + . * . * * * - - - +
Сравнение показало, что лишь UPGMA алгоритм ошибся с одной из ветвей дерева. Вероятно, ошибка в топологии произошла из-за принятой по умолчанию в UPGMA гипотезы "молекулярных часов" (сумма длин ветвей от корня до листа всегда одинакова для любого листа). Корень UPGMA определила верно, что приятно, так как это единственный из всех трех алгоритмов, который реконструирует корневые деревья.
вернуться к 3 семестру