HETNAM 2MG 2N-METHYLGUANOSINE-5'-MONOPHOSPHATE
HETNAM H2U 5,6-DIHYDROURIDINE-5'-MONOPHOSPHATE
HETNAM M2G N2-DIMETHYLGUANOSINE-5'-MONOPHOSPHATE
HETNAM OMC O2'-METHYLYCYTIDINE-5'-MONOPHOSPHATE
HETNAM OMG O2'-METHYLGUANOSINE-5'-MONOPHOSPHATE
HETNAM YG WYBUTOSINE
HETNAM PSU PSEUDOURIDINE-5'-MONOPHOSPHATE
HETNAM 5MC 5-METHYLCYTIDINE-5'-MONOPHOSPHATE
HETNAM 7MG 7N-METHYL-8-HYDROGUANOSINE-5'-MONOPHOSPHATE
HETNAM 5MU 5-METHYLURIDINE 5'-MONOPHOSPHATE
HETNAM 1MA 6-HYDRO-1-METHYLADENOSINE-5'-MONOPHOSPHATE
HETNAM MG MAGNESIUM ION
HETNAM SPM SPERMINE
Последовательность тРНК в формате FASTA.
>1EVV:A|PDBID|CHAIN|SEQUENCE
GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAXCUGGAGGUCCUGUGUXCGAUCCACAGAAUUCGCACCA
В pdb-файле, описывающем тРНК пронумерованы атомы, входящие в структуру аминокислотных остатков, в строке написано в какой остаток они входят и номер этого остатка, при чем вначале перечисляются все атомы, принадлежащие 1 остатку, потом 2 и т.д. по порядку. Остатки пронумерованы от 1 до 76 (вместе с модифицированными основаниями).
Было найдено 2 спирали, в каждой из которых по 29 оснований. На последовательности красным выделены а.о., входящие в разные спирали.
GCGGAUUUAGCUCAGUUGGAGAGCGCCAGACUGAAGAXCUGGAGGUCCUGUGUXCGAUCCACAGAAUUCGCACCA
На рисунке изображена цепь тРНК в остовной модели, найденные спирали покрашены в разные цвета, при атомах фосфора 5'-концевого и 3'-концевого нуклеотидов подписаны номера остатков. Чтобы получить такой рисунок, нужно использовать команды
Найдено 4 стекинг-взаимодействия между основаниями, два из них показано на рисунке слева(взаимодействия двух оснований и четырех), два - справа(каждое взамодействие между тремя основаниями).
Также были найдены водородные связи между основаниями, не сводящиеся к Уотсон-Криковскому спариванию комплементарных оснований
Спирали тРНК больше похожи на A-форму ДНК, т.к. в одном витке спирали 11 оснований, а если посмотреть с торца, то тРНК выглядит как баранка.
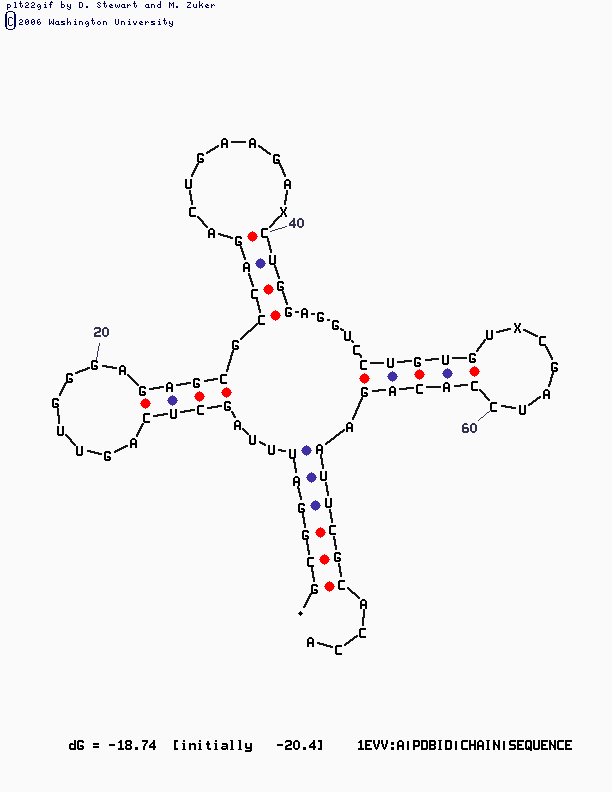
На рисунке показано наиболее реалистичное предсказание. Оно было получено на третьей попытке, когда параметр P был равен 15.
Анализ структуры тРНК программой find_pair
Исследование структуры с помощью программы RasMol
restrict none
restrict rna and not hetero
backbone 100
select 1-7:A
color red
select 49-55:A
color red
select 36:A
color red
select 38-44:A
color red
select 10-15:A
color red
select 19:A
color red
select 56:A
color green
select 48:A
color green
select 8:A
color green
select 22-33:A
color green
select 18:A
color green
select 58:A
color green
select 61-72:A
color green
select g1.p
label 1
select a76.p
label 76
Поиск возможных двуспиральных участков с помощью программы einverted
Было найдено два участка
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
12 tcagt 16
|||||
35 agtca 31
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
Судя 3D структуре, в тРНК 1evv есть 3 двуспиральных участка, к тому же участки длиннее. Вероятно программа неправильно определила участки из-за "неУотсон-Криковских" взаимодействий, а также из-за гетероатомов.
Предсказание структуры тРНК с помощью программы mfold
©Надя Шашина