Отчет по практикуму 15
Подготовка чтений
Я анализировал секвенирование Buchnera aphidicola с кодом доступа SRR4240359 в базе ENA.
Для анализа я сначала скачал чтения в формате .fastq при помощи команды wget:
wget 'ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq
.gz'Затем я создал файл в формате fasta, содержащий последовательности адаптеров, которые необходимо вырезать из чтений:
cat /mnt/scratch/NGS/adapters/* > adapters.fastaДалее я удалил из чтений адаптеры, которые лежат в файле adapters.fasta. При помощи Trimmomatic:
java -jar /usr/share/java/trimmomatic.jar SE -threads 10 -phred33
-trimlog trim.log SRR4240359.fastq.gz SRR4240359_trimmed_adapt.fastq.gz
ILLUMINACLIP:adapters.fasta:2:7:7После работы программы количество чтений сократилось с 13557938 до 13502066. То есть 55872 (0.41%) чтений было удалено.
Далее я удалил с 3'-конца чтений нуклеотиды, которые имели качество меньше 20, оставляя при этом минимальную длину в 32 нуклеотида:
java -jar /usr/share/java/trimmomatic.jar SE -threads 10 -phred33 -trimlog
trim2.log SRR4240359_trimmed_adapt.fastq.gz SRR4240359_full_trimmed.fastq.gz
TRAILING:20 MINLEN:32
Из 13502066 чтений осталось 12184080. Было отброшено 1317986 чтений (9.76%).
Исходный файл (SRR4240359.fastq.gz) имел размер 445M, после обрезки адаптеров размер сократился до 443M (SRR4240359_trimmed_adapt.fastq.gz), а после обрезания 3'-конца упал до 385M (SRR4240359_full_trimmed.fastq.gz).
Формирование k-меров и сборка
Я создал папку Assembly_SRR4240359 с k-мерами длиной 31 из обработанных чтений при помощи программы velveth:
velveth Assembly_SRR4240359 31 -short -fastq.gz SRR4240359_full_trimmed.fastq
.gzСборка при помощи velvetg:
velvetg Assembly_SRR4240359N50 = 70607
Длины трёх самых длинных контигов - 125674, 108447 и 71403. Им соответствуют покрытия 44.55, 42.01 и 39.41.
Анализ контигов
Для картирования я использовал megablast с функцией "align two or more sequences" на сайте NCBI с параметром word size = 48 (иначе количество участков картирования превышало 15), остальные параметры были выставлены по умолчанию.
Контиг длины 125674 картировался на геном по 6 участкам: в него входит точка начала секвенирования хромосомы Buchnera aphidicola.
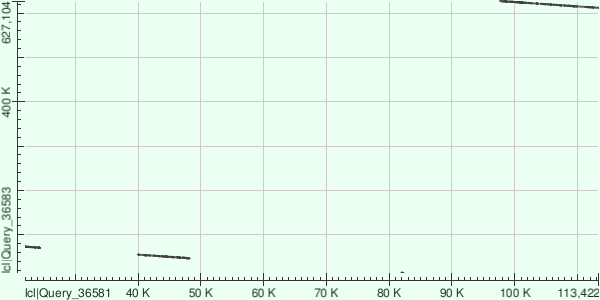
| Фрагмент генома | Identities | Gaps |
| 13994 to 14465 | 393/478(82%) | 9/478(1%) |
| 47158 to 55420 | 6440/8436(76%) | 301/8436(3%) |
| 70970 to 73310 | 1774/2411(74%) | 102/2411(4%) |
| 611633 to 613671 | 1625/2086(78%) | 66/2086(3%) |
| 613658 to 620926 | 5845/7379(79%) | 184/7379(2%) |
| 621055 to 627104 | 4676/6171(76%) | 244/6171(3%) |
Контиг длины 108447 картировался на геном по 10 участкам:
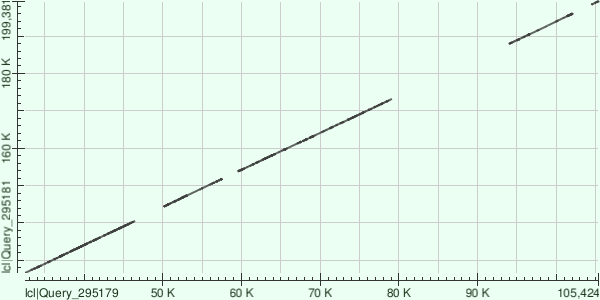
| Фрагмент генома | Identities | Gaps |
| 126623 to 127815 | 1004/1199(84%) | 11/1199(0%) |
| 127825 to 140555 | 9751/13010(75%) | 548/13010(4%) |
| 144368 to 151796 | 5859/7536(78%) | 243/7536(3%) |
| 153752 to 161738 | 6355/8168(78%) | 264/8168(3%) |
| 161898 to 166752 | 3911/4914(80%) | 112/4914(2%) |
| 166750 to 173180 | 4967/6517(76%) | 159/6517(2%) |
| 187938 to 192665 | 3840/4801(80%) | 99/4801(2%) |
| 192777 to 193984 | 985/1209(81%) | 4/1209(0%) |
| 194042 to 196061 | 1640/2070(79%) | 78/2070(3%) |
| 198467 to 199381 | 724/922(79%) | 17/922(1%) |
Контиг длины 71403 картировался на геном по 11 участкам: он был секвенирован с цепи, комплементарной цепи, с которой был секвенирован геном.
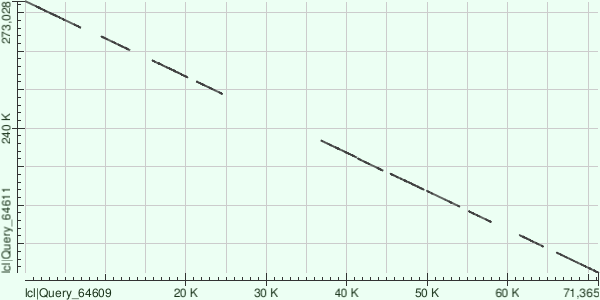
| Фрагмент генома | Identities | Gaps |
| 202390 to 207661 | 4183/5329(78%) | 137/5329(2%) |
| 209294 to 212243 | 2302/3007(77%) | 104/3007(3%) |
| 215717 to 218384 | 2145/2713(79%) | 72/2713(2%) |
| 219625 to 223720 | 3342/4130(81%) | 61/4130(1%) |
| 224057 to 228137 | 3218/4178(77%) | 163/4178(3%) |
| 228944 to 232057 | 2499/3165(79%) | 95/3165(3%) |
| 232358 to 236859 | 3468/4583(76%) | 134/4583(2%) |
| 248967 to 252161 | 2523/3245(78%) | 92/3245(2%) |
| 253223 to 257546 | 3245/4421(73%) | 195/4421(4%) |
| 260224 to 263784 | 2788/3617(77%) | 101/3617(2%) |
| 266073 to 273028 | 5664/7060(80%) | 197/7060(2%) |
В целом хуже всего картировался контиг длины 71403, лучше всего - контиг длины 125674.