Отчет по практикуму 8
Задание 1: Разные варианты BLAST для короткого контига
a) Выбор участка контига
Я выбрал контиг с идентификатором GenBank DS483646.1. Его длина 14098 пар оснований, на нём находится единственный ген CG41561, расположенный на комплементарной цепи 3 хромосомы, состоящий из 4 экзонов и 3 интронов. Это CDS, которая кодирует неохарактеризованный белок.
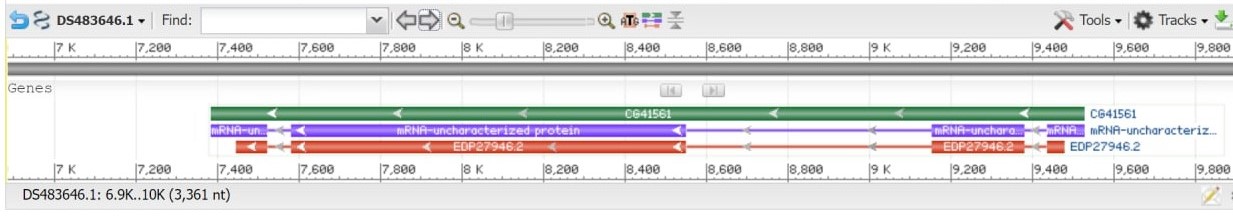
Рис. 1.Схема расположения генов в контиге DS483646.1. Зеленым цветом показан участок ДНК, кодирующий ген. Фиолетовыми цветом показана мРНК данного гена: на ней прямоугольниками показаны экзоны, а соединяющими их линиями - интроны. Красным цветом показана белковая последовательность гена: на ней также прямоугольниками обозначены экзоны, а соединяющими их линиями - интроны. Серая стрелка показывает направление прочтения последовательности (справа налево т.к. ген расположен на комплементарной цепи). Длина белковой последовательности вместе с интронами меньше, чем длина мРНК с интронами из-за 5'- и 3'-нетранслируемых областей.
Согласно таблице особенностей, CDS соответствует участок контига DS483646.1 complement(join(7445..7523,7582..8551,9154..9380, 9435..9478)). Этот участок длиной 1320 пар оснований и был выбран для дальнейшего анализа (скачать файл с участком контига в формате FASTA можно по ссылке).
б) Применение BLAST
Для исключения я выбрал только вид Drosophila melanogaster, т. к. поиск при помощи blastn с исключением более старших таксонов требовал изменения длины слова до 11, чтобы обнаружить хоть какие-то результаты, и обнаруживал выравнивания с покрытием не более 4%.
- Поиск при помощи blastn с параметрами по умолчанию обнаружил 3 результата: предсказанная мРНК предполагаемого неохарактеризованного белка Drosophila sechellia и 2 варианта предсказанного транскрипта мРНК белка подобного аспарагин-богатому (англ. asparagine-rich protein-like) Drosophila mauritiana.
- Поиск при помощи megablast обнаружил те же результаты (процент идентичности 89,76-89,39%).
- Поиск при помощи blastx (по базе refseq_proteins) обнаружил 76 результатов. Многие находки оказались предсказанными или неохарактеризованными белками. Большинство принадлежало видам рода Drosophila, но некоторые - другим родам двукрылых (Zeugodacus, Bactrocera, Lucilia, Ceratitis, Musca)
- Поиск при помощи tblastx с параметрами по умолчанию не обнаружил никаких результатов. При попытке изменить параметры поиск либо зависает и не начинается вовсе, либо сильно затягивается (моего терпения хватило на 30 минут, но поиск так и не закончился, другие blast в тот же временной промежуток искали не больше 5 минут).
в) Выводы
Получается, что blastn следует применять, когда необходимо найти гомологов нуклеотидной последовательности, не кодирующей белков. Например, при поиске гомологичных рРНК, малых ядерных РНК или регуляторных последовательностей ДНК. Megablast стоит использовать, когда есть уверенность, что очень похожая нуклеотидная последовательность есть в базе данных. Например, если нужно определить, сколько повторов общего предшественника рРНК в геноме конкретного организма на определенном этапе развития. Blastx полезен, если небходимо найти гомологов белок-кодирующей последовательности .Например, если хочется изучить, какова частота разных синонимичных кодонов в определенной позиции какого нибудь-белка в некоторой таксономической группе. Tblastx можно применить, если остальные виды blast не дали результатов и есть подозрения, что гомологи исследуемой белок-кодирующей нуклеотидной последовательности не аннотированы или аннотированы неправильно. Например, если в ходе исследований обнаружилось, что некий белок имеет необычную кодировку аминокислот и, вероятно, последовательности его гомологов в известных геномах были автоматически транслированы неверно.
Задание 2: Нахождение генов рРНК по далекому гомологу
Выданные мне последовательности рРНК являются компонентами бактериальной рибосомы (70S): 18S рРНК входит в состав малой субъединицы и комплементарна сайту посадки рибосомы на мРНК (последовательности Шайна-Дальгарно), а 23S рРНК - в состав большой субъединицы и катализирует пептидилтрансферазную реакцию (перенос аминокислоты с тРНК на растущую пептидную цепь).
Для того, чтобы найти гомологи рРНК E. coli в моей геномной сборке Drosophila melanogaster я создал базу данных для локального blast, проиндексировав сборку консольной командой на сервере kodomo:
makeblastdb -in GCF_000001215.4_Release_6_plus_ISO1_MT_genomic.fna -dbtype nuclДалее я применил алгоритм blastn для того, чтобы найти гомологи отдельно 16S рРНК и 23S рРНК из E. coli (результаты доступны по ссылкам: blast16S.out blast23S.out). Поскольку с рРНК не синтезируются белки, ее гомологов бессмысленно искать при помощи blastx и tblastx. Гомологи эволюционно далеки, поэтому использовать megablast представляется также нецелесообразным. Параметры по умолчанию я решил не менять, т. к. в геноме Drosophila melanogaster можно предполагать наличие достаточно близких для обнаружения гомологов.
blastn -task blastn -query 16S.txt -db GCF_000001215.4_Release_6_plus_ISO1_MT_genomic.fna
-out blast16S.out -outfmt 7blastn -task blastn -query 23S.txt -db GCF_000001215.4_Release_6_plus_ISO1_MT_genomic.fna
-out blast23S.out -outfmt 716S рРНК выровнялась, согласно аннотации, с участками половых хромосом, кодирующими 18S рРНК (последовательности, кодирующие рРНК у эукариот организованы в виде повторов и результаты были в участках этих повторов, кодирующих 18S рРНК), последним результатом оказалось выравнивание с последовательностью экзона в гене на 2 хромосоме, кодирующем неохарактеризованный белок. 18S рРНК входит в состав малой субъединицы эукариотических рибосом и действительно считается гомологом 16S рРНК бактерий. То, что на половых хромосомах кодируются рРНК показалось мне странным, но у Drosophila melanogaster эти гены и правда локализованы на хромосомах X и Y, причем экспрессируются у особей мужского пола в основном с Y хромосомы [1].
23S рРНК, согласно аннотации, выровнялась с участками половых хромосом, кодирующими 28S рРНК, а также с геном кадгерина 86C, локализованного на третьей хромосоме. Кроме того, в конце списка результатов были неаннотированные участки 3 хромсомы. 28S рРНК является основной РНК большой субъединицы эукариотических рибосом. К моему удивлению, хотя сборка содержала митохондриальный геном, результатов из него обнаружено не было. Таким образом, для каждой рРНК нашлось по одному гомологу (если считать множественные копии последовательности рРНК одним гомологом).