Отчет по практикуму 4
Составление списка гомологичных белков, включающих паралоги
Для поиска гомологов среди протеомов выбранных бактерий, я скачал данные протеомы и проиндексировал, затем произвел поиск локальной версией blastp при помощи следующих команд (пример для ACIC1):
makeblastdb -in /P/y21/term4/Proteomes/ACIC1.fasta -dbtype prot -out db.fasta
blastp -query CLPX_ECOLI.fasta -db db.fasta -out ./blastp/ACIC1.fasta -evalue 0.001
Реконструкция и визуализация
Для реконструкции филогении была использована программа FastME на сервере NG phylogeny с параметрами:
"Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", количество бутстреп реплик 100.
Для выравнивания была использована программа MAFFT. Укоренение в среднюю точку произведено при помощи онлай-сервиса iTOL.
Этот же сервис был использован для получения визуализаций, представленных ниже.
Полученное дерево в формате newick доступно по ссылке: tree.
Примеры ортологов: RUVB_ACIC1 и RUVB_BIFLO, CLPB_BIFLO и CLPB_CORDI, CLPX_STRAW и QU47MU4_THEFY.
Примеры паралогов: CLPX_BIFLO и Q8G871_BIFLO, RUVB_BIFLO и Q8G852_BIFLO, FTSH_ACIC1 и A0LW31_ACIC1.
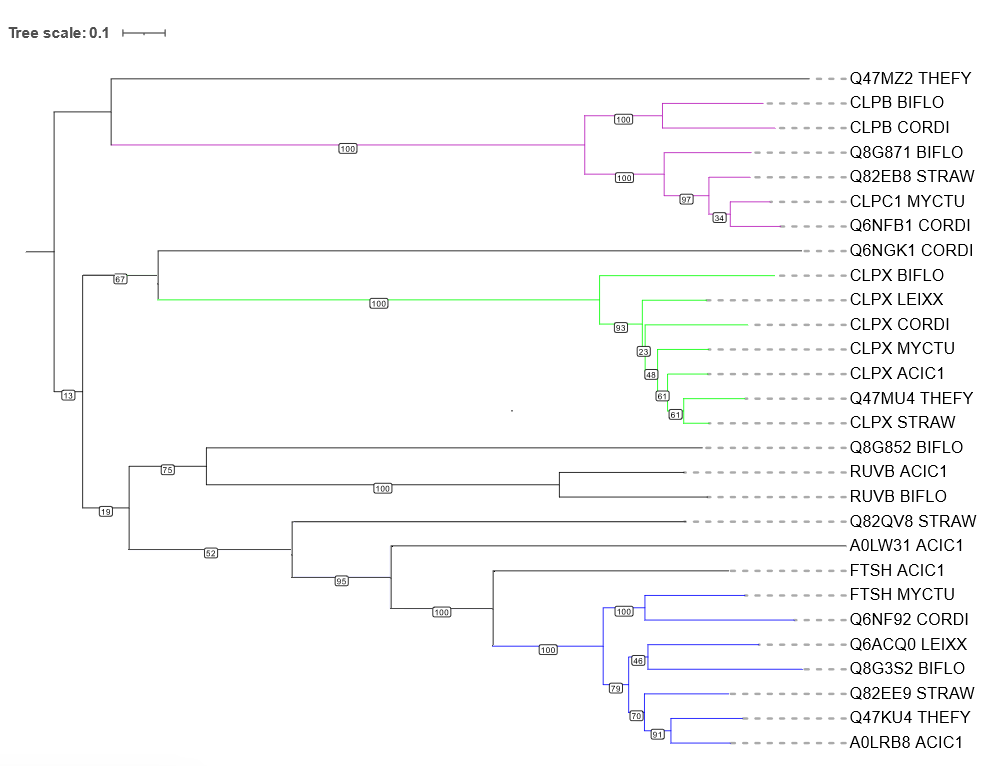
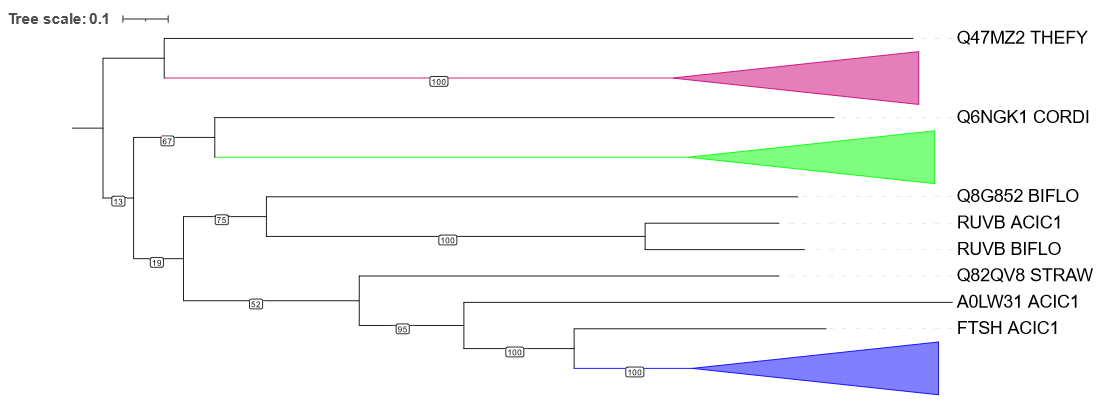
Клада, покрашенная на рис. 2 зеленым, содержит белки всех выбранных бактерий. Топология клады внутри группы {STRAW, THEFY, ACIC1} совпадает с правильной и характеризуется высокой bootstrap-поддержкой, в то же время топология внутри группы {MYCTU, CORDI, LEIXX, BIFLO} не соответствует филогении организмов, поддержка ветвей внутри нее низка.
Клада, покрашенная на рис. 2 синим, тоже содержит белки всех выбранных бактерий. Топология внутри нее соответствует филогении организмов, bootstrap-поддержка ветвей внутри нее также довольно высока.
Клада, покрашенная на рис. 2 розовым, содержит 2 белка BIFLO, 2 белка CORDI, по одному белку STRAW, MYCTU и THEFY. Топология ветвей внутри данной клады не совпадает с филогенией организмов, однако поддержка многих ветвей очень велика.