Хромосома: вторая.
| Команда | Что делает |
|---|---|
| fastqc chr2.fastq | Анализ качества секвенирования, выдает zip-архив и html-отчет |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr2.fastq chr2_trimmed.fastq TRAILING:20 MINLEN:50 | Обрезка с конца чтений нуклеотидов с качеством меньше 20 и отбрасывание чтений короче 50, выдает файл chr2_trimmed.fastq |
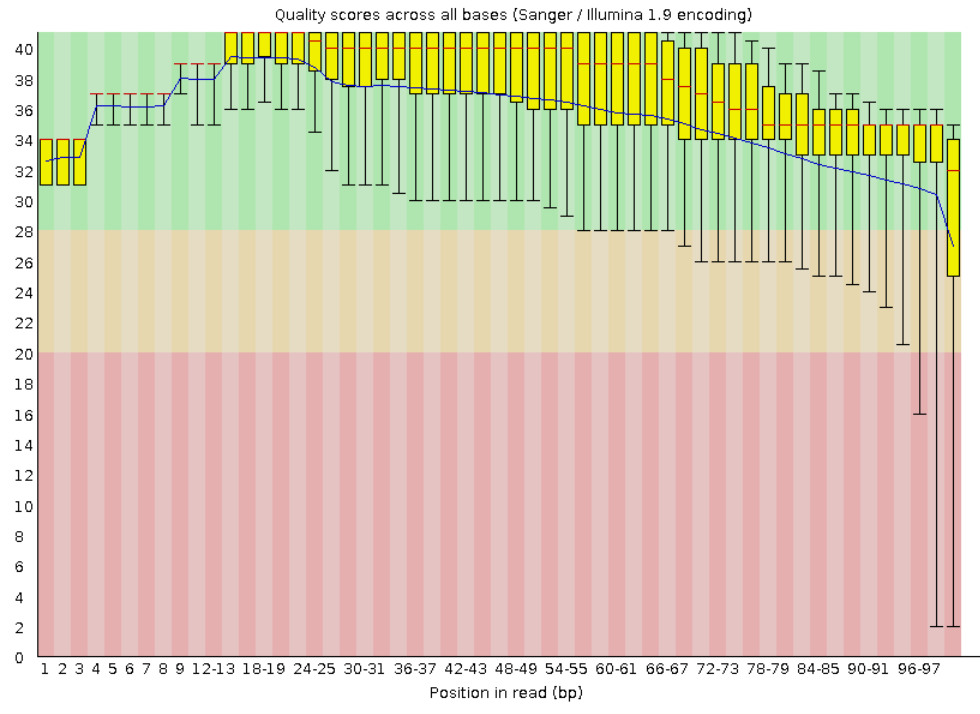
Изображения из отчета fastqc
До trimmomatic
После trimmomatic
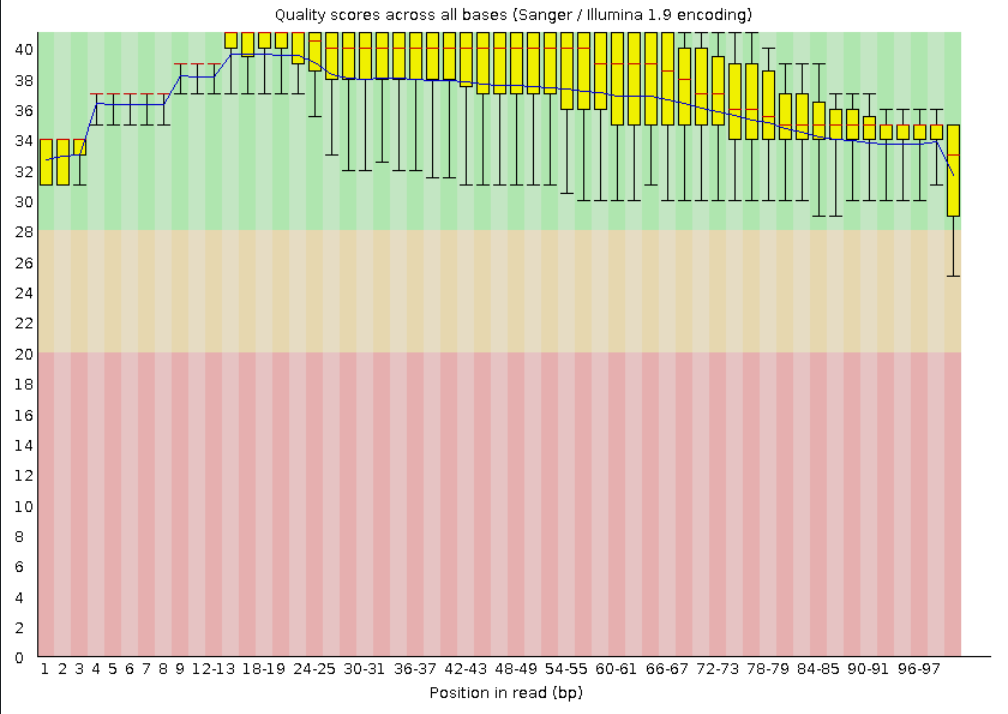
После триммирования от 10410 осталось 10191 ридов. Я считаю, что в данном случае процедура триммирования оправдана, т к в процентном соотношении удалено было немного ридов, но качество в среднем значительно улучшилось.
| Команда | Что делает |
|---|---|
| PATH=$PATH:/home/students/y06/anastaisha_w/hisat2-2.0.5 | Позволяет bash искать исполняемые файлы в указанной директории |
| hisat2-build chr2.fasta chr2_ref | Инсирует референсную последовательность, выдает ht2-файлы |
| hisat2 -x chr2_ref -U chr2_trimmed.fastq aligned.sam --no-spliced-alignment --no-softclip 2> aligned_pl.log | Выравнивает референсную последовательность и прочтения, также выводит log-файл |
| samtools view aligned.sam -bo aligned.bam | Переводит выравнивание в бинарный формат |
| samtools sort aligned.bam aligned_sorted | Сортирует выравнивание |
| samtools index aligned_sorted.bam | Индексирует выравнивание |
Вывод hisat2 :
10191 reads; of these:
10191 (100.00%) were unpaired; of these:
47 (0.46%) aligned 0 times
10140 (99.50%) aligned exactly 1 time
4 (0.04%) aligned >1 times
99.54% overall alignment rate
Таким образом, 99,54% чтений было картировано на геном, из них 99,50% (10140) было картировано один раз и 0,04% (4) более одного раза.
Почти все риды были однозначно картированы на геном, из этого можно сделать вывод о высоком качестве картирования.
| Команда | Что делает |
|---|---|
| samtools mpileup -uf chr2.fasta -o chr2_snp.bcf aligned_sorted.bam | Создает файл с полиморфизмами в формате .bcf |
| bcftools call -cv chr2_snp.bcf -o chr2_snp.vcf | Переводит bcf в vcf |
| Координата | Тип полиморфизма | Референс/чтение | Глубина покрытия (в этом месте) | Качество чтения (в этом месте) |
|---|---|---|---|---|
| 55516323 | Замена | G - C | 112 | 225.009 |
| 55523309 | Делеция | CAAAAAAAAAAA - CAAAAAAAAAAAAA | Не указана | 32.4684 |
| 238408404 | Замена | G - T | 1 | 7.79993 |
Всего 79 полиморфизмов, из них 7 инделей и 72 снп. Информация о трех полиморфизмах представлена в таблице выше. Для инделей, соответственно, глубина покрытия не указывается.
| Команда | Что делает |
|---|---|
| convert2annovar.pl -format vcf4 chr2_snp.vcf > snp.avinput | Переводит .vcf в annovar, выводит snp.avinput |
| annotate_variation.pl -filter -out snp138_filtered -build hg19 -dbtype snp138 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по dbSNP |
| annotate_variation.pl -out refgen -build hg19 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по RefGene |
| annotate_variation.pl -filter -out 1000g -build hg19 -dbtype 1000g2014oct_all snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по 1000 Genomes |
| annotate_variation.pl -regionanno -out gwas -build hg19 -dbtype gwasCatalog snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по GWAS |
| annotate_variation.pl -filter -out clinvar -build hg19 -dbtype clinvar_20150629 snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по ClinVar |
Качество и покрытие полиморфизмов:
| Медиана | Среднее | |
|---|---|---|
| Качество | 31,76 | 70,87 |
| Глубина | 3 | 15,76 |
Программа convert2annovar.pl показала, что замен 50 являются транзициями, а 22 трансверсиями.
Из refgene.variant_function видим, что снп были разделены на категории:
exonic (6)
intronic (57)
ncRNA_exonic (1)
UTR3 (6)
UTR5 (2)
Гены, в которые попали SNP: CCCDC88A, ATG16L1, MLPH. Из refgene.exonic_variant_function видим, что из 6 замен, которые попали в экзоны,
колько одна синонимечная (в гене MLPH).
Всего 67 снп имеют rs (замены, которые есть в базе snp).
Была посчитана средняя частота найденных snp по аннотации по базе 1000 Genomes (37.72%).
Из клинической аннотации снп: одна из замен может вызвать Inflammatory Bowel Disease (воспаление кишнчника), аннотирована в clinvar.
Замены, аннотированные в Gwas: два полиморфизма ассоциируются с болезнью Крона, один - с раком простаты, еще один, не совсем понятно с чем,
так как в графе обозначен как "Height".