Определение функции продукта и таксономии нуклеотидной последовательности
После анализа хроматограмм в практикуме 6 была получена нуклеотидная последовательность. Стоит сказать, что в ней есть один нуклеотид, который я так и не смогла определить (был только в одной хроматограмме и очень плохо). С ней был запущен blastn.
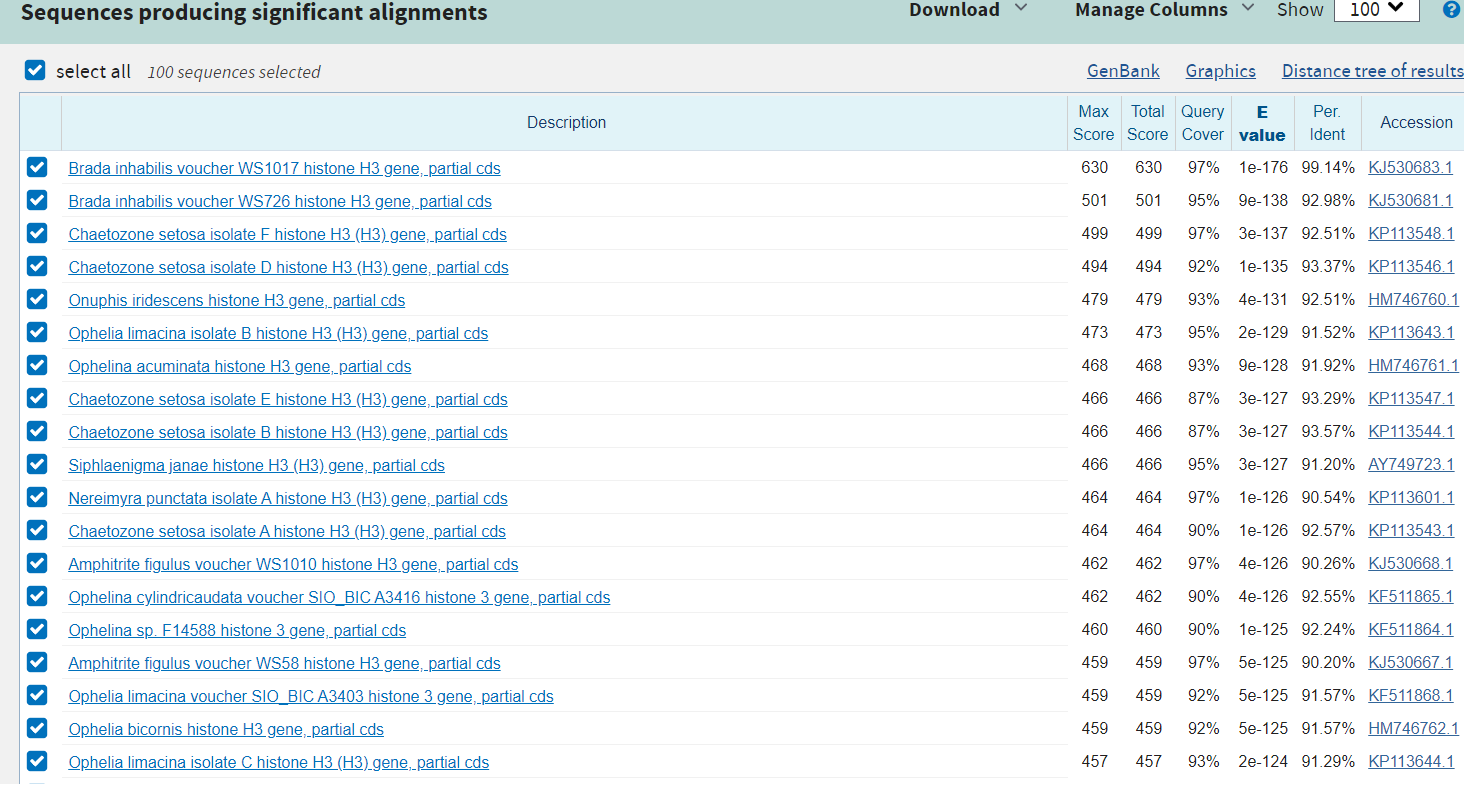
Было получено 100 результатов с per ident от 99,14 до 89,44. Интересно, что
организмы, из которых были получены последовательности, оказались достаточно далекими друг от друга.
Но также было понятно, что последовательность кодирует белок histone h3. Убедиться в этом
можно, посмотрев на картинки выше.
Histone H3 - белок, характерный для всех эукариот и обладающий высокой консервативностью.
Благодаря вкладке Taxonomy и сайту marinespecies
удалось немного разобраться в таксономии организмов из 6 самых блихзких результатов.
Это были Brada inhabilis, Chaetozone setosa Malmgren,
Onuphis iridescens и Ophelia limacina.
Оказалось, что родство этих животных заканчивается на классе - все они относятся к полихетам.
Но при этом их нуклеотидные последовательность очень похожи (видимо, как раз из-за высокой
консервативности белка).
Score Expect Identities Gaps Strand
630 bits(341) 1e-176 344/347(99%) 0/347(0%) Plus/Plus
Query 3 ACCGGAGGCAAGGCCCCCAGGAAGCAGCTGGCCACCAAGGCAGCTCGCAAGAGTGCCCCA 62
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 1 ACCGGAGGCAAGGCCCCCAGGAAGCAGCTGGCCACCAAGGCAGCTCGCAAGAGTGCCCCA 60
Query 63 GCAACTGGTGGAGTGAAGAAGCCCCATCGTTACAGGCCTGGCACAGTTGCTCTGCGTGAG 122
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 61 GCAACTGGTGGAGTGAAGAAGCCCCATCGTTACAGGCCTGGCACAGTTGCTCTGCGTGAG 120
Query 123 ATCCGTCGTTACCAGAAGAGCACAGAGCTGCTGATCCGCAAGCTGCCATTCCAGCGTCTG 182
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 121 ATCCGTCGTTACCAGAAGAGCACAGAGCTGCTGATCCGCAAGCTGCCATTCCAGCGTCTG 180
Query 183 GTGCGTGAGATCGCCCAGGACTTCAAGACTGACCTGCGCTTCCAGAGCTCTGCTGTCATG 242
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 181 GTGCGTGAGATCGCCCAGGACTTCAAGACTGACCTGCGCTTCCAGAGCTCTGCTGTCATG 240
Query 243 GCTTTGCAGGAGGCTAGCGAGGCTTACCTGGTTGGCCTGTTCGAGGACACCAACTTGTGC 302
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct 241 GCTTTGCAGGAGGCTAGCGAGGCTTACCTGGTTGGCCTGTTCGAGGACACCAACTTGTGC 300
Query 303 GCCATCCACGCCAAGCGTGTCACTATTATGCCNAAGGATATGTCATA 349
||||||||||||||||||||||| || ||||| ||||||||||||||
Sbjct 301 GCCATCCACGCCAAGCGTGTCACYATYATGCCTAAGGATATGTCATA 347
Можно посмотреть на выравнивание с последовательностью с самым высоким процентом идентичности, она принадлежит Brada inhabilis. Несовпадение всего в трех нуклеотидах, но и в них все не так уж и плохо, потому что Y это С или Т. Несовпадение в других выравниваниях несколько выше.
Таким образом, я считаю, что это однозначно histone H3 какой-то полихеты, скорее всего Brada inhabilis.
Сравнение разных бластов
1.Cравнение бластов последовательности histone h3.
Пробуя разные варианты ограничения по таксономии и E-value, я пришла к выводу, что самым адекватным является поиск:

| megablast | blastn default | blastn sensitive | |
| Длина слова | 28 | 11 | 7 |
| Match/mismatch | 1,-2 | 2,-3 | 1,-1 |
| Найденных последовательностей | 36 | 44 | 44 |
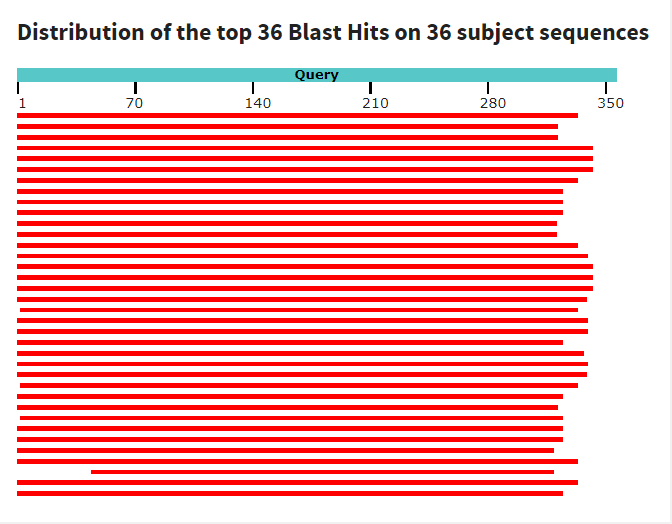
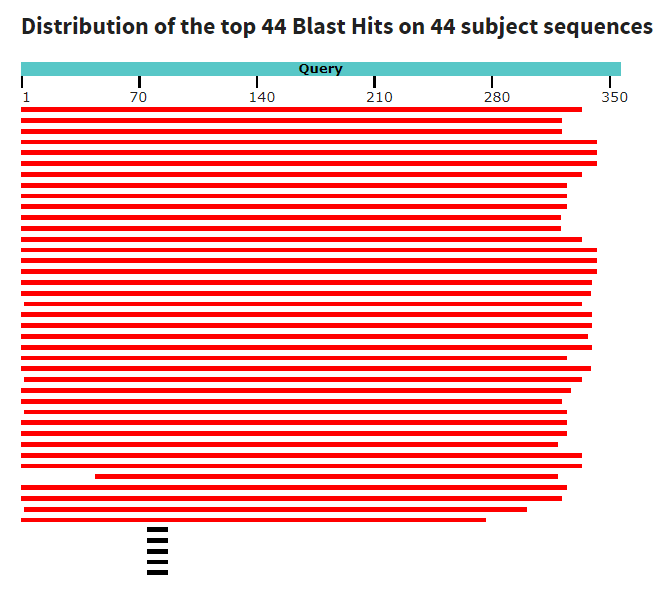
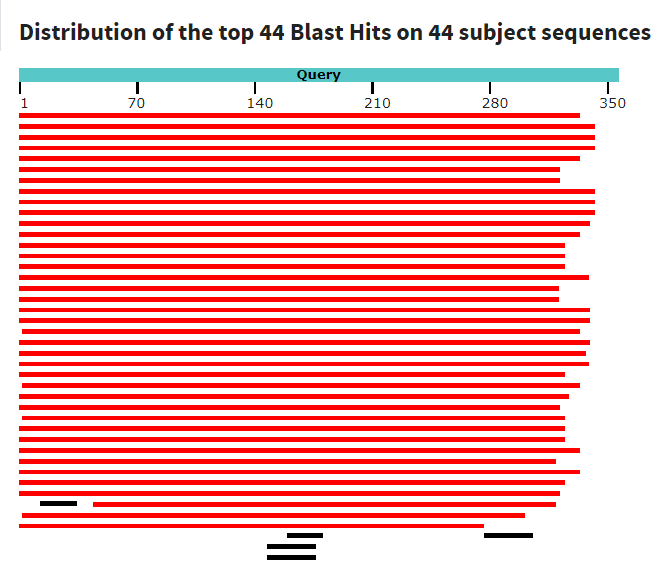
Изображения в порядке, как в таблице. Можно заметить, что blastn, как дефолтный, так и чувствительный, оказался для поиска несколько лучше, чем megablast, потому что с помощью него были найдены несколько очень похожих на исходную последовательностей, которые megablast попросту не обнаружил. Но зато blastn за счет маленькой длины слова выдал пару явно мусорных последовательностей, которые легко откидываются установкой E-value.
2.Сравнение бластов Ecolivirus, которая была получена в прошлом практикуме.
| megablast | blastn default | blastn sensitive | |
| Длина слова | 28 | 11 | 7 |
| Match/mismatch | 1,-2 | 2,-3 | 1,-1 |
| Найденных последовательностей | 11 | 45 | 62 |
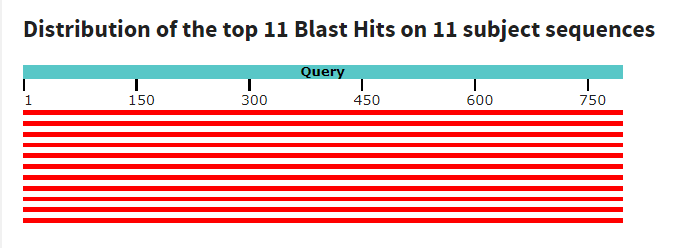

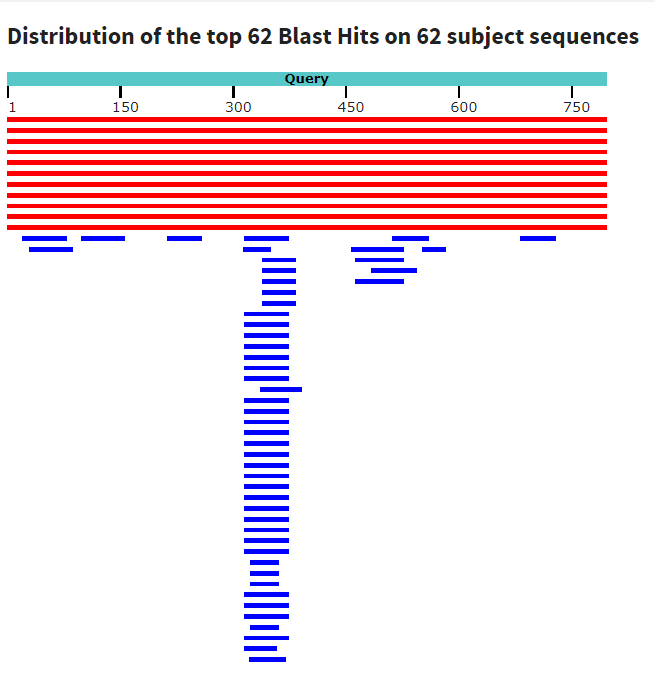
Все три программы нашли одинаковое количество очень похожих друг на друга последовательностей, но blastn нашел еще и последовательности, которые имеют короткий идентичный участок/участки. Можно предположить, что это участки высокой консервативности. При этом чувствительный blastn справился с этой задачей лучше.
Наличие гомологов трех белков в неаннотированном геноме
Искали гомологичные белки в Amoeboaphelidium protococcarum.
H31_HUMAN. Выходной файл.
Это первая форма histine h3. Как я уже писала выше, это достаточно консервативный белок для всех эукариот, и у данного организма тоже есть
очень похожая последовательность. Их Identity 93%, из чего можно сделать вывод, что они гомологичны.
AT1A1_HUMAN. Выходной файл
Альфа 1 субъединица Na+/K+ ATPase. Identity очень низкая - около 40%. Но если посмотеть повнимательнее, то можно заметить, что имеются
короткие идентичные участки (5 участков по 10-15 нуклеотидов каждый). На юнипрот написано, что этот белок должен
выполнять каталитическую функцию (
не только структурную), поэтому можно предположить, что это гомологичные домены.
CDC37_HUMAN. Выходной файл
Хотела поискать какой-нибудь шаперон. Видимо, этого конкретного нет.
Поиск гена белка в контиге
Была взята неаннотированная сборка Amoeboaphelidium protococcarum, которая лежит на кодомо. После н-ного перебора контигов был выбран unplaced-5.
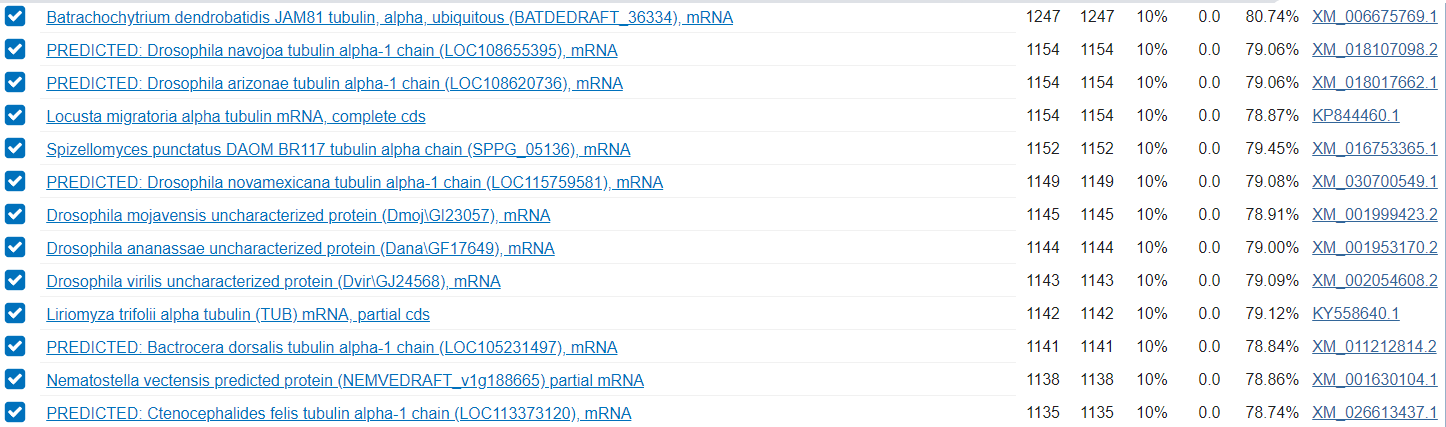
Этот контиг содержит ген, кодирующий альфа цепь тубулина. Ура.