АС сборки RefSeq: GCF_002900385.1
Cтраницa из базы NCBI Datasets Genome
АС сборки INSDC: GCA_002900385.1
Поисковый запрос по UniProt Proteomes: (genome_assembly:GCA_002900385.1)
Протеом избыточный в пользу UP001180515.
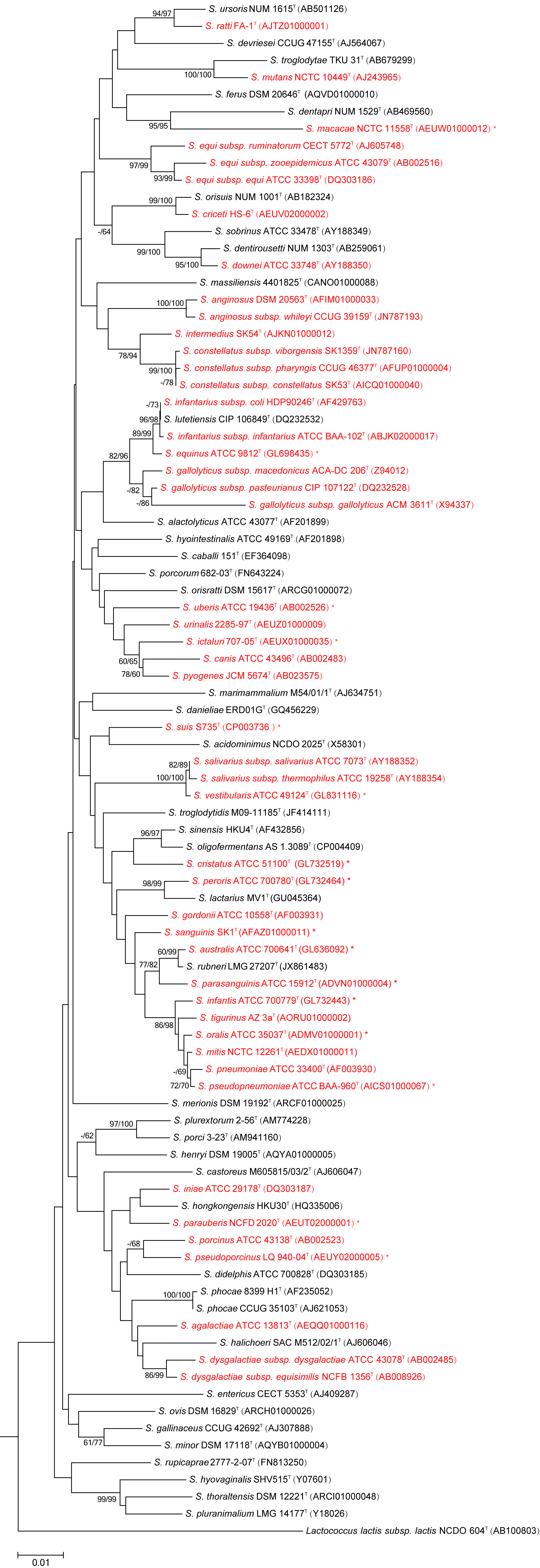
Для начала я ввела в поиск на сайте NCBI Taxonomy название моей бактерии (Streptococcus parauberis). Покликав по названиям нашла TaxID: 1348. В расширенном поиске UniProt ввела это TaxID, а также добавила критерий поиска Proteom Type: Reference. Теперь в поисковой строке стало находиться что-то такое: (taxonomy_id:1348) AND (proteome_type:1). Но, к сожалению, ничего не было найдено. Поэтому я выбрала родительский таксон своей бактерии, а именно Streptococcus. Его TaxID: 1301. Провела такой же поиск на UniProt с учетом нового ID, получила 41 результат. Так как результатов довольно много, как мне кажется, будет сложно вручную найти более близкий референсный протеом. Поэтому я нашла статью с филогинетическим древом рода Streptococcus.

По схеме видно, что ближайший вид для моей S.parauberis с учетом результатов поиска на UniProt это S.henryi, тк они расположены близко и на одном уровне. Значит, беру ее протеом UP000182508.
Команда для скачивания протеома: wget 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=(proteome:UP 000182508)' -O UP000182508.swiss.gz
Для поиска ферментов на сайте я решила ввести 2 разных запроса и сравнить полученный результат:
1. (proteome:UP000182508) AND ((keyword:KW-0560) OR (keyword:KW-0808) OR (keyword:KW-0378) OR (keyword:KW-0456) OR (keyword:KW-0413) OR (keyword:KW-0436)), где под ключевыми словами подразумеваются типы ферментов
2. (proteome:UP000182508) AND ( (go:0003824) OR (ec:*)), где go:0003824 обозначает каталитическую активность.
В первом случае я получила 794 результата, во втором - 1090. Разница значительная. Я думаю, что это связано с тем, что UniProtKB является курируемой базой данных. В связи с этим могло произойти так, что куратор не ко всем белкам с каталитической активностью присвоил ключевые слова с отсылкой на тип фермента. Поэтому второй затрагивает ферменты в более широком понимании, поэтому для дальнейшего сравнения буду использовать значение 1090.
Конвейер bash:
zcat UP000182508.swiss.gz | grep -E '^(DE EC=.*|CC -!- CATALYTIC ACTIVITY:.*)' | wc -l
В результате получилось 1034 фермента. Это чуть меньше ожидаемого результата. Вероятно, это связано с тем, что в конвейере я не учитывала строки KW, а также с тем, что запросы в bash и на сайте несколько отличались друг от друга.
Проверка, является ли в каждом белке Мет первой аминокислотой с помощью Python:
Таким образом, певрой аминокимлотой не в каждом белке протеома был метионин. Это могло бы происходить потому, что метионин в зрелых белках может расщепляться ферментом метионинаминопептидазой (MAP). Эффективность удаления зависит от кислоты после метионина: маленькие незаряженные к-ты (Ala, Ser, Cys и т.д.) способствуют удалению Мет, а большие заряженные аминокислоты обычно препятствуют удалению. Но в FT не было никакой информации об этом, поэтому это маловероятно. Также возможно, что тут содержится неполный белок.