A- и B-формы ДНК
1.
С помощью программы fiber пакета 3DNA построены A- и B-формы дуплекса ДНК, последовательность одной из нитей
которого — 4 раза повторенная последовательность "gatc".
Структура дуплекса в А-форме находится в файле gatc_a.pdb и получена с помощью команды:
fiber -a gatc_a.pdb
А структура дуплекса в В-форме находится в файле gatc_b.pdb и получена с помощью команды:
fiber -b gatc_b.pdb
2.
Таблица ниже заполнена по данным файлов, полученных в первом задании и файла dna36.pdb.
| |
A-форма |
B-форма |
Файл dna36.pdb |
| Тип спирали (правая или левая) |
Правая |
Правая |
Правая |
| Шаг спирали (Å) |
28.028 ангстрем |
33.752 ангстрем |
28.92 ангстрем |
| Число оснований на виток |
11 |
10 |
8 |
| Ширина большой бороздки |
7.98 (от 8 фосфора) |
17.21 (от 8 фосфора) |
15.354 (от 2 фосфора) |
| Ширина малой бороздки |
16.80 (от 8 фосфора) |
11.69 (от 8 фосфора) |
10.360 (от 7 фосфора) |
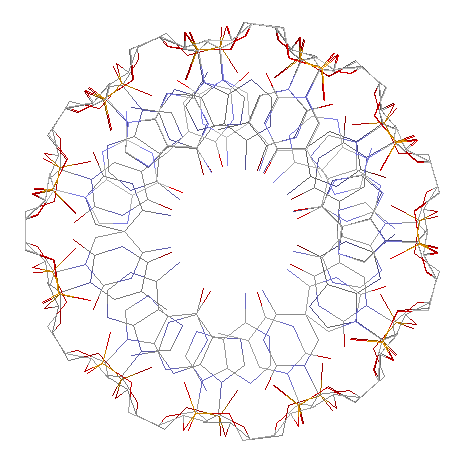 А-структура
А-структура
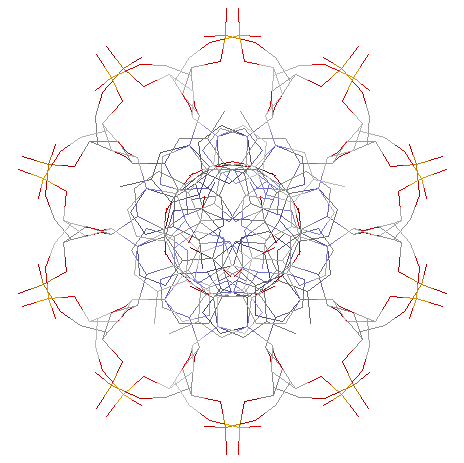 B-структура
B-структура
Как видно из результатов таблицы,обе формы ДНК как А-форма, так и В-форма являются правозакрученными двойными спиралями.
Основным визуальным отличием между ними является то, что на виде с торца у А-формы будет хорошо заметное отверстие по середине, а у В-формы оно совсем незначительное.
Также, воспользовавшись возможностями rasmol была измерена ширина большой и малой бороздок у обеих форм.
У A-формы ширина большой бороздки меньше ширины малой бороздки.
Для В-формы все наоборот, то есть ширина большей бороздки больше ширины малой бороздки.
Данная же мне структура визуально больше похожа на B-форму.
Более того, ширина большой бороздки больше чем малой, это лишний раз подчеркивает, что наше предположение верно.
4.Анализ всех трёх структур ДНК с помощью программ find_pair и analyze
find_pair -t gatc_a.pdb stdout | analyze
find_pair -t gatc_b.pdb.pdb stdout | analyze
find_pair -t dna36.pdb.pdb stdout | analyze
для dna36t.pdb
Strand I
base alpha beta gamma delta epsilon zeta chi
1 C --- --- 170.0 98.1 -164.1 -77.2 -152.7
2 C -70.0 177.0 56.9 148.5 -99.9 165.2 -83.7
3 A -74.7 145.9 52.1 143.0 173.4 -87.1 -82.3
4 A -70.1 177.7 47.5 123.7 173.6 -96.9 -96.8
5 C -65.2 -179.7 49.2 144.8 -114.2 -179.1 -95.6
6 G -75.5 153.4 43.4 130.4 168.8 -88.7 -105.3
7 T -64.4 170.5 65.3 125.6 -163.6 -85.3 -129.6
8 T -81.9 -173.3 45.5 134.3 -100.3 167.3 -88.1
9 G -75.6 152.2 48.3 145.6 -172.8 -87.9 -76.3
10 G -69.4 159.1 52.6 96.2 --- --- -107.4
Strand II
base alpha beta gamma delta epsilon zeta chi
1 G -69.4 159.1 52.6 96.2 --- --- -107.4
2 G -75.6 152.2 48.3 145.6 -172.8 -87.9 -76.3
3 T -81.9 -173.3 45.5 134.3 -100.3 167.3 -88.1
4 T -64.4 170.5 65.3 125.6 -163.6 -85.3 -129.6
5 G -75.5 153.4 43.4 130.4 168.8 -88.7 -105.3
6 C -65.2 -179.7 49.2 144.8 -114.2 -179.1 -95.6
7 A -70.1 177.7 47.5 123.7 173.6 -96.9 -96.8
8 A -74.7 145.9 52.1 143.0 173.4 -87.1 -82.3
9 C -70.0 177.0 56.9 148.5 -99.9 165.2 -83.7
10 C --- --- 170.0 98.1 -164.1 -77.2 -152.7
для gtc_a.pdb
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2
2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2
7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2
9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2
13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
16 C -51.7 174.8 41.7 79.1 --- --- -157.2
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -51.7 174.8 41.7 79.0 --- --- -157.2
2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2
16 G --- 174.8 41.7 79.1 -147.7 -75.1 -157.2
для gtc_b.pdb
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0
2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0
14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
16 C -29.9 136.4 31.1 143.4 --- --- -98.0
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -29.9 136.4 31.1 143.4 --- --- -98.0
2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0
5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
16 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0
|
| α
| β
| γ
| δ
| ε
| ζ
| χ
|
| А
| -51.7
| 174.8
| 41.7
| 79.1
| -147.79
| -75.1
| -157.2
|
| B
| -29.9
| 136.3
| 31.14
| 143.3
| -140.8
| -160.5
| -97.98
|
| DNA36*
| -71.87
| 86.98
| 63.08
| 129.02
| -33.23
| -41.08
| -101.78
|
*были взяты средние значения величин, расчеты проведены были с помощью excel.
Результаты можно найти по адресу shure_beta/public_html/Term3/Practice7/вычисления.xml
Проанализировав результаты заданных нам торсионных углов, можно заключить, что данная структура является B-формой,
т.к. мы будем опираться на ее сходство углов chi c B-формой.
Анализ проводился именно по этому углу, т.к. именно он показывает расположение азотистых оснований относительно основной цепи.
Более того, интересно отметить, что в документе, выданном программой analyze в таблице structure classification было сказано,
что данная структура является B-формой.
5.Изображения структур в виде стопочных моделей, созданные с помощью программы pdb2img
Стопочные структуры для А-формы ДНК из файла gatc_a.pdb созданы командами
pdb2img -c gatc_a.pdb gatc_a.ps
rotate_mol -b gatc_a.pdb gatc_a2.pdb
pdb2img -c gatc_a2.pdb gatc_a2.ps
Стопочные структуры для B-формы ДНК из файла gatc_b.pdb созданы командами
pdb2img -c gatc_b.pdb gatc_b.ps
rotate_mol -b gatc_b.pdb gatc_b2.pdb
pdb2img -c gatc_b2.pdb
Стопочные структуры для РНК из файла dna36.pdb созданы командами
pdb2img -c dna36.pdb dna36.ps
rotate_mol -b dna36.pdb dna36_2.pdb
pdb2img -c dna36_2.pdb
| вид сверху | вид сбоку |
 |
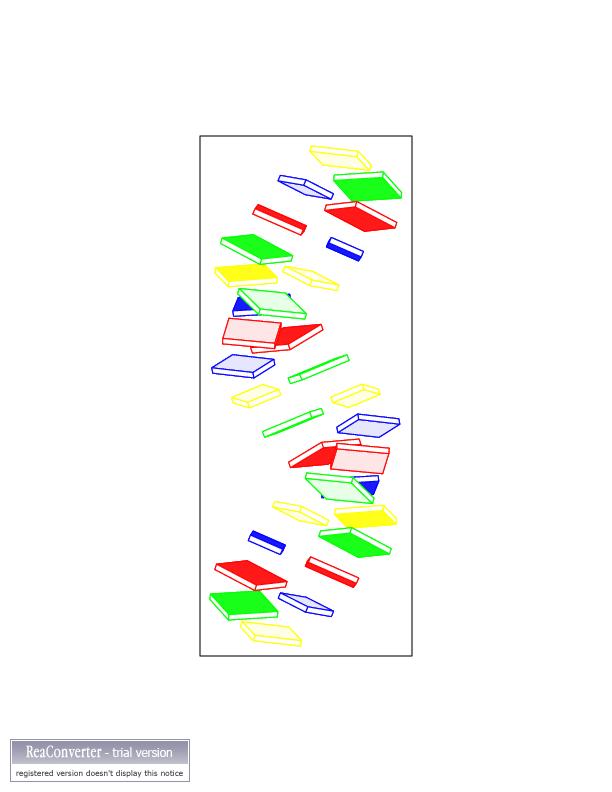 |
Видна характерная для А-формы ДНК полость внутри спирали.
На виде сбоку заметно, что азотистые основания наклонены.
| вид сверху | вид сбоку |
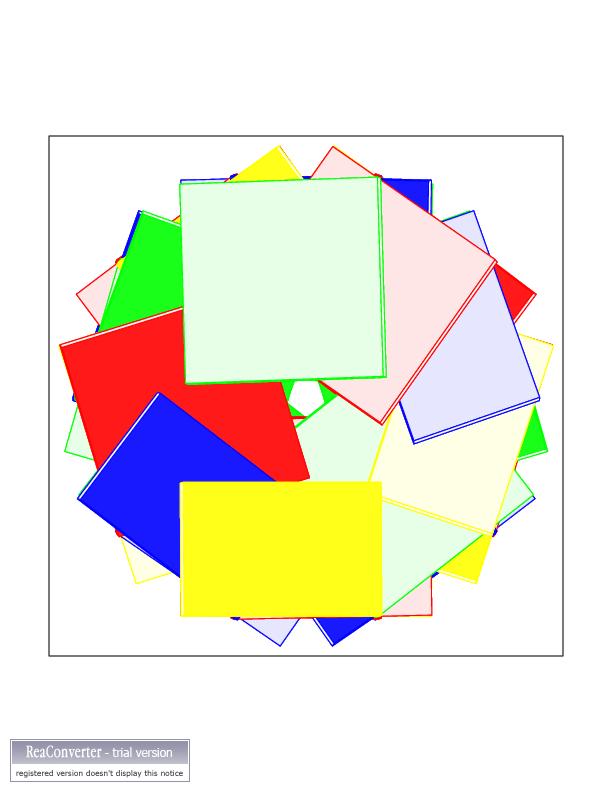 |
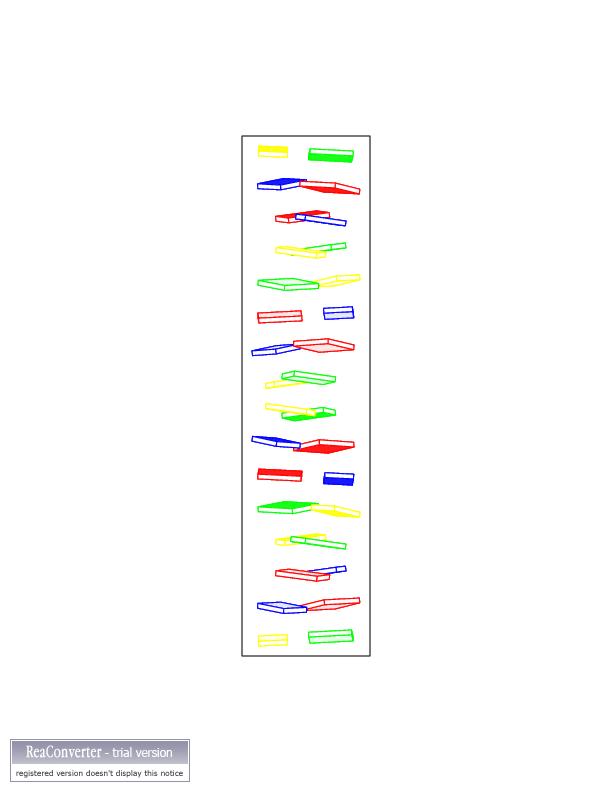 |
Внутри структуры отсутствует полость,
что говорит о том, что это В-форма ДНК.
На виде сбоку заметно,что азотистые основания находятся практически
перпендикулярно к оси спирали.
| вид сверху | вид сбоку |
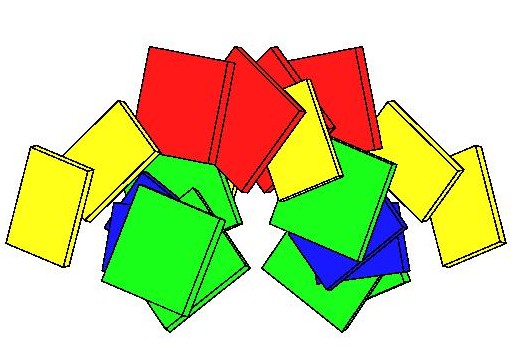 |
 |
На виде с торца структура почему-то не до конца довернулась.
На виде сбоку азотистые основания практически перпендикулярны оси вращения, соответственно, это указывает нам, что это В-форма.
На главную
На страницу третьего семестра
©Вахрушева Анна Алексеевна


 А-структура
А-структура
 B-структура
B-структура





