Моделирование и реконструкция эволюции гена
1.Создание изображения дерева, описанного заданной формулой
((((А:80,B:80):15,C:90):10,D:90):10,(E:50,F:50):50);
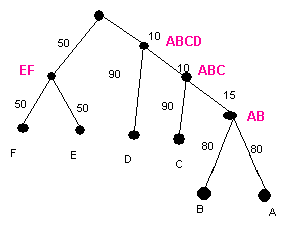
2.Считая дерево бескорневым, описаны ветви дерева как разбиения множества листьев.
| A | B | C | D | E | F |
| . | . | * | * | * | * |
| . | . | . | * | * | * |
| . | . | . | . | * | * |
3.Получение искуственных мутантных последовательностей
В корне находится последовательность гена белка SYD_ECOLI, длина которой составляет 1833 нуклеотида.
С помощью данного скрипта были созданы возможные мутантные последовательности для листьев дерева:
msbar x53863.fasta EF.fasta -point 4 -count 917 -auto
msbar x53863.fasta ABCD.fasta -point 4 -count 183 -auto
msbar ABCD.fasta ABC.fasta -point 4 -count 183 -auto
msbar ABCD.fasta D.fasta -point 4 -count 1650 -auto
msbar ABC.fasta AB.fasta -point 4 -count 275 -auto
msbar ABC.fasta C.fasta -point 4 -count 1650 -auto
msbar AB.fasta A.fasta -point 4 -count 1466 -auto
msbar AB.fasta B.fasta -point 4 -count 1466 -auto
msbar EF.fasta E.fasta -point 4 -count 917 -auto
msbar EF.fasta F.fasta -point 4 -count 917 -auto
Формула для пересчёта расстояний в число мутаций в заданном гене:
[(Длина гена)/100]*длину ветви
4.На основе последовательностей, соответствующих листьям, реконструировано дерево алгоритмами UPGMA, Neighbor-joining и максимального правдоподобия.
С помощью следующей команды было построено филогенетичексое дерево алгоритмом максимального подобия:
fdnaml 1.fasta -ttratio 1 -auto
В итоге получилось следующее дерево:
+-----------------B
|
| +----------F
| +--------------4
| +---3 +-------------E
| | |
1----2 +--------------------D
| |
| +--------------------C
|
+------------------A
| A | B | C | D | E | F |
| . | . | * | * | * | * |
| . | . | . | * | * | * |
| . | . | . | . | * | * |
С помощью следующих команд было построено филогенетичексое дерево алгоритмом Neighbor-joining:
fdnadist 1.fasta -ttratio 1 -auto
fneighbor 1.fdnadist -auto
В итоге получилось следующее дерево:
+-----------------B
!
! +--------------------C
2-----3
! ! +--------------------D
! +---4
! ! +-------------E
! +---------------1
! +----------F
!
+-------------------A
| A | B | C | D | E | F |
| . | . | * | * | * | * |
| . | . | . | * | * | * |
| . | . | . | . | * | * |
С помощью следующих команд было построено филогенетичексое дерево алгоритмом UPGMA:
fdnadist 1.fasta -ttratio 1 -auto
fneighbor 1.fdnadist -treetype u -auto -outfile 2
В итоге получилось следующее дерево:
+-------------------------------------A
+-------2
+-3 +-------------------------------------B
! !
+-----4 +---------------------------------------------C
! !
--5 +-----------------------------------------------D
!
! +------------------------E
+----------------------------1
+------------------------F
Aлгоритм UPGMA (Unweighted Pair Group Method with Arithmetic Mean или невзвешенная попарная кластеризация)�
Кластерный метод, в котором расстояние между кластерами вычисляется как среднее арифметическое расстояний между их элементами.
Как видно, по алгоритму UPGMA мы получаем укорененное (в отличие от предыдущим алгоритмов) ультраметрическое дерево, т.е. дерево, в котором суммарные расстояния от каждого листа до корня одинаковы.
Этот факт можно обьяснить тем, что этот алгоритм основывается на гипотезе молекулярных часов, которая гласит, что за равное время во всех ветвях
накапливается равное число мутаций.
| A | B | C | D | E | F |
| . | . | * | * | * | * |
| . | . | . | * | * | * |
| . | . | . | . | * | * |
Сравнение полученных разными программами филогенетических деревьев:
| Ветвь
|
Исходное дерево |
Дерево, построенное алгоритмом максимального подобия |
Дерево, построенное алгоритмом Neighbor-joining |
Дерево, построенное алгоритмом UPGMA |
| A |
B |
C |
D |
E |
F |
| . |
. |
* |
* |
* |
* |
+ |
+ |
+ |
+ |
| . |
. |
. |
* |
* |
* |
+ |
+ |
+ |
+ |
| . |
. |
. |
. |
* |
* |
+ |
+ |
+ |
+ |
Проанализировав полученные схемы филогенетических деревьев можно заметить,
что они имеют те же ветви что и исходное бескорневое дерево.
То есть можно заключить, что приведенные выше программы успешно справились с поставленной задачей.
На главную
На страницу четвертого семестра
©Вахрушева Анна Алексеевна