Исследование структуры тРНК
I.Краткое описание структуры в файле 1F7U.pdb
В файле приведены координаты атомов следующих молекул
- ARGINYL-TRNA SYNTHETASE (Аргинил-тРНК синтетаза EC: 6.1.1.19)
- TRNA(ARG) (Аргининовая тРНК)
Аргинил-тРНК синтетаза была обнаружена в SACCHAROMYCES CEREVISAE (тривиальное название YEAST) и экспрессирована при помощи ESCHERICHIA COLI. Молекула тРНК была синтезирована искусственно по прообразу Аргининовой тРНК из SACCHAROMYCES CEREVISAE.
.
Для исследования была выбрана цепь B, представляющая Аргинин-спецефичную тРНК со следующей последовательностью:
[901] 5' - PSU U C C U C G U 1MG 2MG C C C A A H2U G G H2U C A C G G C M2G PSU C U G G C U I C G A A C C A G A A G A H2U U 5MC C A G G 5MU PSU C A 1MA G U C C U G G C G G G G A A G C C A - 3' [976], где
901 и 976 - номера первого и последнего нуклеотида.
Как видно из последовательности, на 3' конце присутсвтует ССА триплет, к которому присоединяется аминокислота, в данном случае аргинин.
II.Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (hbonds).
В соответствии с полученными данными
Акцепторный стебель...............902-907 -- 971-966
Т-стебель...................................950-953 -- 964-961
D-стебель..................................911-913 -- 924-922
Антикодоновый стебель............928-931 -- 942-939
Вторичная структура аргининовой тРНК
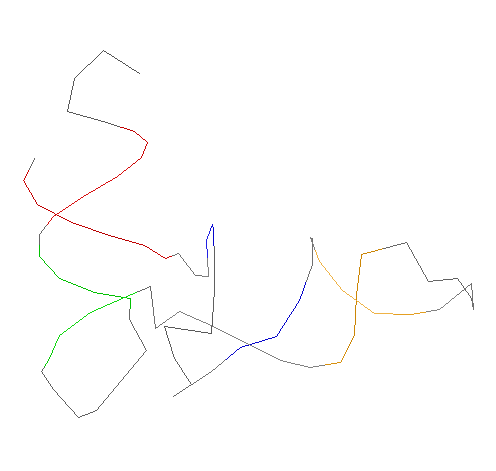
|
echo "Hello, this RasMol script uses 1f7u.pdb"
echo "file and shows RNA backbone with stem color."
echo "Please, open 1f7u.pdb in RasMol window,"
echo "or using load before using this script"
pause
echo "If 1f7u.pdb is loaded please press "
echo "Predefined sets of atoms:"
echo "Acceptable stem : acStem (902-907) and (966-971)"
echo "D-stem : dStem (911-913) and (922-924)"
echo "T-stem : tStem (950-953) and (961-964)"
echo "AntiKodon-stem : akdStem (928-931) and (939-942)"
pause
define acStem ((902-907:B) or (966-971:B))
define dStem ((911-913:B) or (922-924:B))
define tStem ((950-953:B) or (961-964:B))
define akdStem ((928-931:B) or (939-942:B))
restrict rna
background white
select rna
color gray
wireframe off
backbone on
select acStem
color red
select tStem
color green
select dStem
color blue
select akdStem
color orange
echo "Backbone RNA model with stem color"
echo "Backbone is gray"
echo "Acceptable stem is red"
echo "D-stem is green"
echo "T-stem is green"
echo "AntiKodon stem is orange"
echo "GoodBye!"
|
Структуру стеблевых дуплексов поддерживают 15 канонических и 4 неканонических пар оснований.
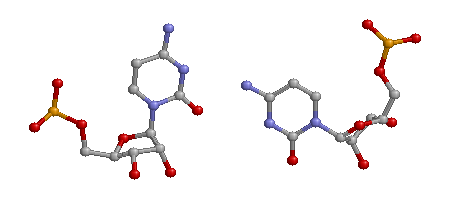
Пример неканонической С(913) -- С(922) пары
Особенности структуры тРНК
а)Судя по изображению, полученному в RasMol, вариабельную петлю составляют 946-947-948 нуклеотиды.
б)Отсутствуют остатки тимидина в Т-петле.
в)Имеются остатки дигидроуридинов в D-петле (916,919).
Антикодон. Исходя из этого, я затрудняюсь указать антикодон, так как основания нуклеотидов 934 и 935 лежат параллельно за пределами внутреннего пространства упакованной тРНК, однако 3-го нуклеотида вписывающегося в эту картину нет. По всей видимости, это все-таки 935-933 (C-I(=G)-U). Так как по таблице, кодон ,кодирующий аргинин, должен на 2-ой позиции иметь G, однако в данном случае G 936 нуклеотид, а 937 обращен внутрь спирали, что не позволяет ему быть в составе антикодона. А I (по всей видимости Гипоксантин) по своему строению идентичен G, за исключением аминогруппы в С2 положении.
III.Предсказание вторичной структуры тРНК
1. По всей видимости, стекинг-взаимодействие между основаниями конца акцепторно стебля и начала Т-стебля отсутсвуют.
(и/или между антикодоновым и D-стеблем). Значение перекрывания 0.00. (Исследовать вариант антикодон и D-стебель не представляется возможным, так как в списке позиций между ними добавлена свзяь D-T) 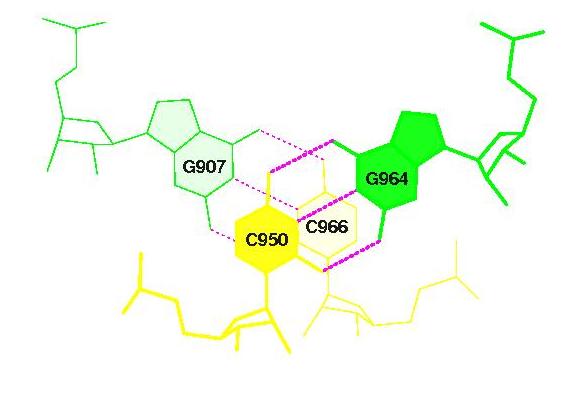
2. Присутствует дополнительная водородная связь D и T стеблей. Это связь G 918 -- 956 C
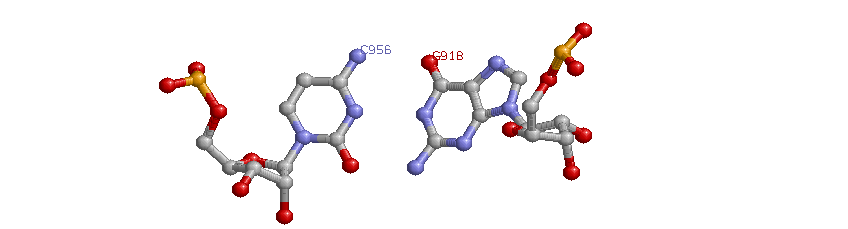
Предсказание вторичной структуры тРНК
Участок структуры
|
Позиции в структуре
(по результатам find_pair)
|
Результаты предсказания
с помощью einverted
|
Результаты предсказания
по алгоритму Зукера*
|
| Акцепторный стебель
|
5' 902-907 3'
3' 971-966 5'
Всего 6 пар
|
предсказано 0 пар из 6 реальных
|
предсказано 4 пары из 6 реальных
|
| D-стебель
|
5' 911-913 3'
3' 924-922 5'
Всего 3 пары
|
предсказано 0 пар из 3 реальных
|
предсказано 0 пар из 3 реальных
|
| T-стебель
|
5' 950-953 3'
3' 964-961 5'
Всего 4 пары
|
предсказано 4 пары из 4 реальных
|
предсказано 4 пары из 4 реальных
|
| Антикодоновый стебель
|
5' 928-931 3'
3' 942-939 5'
Всего 4 пары
|
предсказано 0 пар из 4 реальных
|
предсказано 4 пары из 4 реальных
|
| Общее число канонических пар нуклеотидов
|
16
|
4
|
11
|
*Для реализации алгоритма Зукера использовался пакет UNAFold.
| UNAFold при -P 10 Вариация 2 |
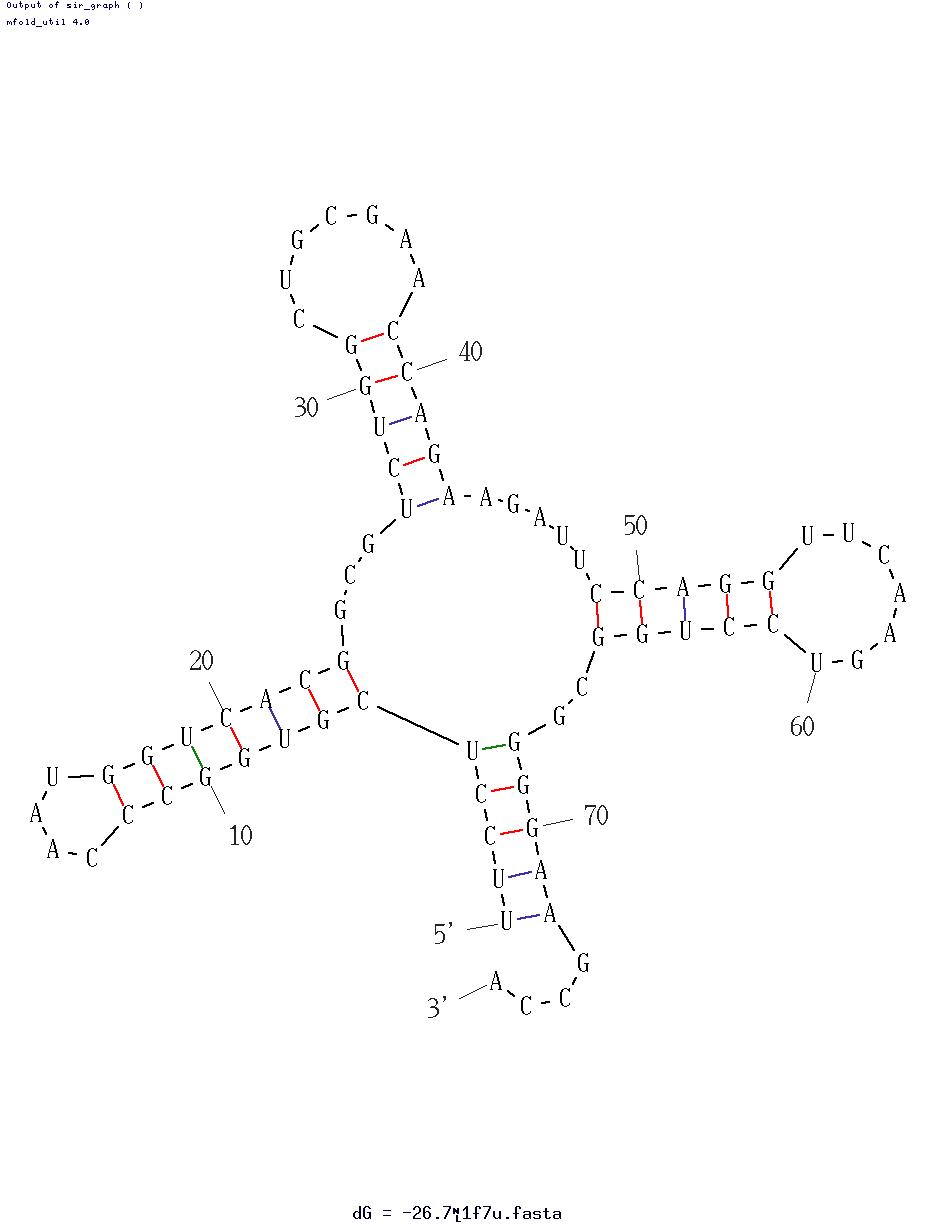 |
| Комментарии |
Команда einvert пактеа EMBOSS ищет комплементарные участки в цепи, но она способна "согнуть" последовательность только 1 раз, к тому же предлагается использовать довольно высокие штрафы за гэп и несовпадение, что в случае целой РНК не является корректным, так как каждый стебель это "складка" на нуклеотидной цепочке. Поэтому и был найден только один стебель. На мой взгляд корректнее использовать palindrom из пакета EMBOSS, хотя и его результаты далеки от идеала.
Пакет UNAFold, реализующий алгоритм Зукера, является логическим продолжением mfold, однако обеспечивает большую скорость и удобство работы. По умолчанию он запускается с параметром -P 5. Однако в этом случае предлагается всего лишь одна вариация с несколькими длинными стеблями и практически отсутсвующими петлями, даже отдаленно не напоминающая каноническую вторичную структуру тРНК. Следующий запуск с -P 10 дал еще 2 вариации. 3-я вариация похожа на 1-я, как следствие нас не интересует. А вот 2-я вариация внешне очень похожа на каноническую структуру тРНК имеется 4 хорошо разделенных стебля, 4 полноценных петли. Как оказалось при более подробном рассмотрении, эта структура довольно хорошо корреллирует с реальной. При пробных запусках с щагом 5% было получено все увеличивавшееся количество вариаций, однако ни одна не превосходила вариацию 2, а в большинстве своем они были похожи на все что угодно, кроме "клеверного листа" тРНК.
|
|




