| Главная |
| Учебные материалы |
Занятие 10-11
Выбранный домен:AC: PF02065
ID: Melibiase
Glycoside hydrolase - гликозид гидролаза
Всего - 44 архитектуры в ~2к последовательностей у ~900 видов. Известно 24 структуры.
Выравнивание в java
Выбранные доменные архитектуры:
1650 последовательностей с монодоменной архитектурой:

Встречается как у Архей, Бактерий и Еукариот.
101 последовательность с дидоменной архитектурой:
Встречается только у Бактерий и Эукариот.
Таблица в Excel
Выравнивание в jar
Архитектуры и таксоны зашифрованы следующим образом: 1 - монодоменная архитектура, 2 - дидоменная,
A - Археи, B - Бактерии, E - Еукариоты.
По соответствующему выравниваю при помощи алгоритма Neighbor joining было выстроено филогенетическое древо.
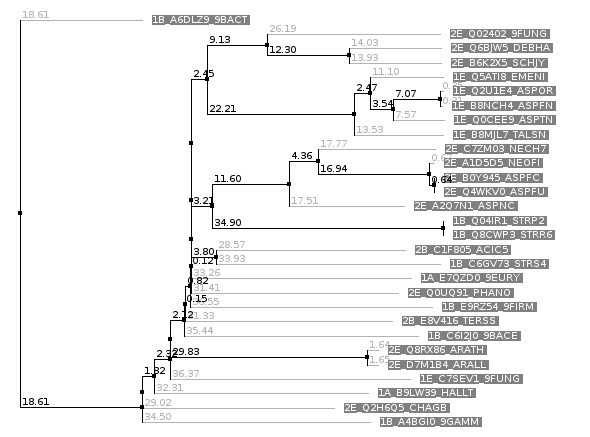
Хорошо видно, что одно и двух доменные архитектуры хорошо разделены, при этом для каждой архитектуры видно четкое разбиение на бактериальные и эукариотические клады. Археи также попали на отдельную кладу. Исходя из такой картины можно предположить, что исходно существовал вариант с монодоменной архитектурой, который однажды удвоился и закрепился.
Выравнивание было разделено на 2 части по доменной архитектуре: 1d.msf и 2dn.msf. На основе них были построены профили.1df.p и 2df.p. При этом в случае монодоменной архитектуры для обеспечения лучшего качества профиля были исключены археи а в случае дидоменной бактерии. При этом для 2го профиля получаются довольно интересные результаты:
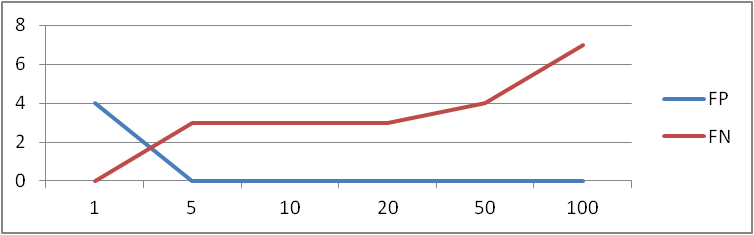
110.050 11005 pos. 25 - 616 2E_Q02402_9FUNG/1-834 99.350 9935 pos. 25 - 619 2E_Q6BJW5_DEBHA/1-834 1.160 116 pos. 731 - 834 2E_Q6BJW5_DEBHA/1-834 109.560 10956 pos. 22 - 616 2E_B6K2X5_SCHJY/1-830 1.820 182 pos. 798 - 830 2E_B6K2X5_SCHJY/1-830 87.130 8713 pos. 27 - 602 2B_E6MLM8_9BACT/1-816 133.400 13340 pos. 19 - 608 2E_C7ZM03_NECH7/1-814 266.790 26679 pos. 26 - 615 2E_A1D5D5_NEOFI/1-812 267.910 26791 pos. 26 - 615 2E_B0Y945_ASPFC/1-812 1.030 103 pos. 1 - 9 2B_C1F805_ACIC5/1-812 55.760 5576 pos. 40 - 618 2B_C1F805_ACIC5/1-812 266.850 26685 pos. 26 - 615 2E_Q4WKV0_ASPFU/1-812 86.250 8625 pos. 29 - 604 2E_Q2H6Q5_CHAGB/1-800 82.950 8295 pos. 37 - 603 2E_Q0UQ91_PHANO/1-792 80.310 8031 pos. 36 - 603 2E_Q8RX86_ARATH/1-792 80.170 8017 pos. 34 - 601 2E_D7M1B4_ARALL/1-788 65.060 6506 pos. 26 - 604 2B_E8V416_TERSS/1-788 169.630 16963 pos. 31 - 603 2E_A2Q7N1_ASPNC/1-782 1.030 103 pos. 703 - 731 1E_C7ZIF8_NECH7/1-731 1.090 109 pos. 693 - 719 1B_C6I2J0_9BACE/1-719 1.100 110 pos. 706 - 712 1B_A6DLZ9_9BACT/1-712 1.060 106 pos. 1 - 14 1A_E7QZD0_9EURY/1-734Откуда видно, что FP при весе равном 2 уже будет равняться нули и расти не будет. А FN до веса ~60 будет оставаться равным 3м.
Для профиля 1d картина аналогична:
45.780 4578 pos. 171 - 601 1B_E9RZ54_9FIRM/1-732 103.880 10388 pos. 179 - 621 1E_Q5ATI8_EMENI/1-732 35.950 3595 pos. 175 - 607 1E_C7ZIF8_NECH7/1-731 71.770 7177 pos. 171 - 600 1B_C6GV73_STRS4/1-724 26.810 2681 pos. 170 - 603 1E_C7SEV1_9FUNG/1-724 71.530 7153 pos. 167 - 596 1B_Q04IR1_STRP2/1-720 103.270 10327 pos. 159 - 601 1E_Q2U1E4_ASPOR/1-720 71.530 7153 pos. 167 - 596 1B_Q8CWP3_STRR6/1-720 103.720 10372 pos. 159 - 601 1E_B8NCH4_ASPFN/1-719 21.700 2170 pos. 154 - 583 1B_C6I2J0_9BACE/1-719 18.100 1810 pos. 173 - 605 1B_A6DLZ9_9BACT/1-712 103.360 10336 pos. 157 - 599 1E_Q0CEE9_ASPTN/1-709 31.510 3151 pos. 151 - 566 1B_A4BGI0_9GAMM/1-708 98.790 9879 pos. 148 - 590 1E_B8MJL7_TALSN/1-700
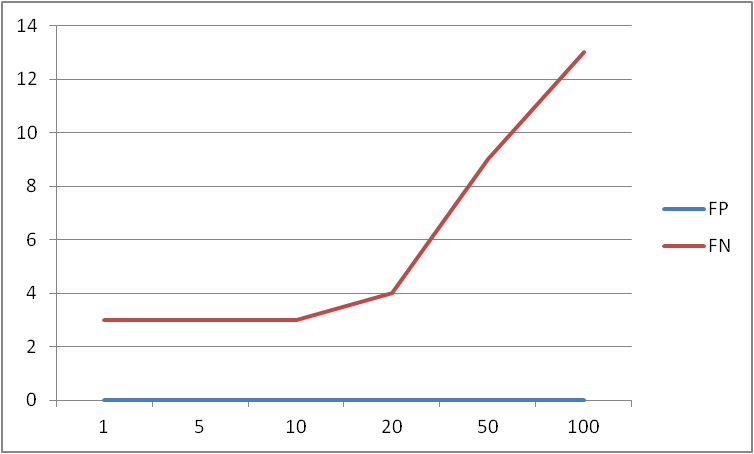
Уже при весе равном 1 FP 0, а FN 3 не будет увеличиваться до веса ~20. Таким образом, учитывая особенности подготовки профилей, они неплохо подходят для поиска последовательностей бактерий и эукариот с однодоменной архитектурой (с трэшхолдом по весу 10) и еукариот и части бактерий с дидоменной архитектурой (трешхолд по весу 20)
| ©Залевский, Артур, 2007 |