Практикум 11
Описание семейства доменов из Pfam
Для данного практикума я выбрала семейство, характеристики которого представлены в таблице 1.
Таблица 1. Характеристики выбранного семейства доменов из Pfam.
| Характеристика |
Информация |
| AC Pfam |
PF16876 |
| ID Pfam |
Lipin/Ned1/Smp2 multi-domain protein middle domain |
| #Seed |
41 |
| #All |
7k |
| #SW |
9 |
| #Architectures |
65 |
| #3D |
3 |
| #Eukaryota |
7013 |
| #Archaea |
0 |
| #Bacteria |
1 |
Данный домен является средним доменом липинов – ферментов, которые катализируют реакцию превращения фосфатидной кислоты в диацилглицерол в процессе биосинтеза триглицеридов, фосфатидилхолина и фосфатидилэтаноламина.
Данный домен распространен в основном среди эукариот. Среди бактерий обнаружен только один белок из этого домена.
Выравнивание seed с точки зрения гомологичности всех последовательностей или их подмножества
Выравнивание seed было открыто с помощью Jalview.
Выравнивание в Jalview
Характеристики выравнивания seed представлены в таблице 2.
Таблица 2. Характеристики выравнивания seed.
| Характеристика |
Информация |
| Выравнивание seed |
145 колонок |
| Максимальный достоверный блок, включающий все последовательности (МДБ-all) |
68-84 |
| 100% консервативные колонки в МДБ-all |
74:[A](37 из 41) |
| Максимальный достоверный блок, включающий НЕ все последовательности (МДБ-notAll) |
Колонки: 67-89, последовательности: 1-22 |
| 100% консервативные колонки в МДБ-notAll |
74:A; 76:P; 89:P |
| Участок выравнивания, в котором нет никаких достоверных подблоков, и потому маловероятно, что выравнивание на этом участке отражает ход эволюции. |
90-100 (см. пояснения ниже) |
В максимальном достоверном блоке не нашлось ни одной колонки с абсолютной консервативностью. Однако есть колонка 74, где в 37 из 41 последовательности в данной позиции находится аланин. Для того, чтобы обнаружить эту колонку я снизила порог на Identity threshold до 90%.
При поиске участков выравнивания, в которых нет достоверных подблоков, я не нашла участков, на которых нет сходства даже для двух последовательностей.
Однако, например, есть участок 90-100. На данном участке я выбрала неконсервативные колонки, сделала по ним группы. Есть две группы, которые состоят из двух последовательностей, но все остальные последовательности единственные в своей группе.
Следовательно, маловероятно, что на этом участке выравнивание отражает ход эволюции.
В целом на всей длине выравнивания прослеживаются консервативные участки, которые отражают гомологию данных последовательностей. Участки неконсервативных колонок, идущих подряд, присутствуют, но, как правило, они достаточно короткие (2-3 колонки).
Карта локального сходства (dotplot) двух последовательностей с одним и тем же доменом, но с разной доменной архитектурой
Для построения карты локального сходства (dotplot) я выбрала две последовательности с одним и тем же доменом, но с разными доменными архитектурами (таблица 3).
Таблица 3. Информация о выбранных доменных архитектурах и их белках.
| Доменная архитектура |
Информация о доменной архитектуре |
Идентификатор белка с данной архитектурой |
| 1 |
PF04571 - PF16876 - PF08235 |
Q92539 |
| 2 |
PF16876 - PF08235 |
Q962L8 |
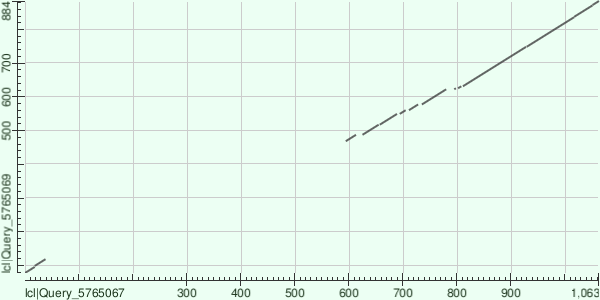 Рисунок 1. Карта локального сходства (dotplot) выбранных последовательностей.
Рисунок 1. Карта локального сходства (dotplot) выбранных последовательностей.
При помощи BLASTp на сайте NCBI была построена карта локального сходства (рис. 1). По вертикальной оси отложена последовательность белка Q92539, по горизонтальной – Q962L8.
На карте можно заметить два участка локального сходства (то есть на данных участках мы наблюдаем сходство двух белковых последовательностей). Стоит заметить, что одна из линий прерывиста. Разрывы соответствуют инделям в выравнивании данных двух последовательностей.