Практикум 14
Для выполнения данного практикума мне был предоставлен код доступа проекта по секвенированию бактерии Buchnera aphidicola str. Tuc7.
Код доступа: SRR4240356.
Далее был скачан архив с чтениями посредством следующей команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/006/SRR4240356/SRR4240356.fastq.gz
Подготовка чтений
Для начала я удалила возможные остатки адаптеров. Для этого я объединила файлы с адаптерами в один при помощи следующей команды:
cat /mnt/scratch/NGS/adapters/*.fa > adapters.fa
После этого я запустила следующую команду:
TrimmomaticSE -phred33 SRR4240356.fastq.gz SRR4240356_noadapt.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7
Результат работы:
Input Reads: 7511529 Surviving: 7358438 (97.96%) Dropped: 153091 (2.04%)
То есть 2.04% последовательностей оказались адаптерами.
После этого с правых концов чтений были удалены нуклеотиды с качеством ниже 20, а также были оставлены только чтения с длиной не меньше 32 нуклеотидов с помощью следующей команды:
TrimmomaticSE -phred33 SRR4240356_noadapt.fastq.gz SRR4240356_noadapt2.fastq.gz TRAILING:20 MINLEN:32
Результат работы:
Input Reads: 7358438 Surviving: 7053346 (95.85%) Dropped: 305092 (4.15%)
Было удалено 4.15% чтений.
При этом размер изначального файла с чтениями 167 Мб, после удаления адаптеров – 164 Мб, размер финального файла – 155 Мб.
Подготовка k-меров
Следующим шагом я подготовила k-меры длины 31 при помощи следующей команды:
velveth k-mers 31 -fastq.gz -short SRR4240356_noadapt2.fastq.gz
Сборка на основе k-меров
После этого я запустила следующую команду:
velvetg k-mers
N50: 65554
Информация о контигах содержится в файле contigs.fa, полученном в результате работы программы.
Для того, чтобы узнать длины трёх самых длинных контигов я воспользовалась следующим конвейером:
grep -e '>' contigs.fa | sort -t "_" -k4 -n -r
Три самых длинных контига:
- Контиг 8, длина: 111962, покрытие: 38.660198
- Контиг 6, длина: 107488, покрытие: 34.174030
- Контиг 10: длина: 80939, покрытие: 37.524174
Медианное покрытие: 16.63705
После этого я отсортировала контиги по покрытию с помощью следующего конвейера:
grep -e '>' contigs.fa | sort -t "_" -k6 -n -r
Найдены контиги с аномально большим покрытием. Например, контиг 27 с покрытием 458.429077, контиг 17 с покрытием 447.494751, контиг 14 с покрытием 444.608124.
Также есть контиги с аномально малым покрытием: контиг 74 с покрытием 3.064516 и контиг 123 с покрытием 2.362637.
Анализ
После этого при помощи megablast я сравнила каждый из трёх самых длинных контигов с хромосомой Buchnera aphidicola (AC: CP009253).
Контиг 8
Координаты участка хромосомы, соответствующего контигу: 451729-555905
Процент гэпов: 2.6%
Всего получилось 15 участков, на которых последовательности выровнялись (рисунок 1).
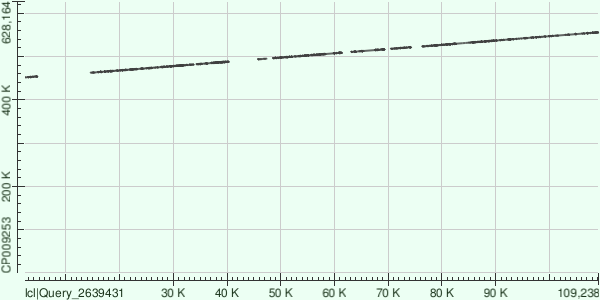 Рисунок 1. Карта локального сходства для контига 8 и хромосомы Buchnera aphidicola.
Рисунок 1. Карта локального сходства для контига 8 и хромосомы Buchnera aphidicola.
Контиг 6
Координаты участка хромосомы, соответствующего контигу: 220869-323043
Процент гэпов: 2.6%
Всего получилось 18 участков, на которых последовательности выровнялись (рисунок 2).
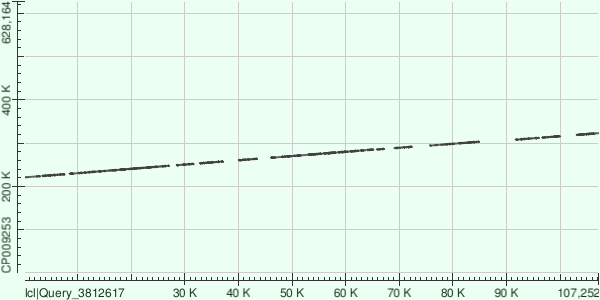 Рисунок 2. Карта локального сходства для контига 6 и хромосомы Buchnera aphidicola.
Рисунок 2. Карта локального сходства для контига 6 и хромосомы Buchnera aphidicola.
Контиг 10
Координаты участка хромосомы, соответствующего контигу: 126623-195400
Процент гэпов: 1.7%
Всего получилось 11 участков, на которых последовательности выровнялись (рисунок 3).
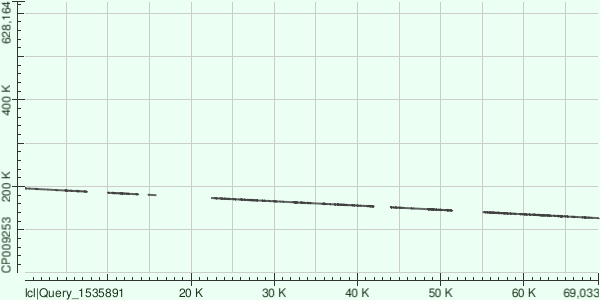 Рисунок 3. Карта локального сходства для контига 10 и хромосомы Buchnera aphidicola.
Рисунок 3. Карта локального сходства для контига 10 и хромосомы Buchnera aphidicola.
Можно заметить, что на всех картах локального выравнивания есть "разрывы". Возможно, эти разрывы соответствуют вариабельным участкам.