Практикум 10
Мотивы в белках
Поиск консервативных мотивов в выравнивании
Для выполнения данного практикума я выбрала домен из базы данных Pfam.
Это С-концевой домен бактериального тетрациклина (AC Pfam: PF18556).
Выравнивание seed содержит 55 последовательностей. Оно было скачано. Расширение файла было изменено на .msf.
Далее данное выравнивание было открыто при помощи программы Jalview. Всего в выравнивании было представлено 55 последовательностей. Полностью идентичных последовательностей в выравнивании не было.
После этого было включено отображение цветом позиций с высоким уровнем идентичности. Полностью идентичных колонок было только две (позиции 1 и 99). Порог идентичности постепенно снижался. Таким образом, мы наглядно можем увидеть мотивы. Мотивом считались вплотную или рядом расположенные консервативные позиции.
Следующим шагом был составлен паттерн для найденного мотива и был произведен поиск по этому паттерну во всем выравнивании.
Паттерн: [RQ].{3}[SA].{4}P
Данный паттерн встретился 63 раза (при том, что последовательностей 55). То есть паттерн не является высокоспецифичным. Это не очень хорошо, однако если смотреть из представленных вариантов при данном пороге идентичности (90%), он лучший.
После этого паттерн был переведен в формат Prosite.
Паттерн в формате Prosite: [RQ]-x(3)-[SA]-x(4)-P
Далее я попыталась по данному паттерну провести поиск в базе данных SwissProt в Prosite, однако мне выдало ошибку: мой паттерн слишком вырожденный. Тогда я вернулась на этап составления паттерна мотива в Jalview, еще более уменьшив порог (до 70%).
Паттерн: [LM][LMHV].[LIYFH].[TA][TLIAVQSN]
Данный паттерн встретился в выравнивании 56 раз. То есть он более специфичный, чем предыдущий.
Паттерн в формате Prosite: [LM]-[LMHV]-x-[LIYFH]-x-[TA]-[TLIAVQSN]
По этому паттерну был выполнен поиск в базе данных SwissProt в Prosite.
Результат: 11646 находок в 10000 последовательностях.
Отсюда можно сделать вывод, что мотив неспецифичен: число находок больше, чем общее число последовательностей, то есть в некоторых последовательностях он встретился больше одного раза. Я думаю, что это связано с тем, что под данный паттерн могут подходить случайные участки, ведь в нем нет ни одной полностью консервативной позиции.
Поиск мотива, специфичного для одной клады филогенетического дерева
В Jalview было построено филогенетическое дерево методом NJ.
Далее в данном филогенетическом дереве была выбрана ветвь, отрезающая одну кладу (рисунок 1).
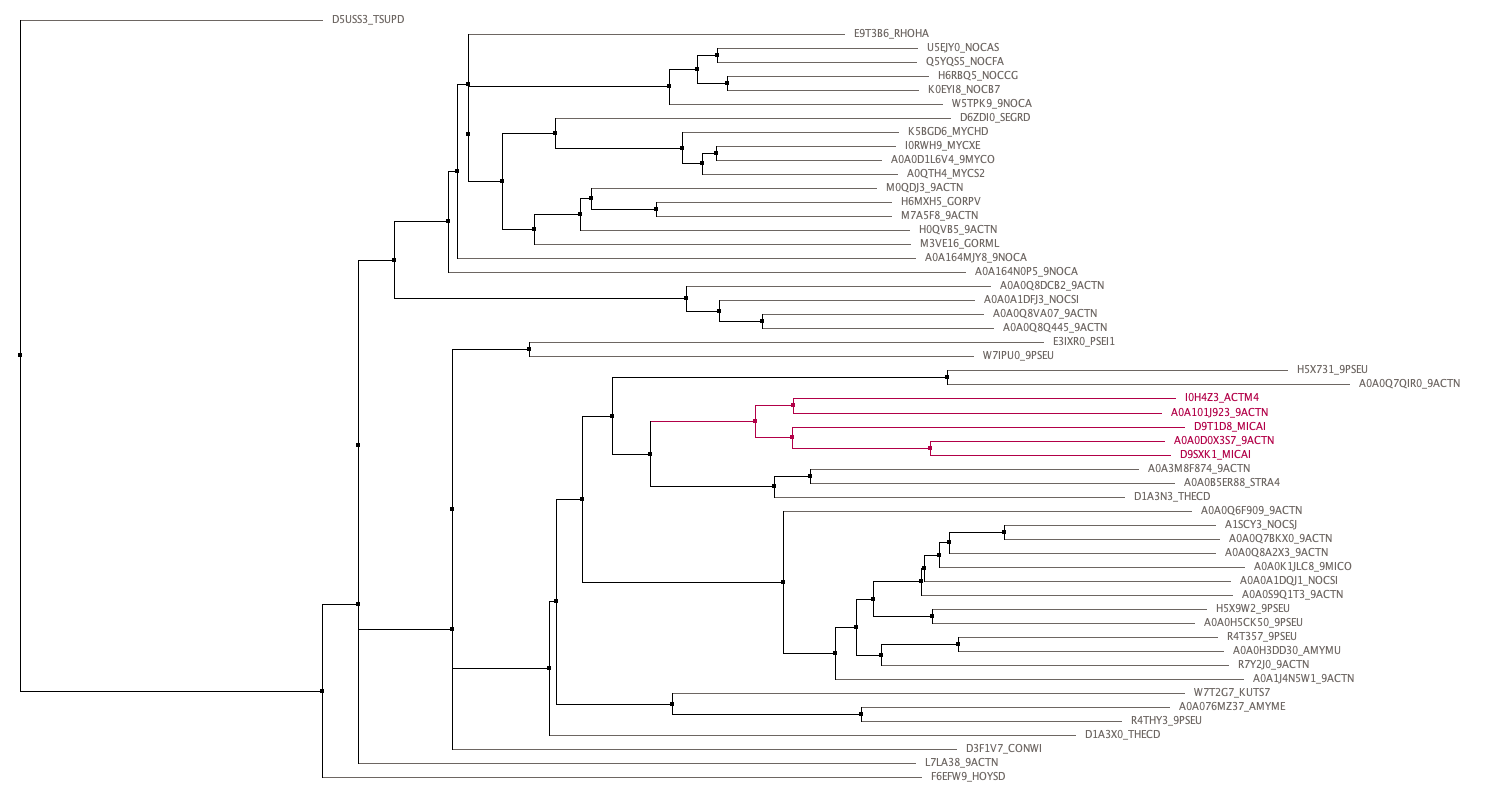 Рисунок 1. Филогенетическое дерево, построенное методом NJ в JalView. Красным цветом выделена клада, которую было решено отделить.
Рисунок 1. Филогенетическое дерево, построенное методом NJ в JalView. Красным цветом выделена клада, которую было решено отделить.
Выравнивание данной клады было открыто в отдельном окне. Далее было включено отображение цветом позиций с уровнем идентичности 100%. Для одного из мотивов был составлен паттерн.
Паттерн: LLP[YF]LTTR
После этого был проведен поиск этого мотива во всем выравнивании.
Этот мотив был обнаружен только в 5 последовательностях. Это именно те 5 последовательностей, которые относятся к выделенной кладе.
То есть данный мотив является специфичным для этой клады: он встречается во всех последовательностях клады и не встречается больше нигде в выравнивании.
PSI-BLAST
Для начала я выбрала AC белка, с которым мне предстояло дальше работать.
AC: B2V8C0
Организм: Sulfurihydrogenibium sp. (strain YO3AOP1)
Этот белок является ингибитором клеточного деления, он блокирует образование Z-колец, мешая полимеризации FtsZ.
Далее данный идентификатор я внесла с специальное окошко на сайте белкового BLAST, выбрала в качестве алгоритма PSI-BLAST и в качестве базы данных Swiss-Prot. Порог e-value = 0.005.
Далее было проведено несколько итераций поиска. Результаты приведены в таблице 1.
Таблица 1. Результаты поиска PSI-BLAST.
| Номер итерации |
Число находок выше порога (0,005) |
Идентификатор худшей находки выше порога |
E-value худшей находки |
Идентификатор лучшей находки ниже порога |
E-value лучшей находки |
| 1 |
163 |
Q88M41.2 |
0.004 |
Q4US07.1 |
0.006 |
| 2 |
188 |
Q9ZM51.1 |
6e-07 |
A7H8E6.1 |
0.038 |
| 3 |
188 |
Q9ZM51.1 |
2e-11 |
A7H8E6.1 |
0.025 |
На третьей итерации новые последовательности перестали добавляться, поэтому поиск был завершен.
При этом разница между худшей "правильной" и лучшей "неправильной" находокой по p-value большая. То есть вероятность, что находки составляют семейство гомологичных белков, большая, причем семейство хорошее.
MEME
Для начала я скачала выборку полных последовательностей, содержащих выбранный домен. Эти последовательности относятся к бактериям из рода Actinomadura.
В выборке всего 118 последовательностей.
Для данных последовательностей в JalView было построено выравнивание. Далее были удалены высокосходные последовательности (порог идентичности = 97%). В результате осталось 80 последовательностей в выборке.
После этого файл с оставшимися последовательностями был передан на вход MEME:
meme pf18556_reduced.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4
После параметра -o указывается имя директории, куда сохраняются результаты.
-mod anr указывает на то, что в одной последовательности может быть сколько угодно находок сигнала.
После -minw задается минимальная длина сигнала, после -maxw – максимальная. После -nmotifs задается число мотивов, которые будут в выдаче.
Результаты
Все четыре мотива имеют довольно маленькие значения e-value. На рисунке 2 представлено LOGO сигнала для находки с наилучшим e-value.
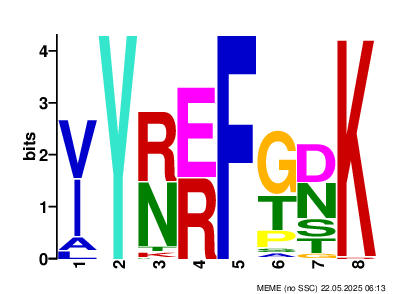 Рисунок 2. LOGO сигнала с наилучшим значением e-value.
Рисунок 2. LOGO сигнала с наилучшим значением e-value.
Представленность сайта GATC в геноме Sulfurimonas aquatica
Для оценки представленности сайта GATC в геноме Sulfurimonas aquatica была запущена следующая команда:
cbcalc -s sites.txt -o result_pr10_5 -M ~/term1/genome/GCF_017357825.1_ASM1735782v1_genomic.fna,
где sites.txt – файл со всеми сайтами длины 4, полученными перестановками без повторений букв A, T, G, C. Для создания данного файла я написала скрипт на Python.
Скрипт
Результат работы программы
По полученным данным посредством Excel была построена гистограмма контрастов (рисунок 3).
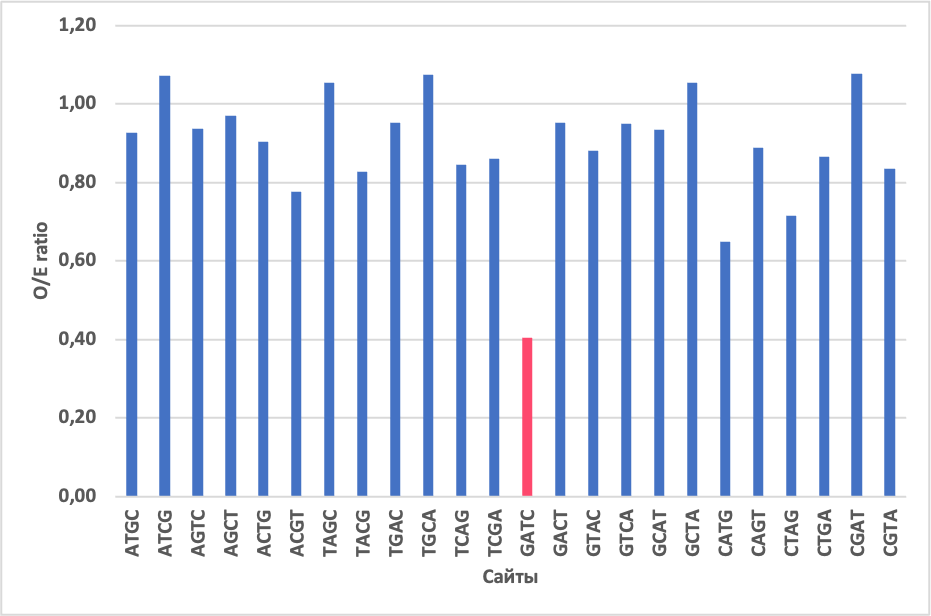 Рисунок 3. Гистограмма контрастов. Розовым цветом выделен GATC сайт.
Рисунок 3. Гистограмма контрастов. Розовым цветом выделен GATC сайт.
Можно заметить, что GATC сайт имеет самое маленькое значение O/E, то есть он представлен реже других сайтов.