Практикум 3
Реконструкция дерева по нуклеотидным последовательностям
В данном задании передо мной стояла цель реконструировать филогенетическое дерево по последовательностям малой РНК митохондриальных рибосом (12S рРНК).
Однако здесь возникла проблема: для большинства выбранных мной в первом практикуме организмов нет полных последовательностей митохондриального генома. Именно из-за этого некоторые из выбранных организмов я заменила близкородственными им (таким образом, чтобы они принадлежали одному роду). Однако и при таком раскладе не для всех организмов была последовательность митохондриального генома, и, к сожалению, некоторых животных пришлось убрать из рассмотрения.
Для оставшихся организмов были скачаны последовательности гена 12S рРНК.
Далее эти последовательности были выровнены при помощи muscle. После этого дерево было непосредственно реконструировано посредством программы FastME, модель p-distance. Из-за того, что последовательности нуклеотидные, была применена опция -d.
Дерево было визуализировано при помощи ITOL (рисунок 1, нижняя часть). Для некоторых организмов через пробел указано две мнемоники: первая – мнемоника близкородственного организма-"заменителя"; вторая – мнемоника организма из первого практикума.
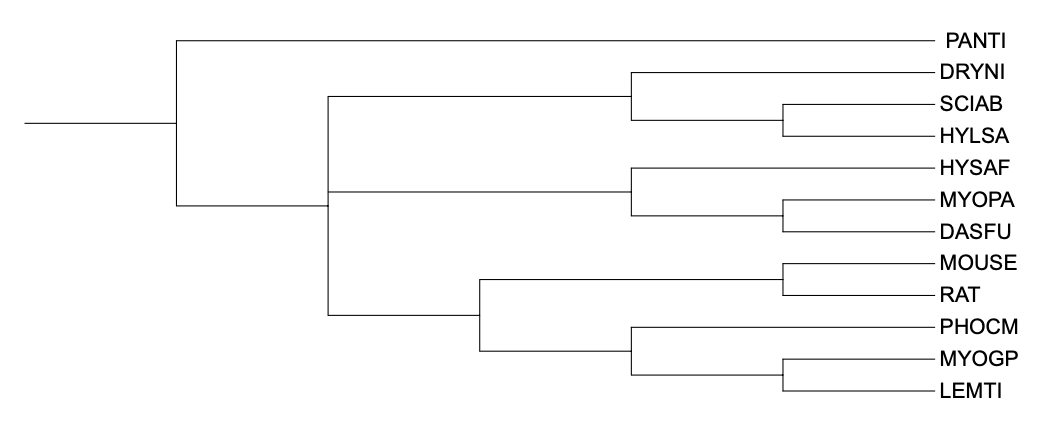
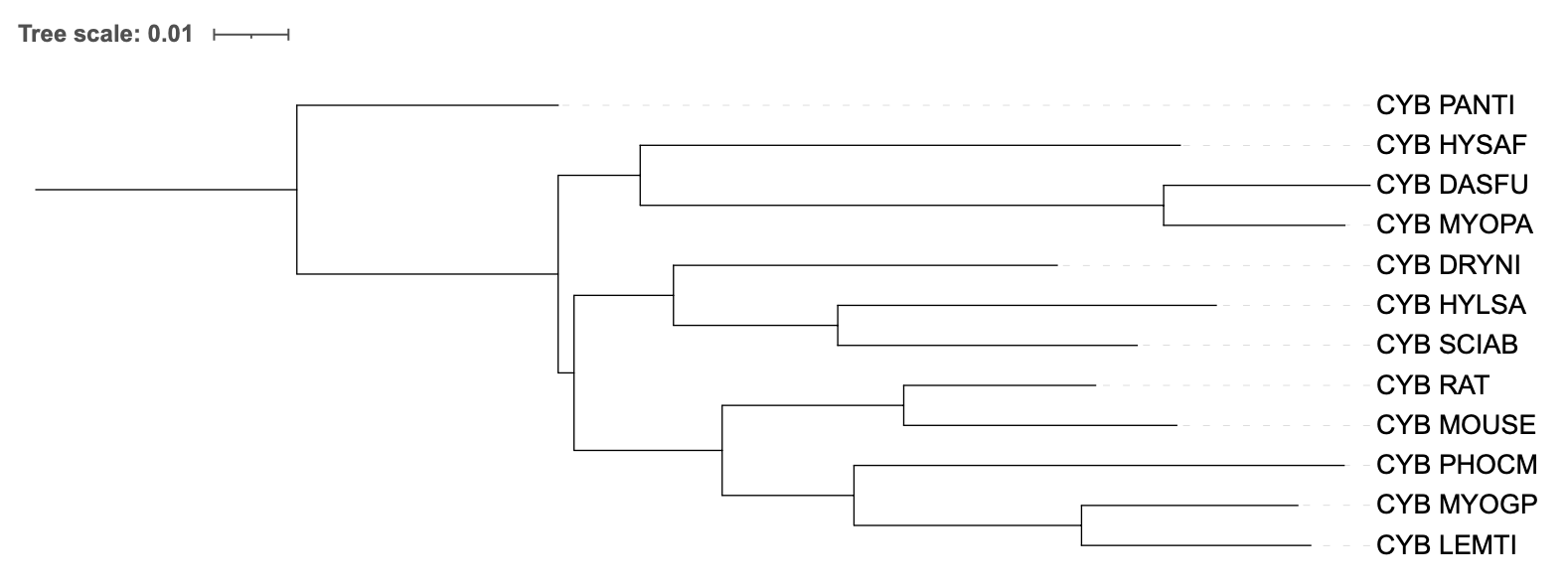
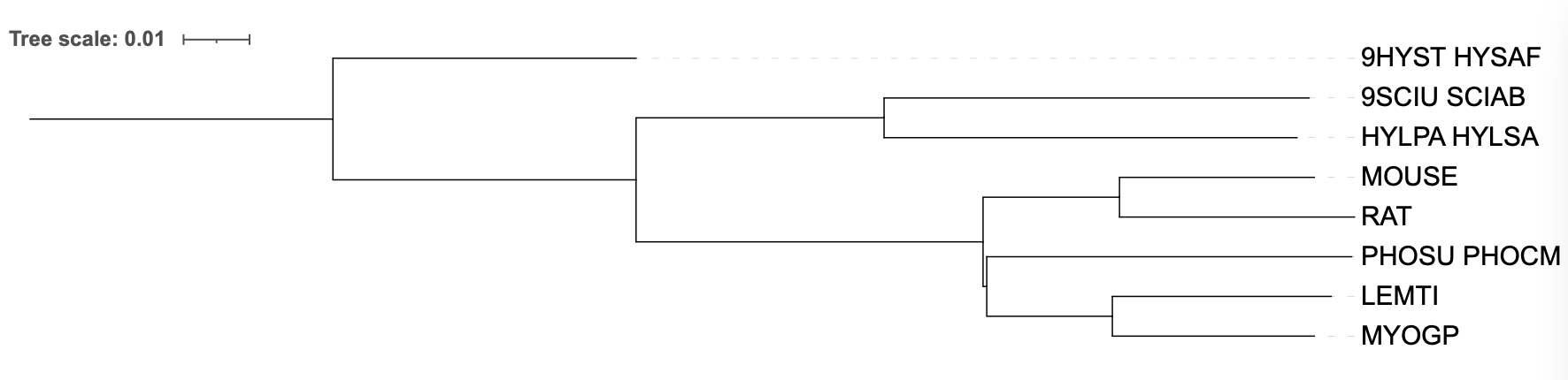 Рисунок 1. Филогенетическое дерево, построенное по таксономии (сверху), реконструированное программой FastMe (модель – p-distance) по последовательностям белка цитохрома В (в середине) и реконструированное по последовательностям 12S рРНК (снизу).
Рисунок 1. Филогенетическое дерево, построенное по таксономии (сверху), реконструированное программой FastMe (модель – p-distance) по последовательностям белка цитохрома В (в середине) и реконструированное по последовательностям 12S рРНК (снизу).
Можно заметить, что в целом топология деревьев схожа (если учесть, что некоторые организмы "выпали" из рассмотрения).
Укоренение во внешнюю группу
По сути это задание я проделала в предыдущем практикуме, добавив к выбранным организмам, которые являются грызунами, тигра.
Тигр относится к другому отряду Млекопитающих – Хищные.
Более подробно свое решение я аргументировала в предыдущем практикуме, здесь я лишь продублирую одно из получившихся реконструированных деревьев (рисунок 2).

 Рисунок 2. Филогенетическое дерево с добавлением внешней группы, построенное по таксономии (сверху) и реконструированное по последовательностям цитохрома B программой FastME (модель – p-distance) (снизу).
Рисунок 2. Филогенетическое дерево с добавлением внешней группы, построенное по таксономии (сверху) и реконструированное по последовательностям цитохрома B программой FastME (модель – p-distance) (снизу).
Дерево было соответственно укоренено в ветвь, ведущую к внешней группе. То есть добавление внешней группы позволяет нам относительно легко определить ветвь, в которую надо укоренять.
В целом состав основных ветвей и положение организмов внутри них остались прежними по сравнению с деревом, построенном на таксономии. Однако можно заметить, что был разрешен один из узлов.
Бутстреп
Я решила добавить 100 бутстреп-реплик для филогенетического дерева, реконструированного по последовательностям цитохрома B
Для этого я воспользовалась следующей командой:
fastme -b 100 -pm -i cyb.phy -o tree2
Полученное дерево я визуализировала при помощи ITOL (рисунок 3).
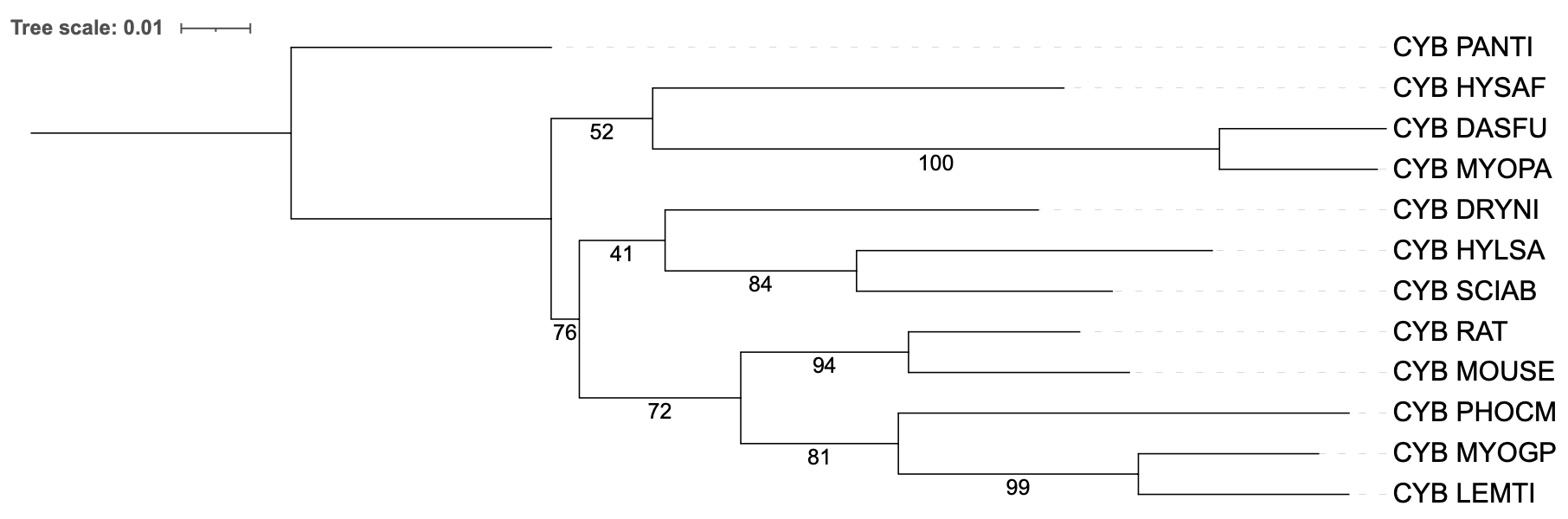 Рисунок 3. Филогенетическая реконструкция программой FastMe (модель – MtREV) c использованием 100 реплик бутстрепа.
Рисунок 3. Филогенетическая реконструкция программой FastMe (модель – MtREV) c использованием 100 реплик бутстрепа.
Можно заметить, что ветви, которые в предыдущем практикуме реконструировались при любом методе реконструкции, имеют высокий уровень достоверности.
Однако также в предыдущем практикуме мы видели, что разные программы разрешают неразрешенный узел разными способами – именно в это месте мы видим "сомнительные" значения (низкий уровень поддержки).