Практикум 4
Составление списка гомологичных белков
Для начала были выбраны полные протеомы 8 бактерий отдела Pseudomonadota. Для удобства я скопировала выбранные протеомы к себе в директорию при помощи следующей команды:
cp AROAE.fasta BORPE.fasta BRUSU.fasta HAEIN.fasta NEIMA.fasta PASMU.fasta RHIME.fasta YERPE.fasta
Далее я объединила выбранные протеомы в один файл:
cat * > prot.fasta
Следующим этапом я создала белковую базу посредством следующей команды:
makeblastdb -in prot.fasta -dbtype prot -out protdb.fasta
После этого был произведен поиск гомологов белка CLPX_ECOLI в выбранных протеомах при помощи программы blastp (порог e-value = 0,0001):
blastp -task blastp -query P0A6H1.fasta -db protdb.fasta -out result -evalue 0.0001
Список находок без выравниваний и заголовка выдачи
Реконструкция и визуализация
Последовательности находок были помещены в fasta-файл. Названия были отредактированы таким образом, чтобы остались только ID.
Файл с последовательностями находок
Далее данные последовательности были выровнены при помощи следующей команды:
muscle -align spisok.fasta -output spisok-alignment.fasta
Далее полученное выравнивание было переведено в формат phylip-relaxed при помощи пакета BioPython.
После этого была произведена реконструкция дерева при помощи программы IQ-Tree (параметры по умолчанию):
iqtree -s spisok.phy
Файл с формулой Newick
Далее я визуализировала данное дерево сервисом ITOL и укоренила в среднюю точку.
Ортологи: CLPX_RHIME и CLPX_BRUSU, CLPX_AROAE и CLPX_BORPE, HSLU_PASMU и HSLU_HAEIN.
Паралоги: HSLU_BORPE и CLPX_BORPE, HSLU_BRUSU и CLPX_BRUSU, HSLU_PASMU и CLPX_PASMU.
В начале я раскрасила разные ортологические группы (наборы попарно ортологичных белков) в разные цвета (рисунок 1).
При этом группы, которые содержат три и менее последовательностей, я оставила черными.
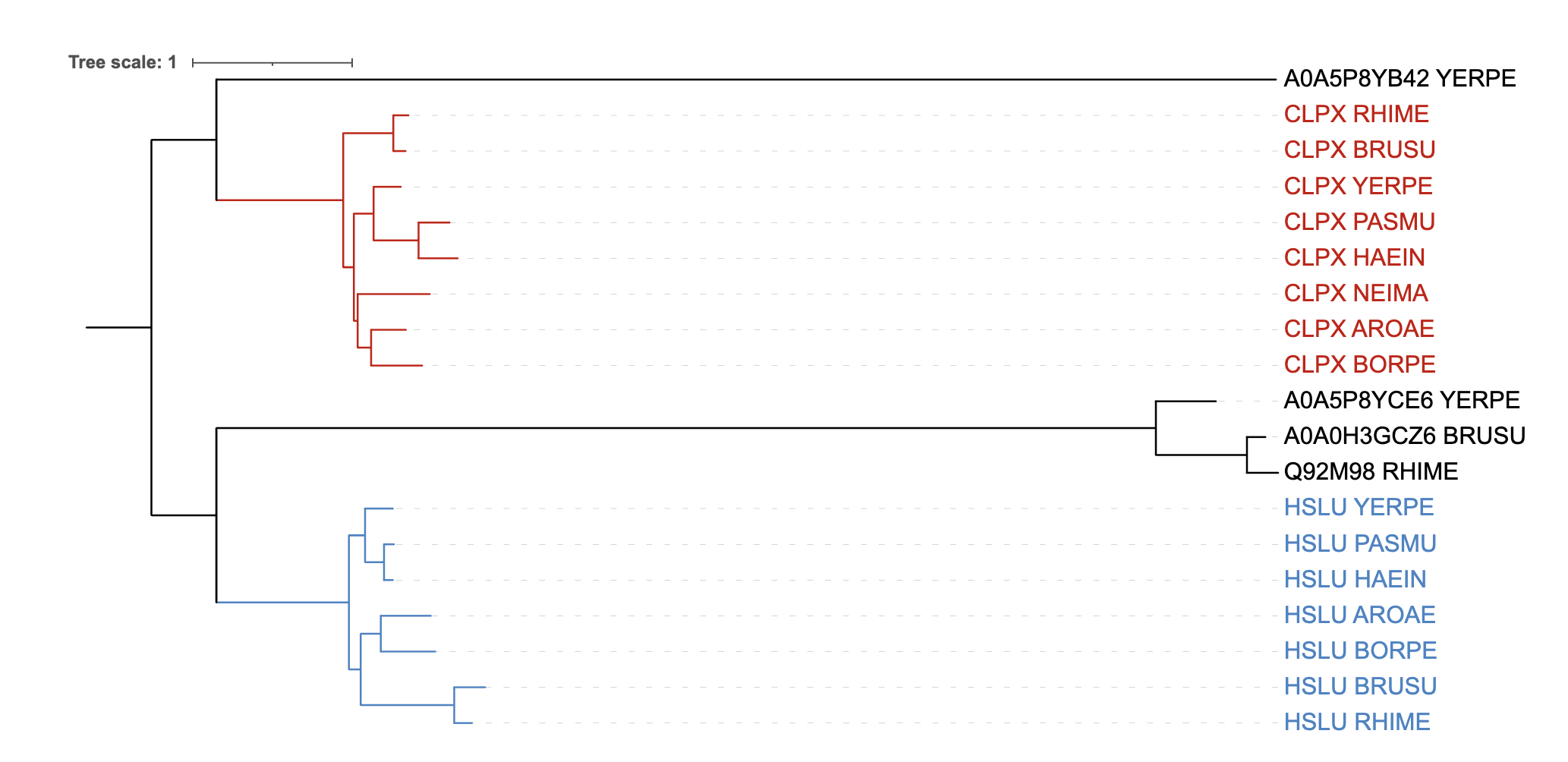 Рисунок 1. Филогенетическая реконструкция программой IQ-Tree. Разными цветами показаны разные ортологические группы.
Рисунок 1. Филогенетическая реконструкция программой IQ-Tree. Разными цветами показаны разные ортологические группы.
После этого все ортологические группы, содержащие больше трех последовательностей, были "схлопнуты" (рисунок 2).
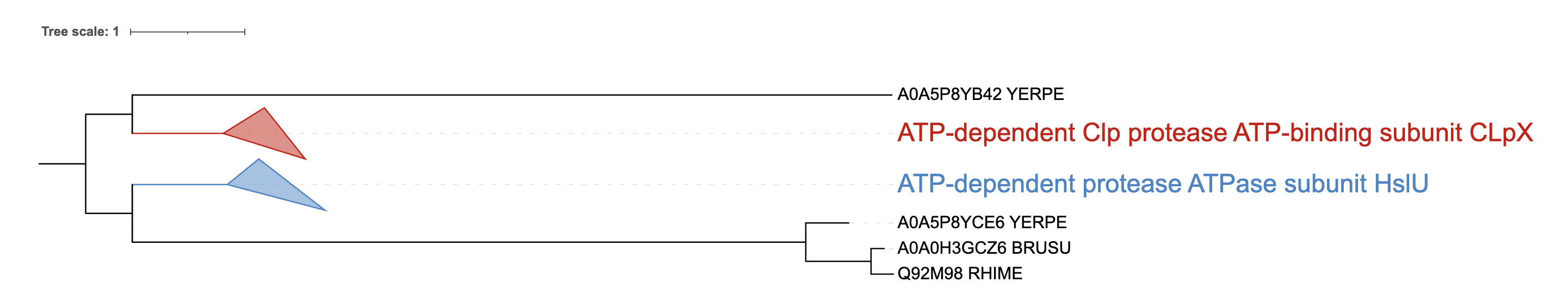 Рисунок 2. Изображение реконструированного дерева с "схлопнутыми" ортологическими группами.
Рисунок 2. Изображение реконструированного дерева с "схлопнутыми" ортологическими группами.
Ортологическая группа, окрашенная красным цветом, содержит белки из всех 8 выбранных бактерий. При этом реконструированная филогения данного белка (CLPX) соответствует филогении бактерий.
Ортологическая группа, окрашенная голубым цветом, содержит белки из 7 бактерий (всего – 8). Реконструированная филогения данного белка (HSLU) лишь частично соответствует филогении бактерий: на реконструированном дереве клада (HSLU_AROAE,HSLU_BORPE) более сближена с кладой (HSLU_BRUSU,HSLU_RHIME) по сравнению с кладой (HSLU_YERPE,(HSLU_PASMU,HSLU_HAEIN)).
Однако если мы рассмотрим филогению бактерий (рисунок 3), то увидим, что клада (AROAE,BORPE) более близка к кладе (YERPE,(PASMU,HAEIN)) по сравнению с кладой (BRUSU,RHIME).
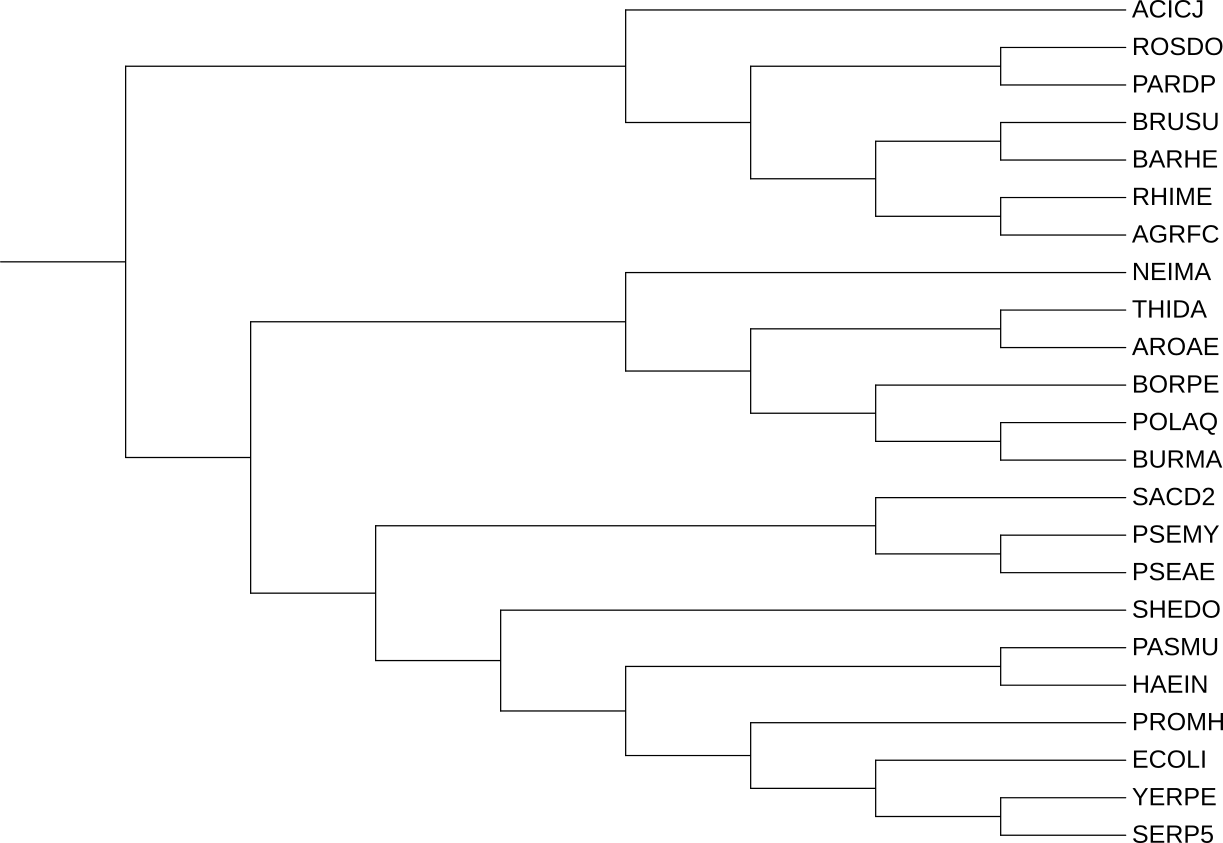 Рисунок 3. Филогения бактерий, которую мы считаем верной при выполнении данного практикума.
Рисунок 3. Филогения бактерий, которую мы считаем верной при выполнении данного практикума.