Практикум 6
Базы данных
Для выполнения данного практикума мне был предоставлен список ID генов человека.
Список
В данном списке 128 ID генов.
Вероятно, эти гены как-то между собой связаны раз оказались в одном списке. Однако список довольно большой, и с ходу что-то сказать об этих генах непросто.
Целью данного практикума было проанализировать данный список при помощи различных сервисов.
GO
Для начала я решила проанализировать список при помощи базы данных GO (Gene Ontology). Эта база данных представляет собой граф биологических терминов.
Эта база данных позволяет понять, в каких процессах участвуют белки, закодированных в этих генах.
Для начала я загрузила список ID и выставила параметры: использование точного теста Фишера и использование поправки Бонферонни на множественное тестирование. В результате была получена таблица находок, которую я отсортировала по p-value с поправкой Бонферонни. Всего таблица содержит 117 терминов. Таблица получилась слишком большой, поэтому полностью ее можно посмотреть по ссылке:
Таблица с выдачей
Наиболее значимые находки привожу ниже (рисунок 1).
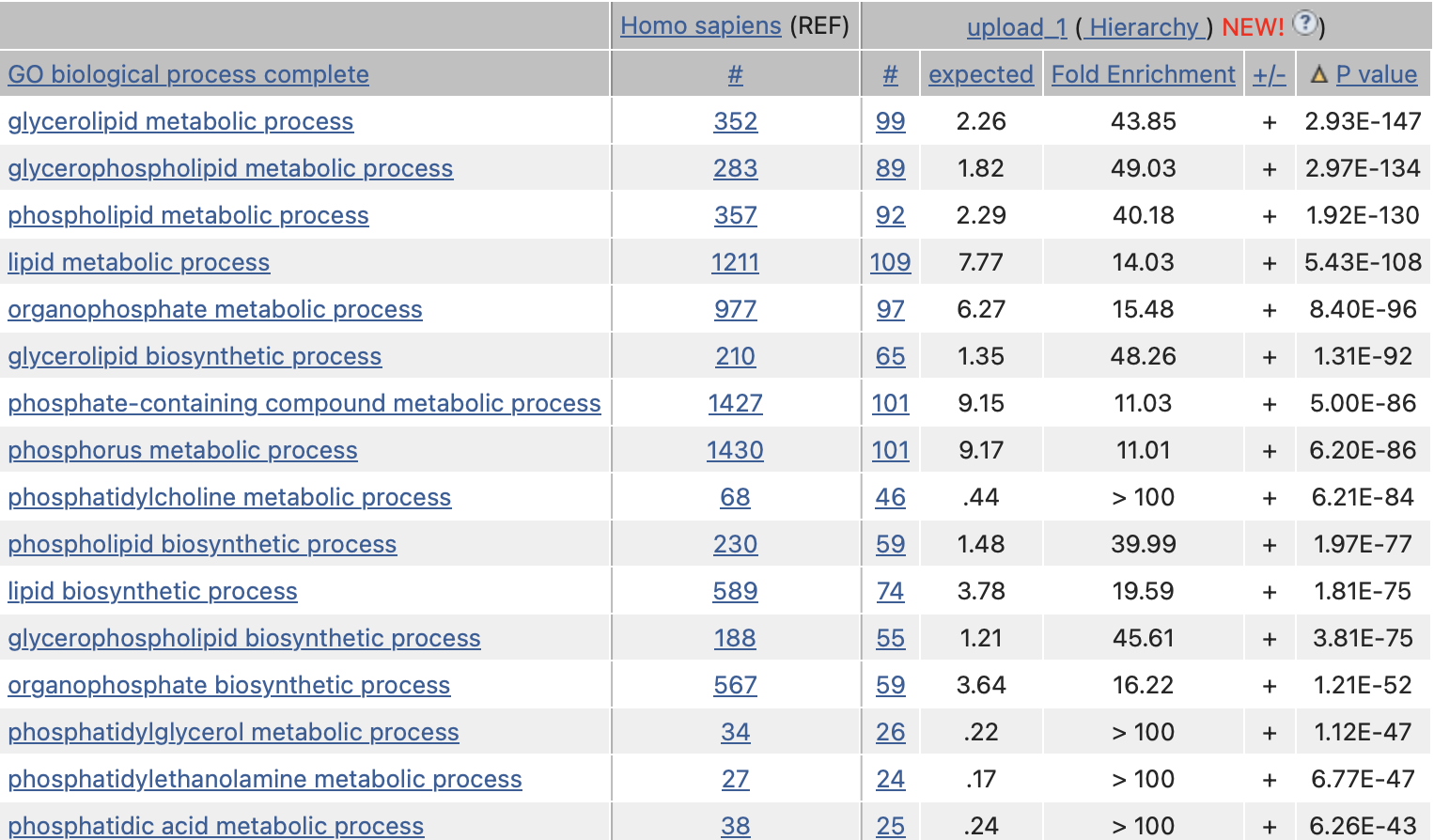 Рисунок 1. Наиболее значимые находки в выдаче.
Рисунок 1. Наиболее значимые находки в выдаче.
Можно заметить, что данные гены задействованы в таких биологических процессах, как метаболизм фосфолипидов (в том числе глицерофосфолипидов), их биосинтез.
Стоит отметить, что веб-интерфейс довольно удобный, было интуитивно понятно, как загружать список ID и выставлять параметры. Результат выдается практически мгновеено. В таблице отражены наиболее важные столбцы, можно выбрать сортировку по разным столбцам (в нашем случае по p-value). Все биологические процессы (первый столбец) "кликабельные". Так мы, например можем узнать, что имеется в виду под тем или иным термином, а также какие еще гены участвуют в этом процессе.
The Human Protein Atlas
Еще на занятии меня довольно-таки впечатлила данная программа, поэтому я решила попробовать поработать с ней. Она позволяет получить информацию об экспрессии РНК и белка, причем в довольно понятном интерактивном виде.
На вход данной программе передается ID гена. Список у меня очень большой, ID же генов в большинстве своем мне мало о чем говорят (можно только догадываться). Поэтому мой выбор ID практически никак не обоснован: я остановилась в случайном месте списка и выбрала ID LPIN2, который показался мне знакомым (видимо, я натыкалась на него когда-то при выполнении предыдущих практикумов).
Итак, в результате работы программы стало понятно, что это ген белка липина-2. Этот белок относится к ферментам, участвующим в метаболических путях. А также этот белок связан с человеческими заболеваниями и является возможной мишенью для лекарств.
Также в выдаче представлено описание функции белка из Uniprot. Если кратко, этот белок играет важную роль в контроле метаболизма жирных кислот. А если более подродбно, то он катализирует реакцию превращения фосфатидной кислоты в диацилглицерол через триглицериды, а также реакции биосинтеза фосфатидилхолина и фосфатидилэтаноламина.
Этот фермент относится к классу гидролаз.
Также данная программа позволяет узнать информацию и локализации экспрессии белка.
Данный белок имеет внутриклеточную локализацию, он экспрессируется в цитоплазме в различных тканях, однако повышенная экспрессия наблюдается в печени и желудочно-кишечном тракте. Также высокий уровень экспрессии белка наблюдается в почках и плаценте (рисунок 2)
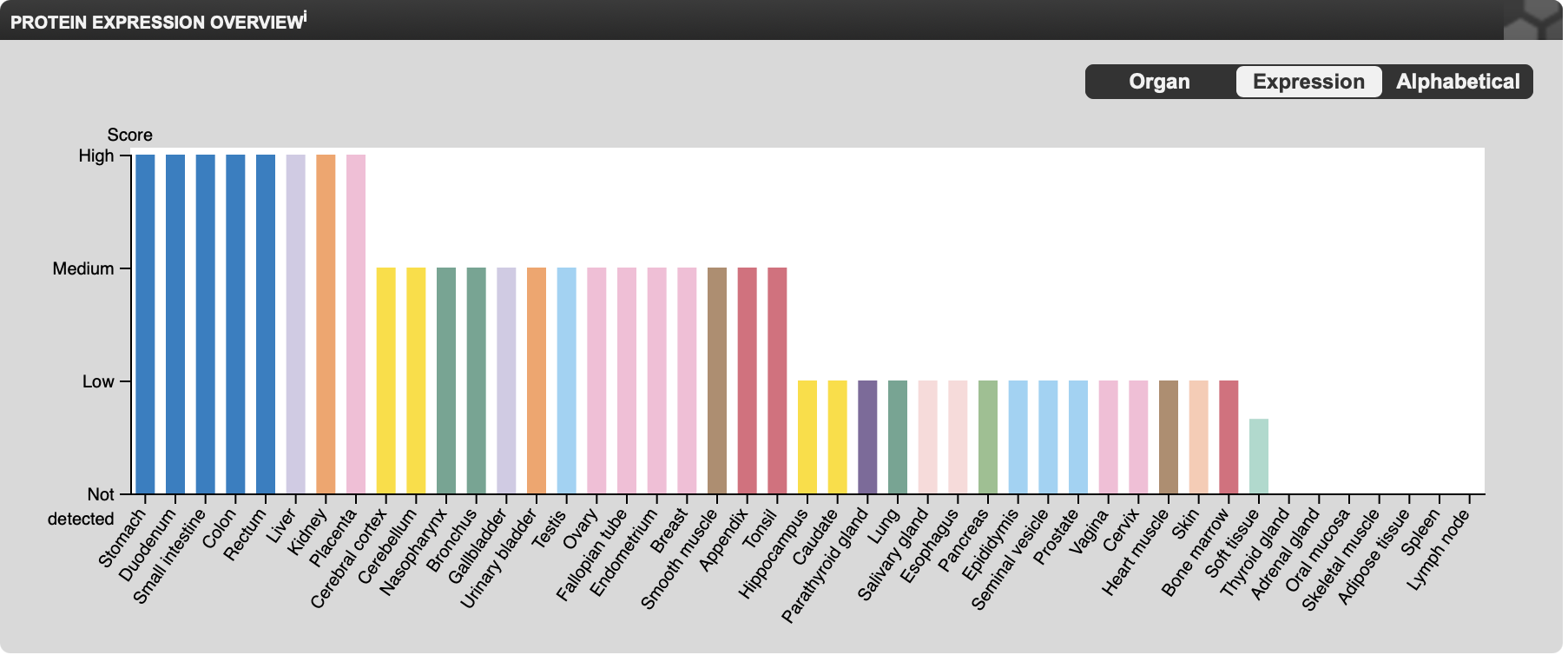 Рисунок 2. Уровни экспрессии белка липина-2 в различных тканях. По горизонтали отложены различные ткани (органы). По вертикали разные уровни экспрессии белка.
Рисунок 2. Уровни экспрессии белка липина-2 в различных тканях. По горизонтали отложены различные ткани (органы). По вертикали разные уровни экспрессии белка.
Также можно пронаблюдать за локализацией экспрессии РНК. Уровень экспрессии РНК в печени сильно преобладает над экспрессией в других тканях (рисунок 3).
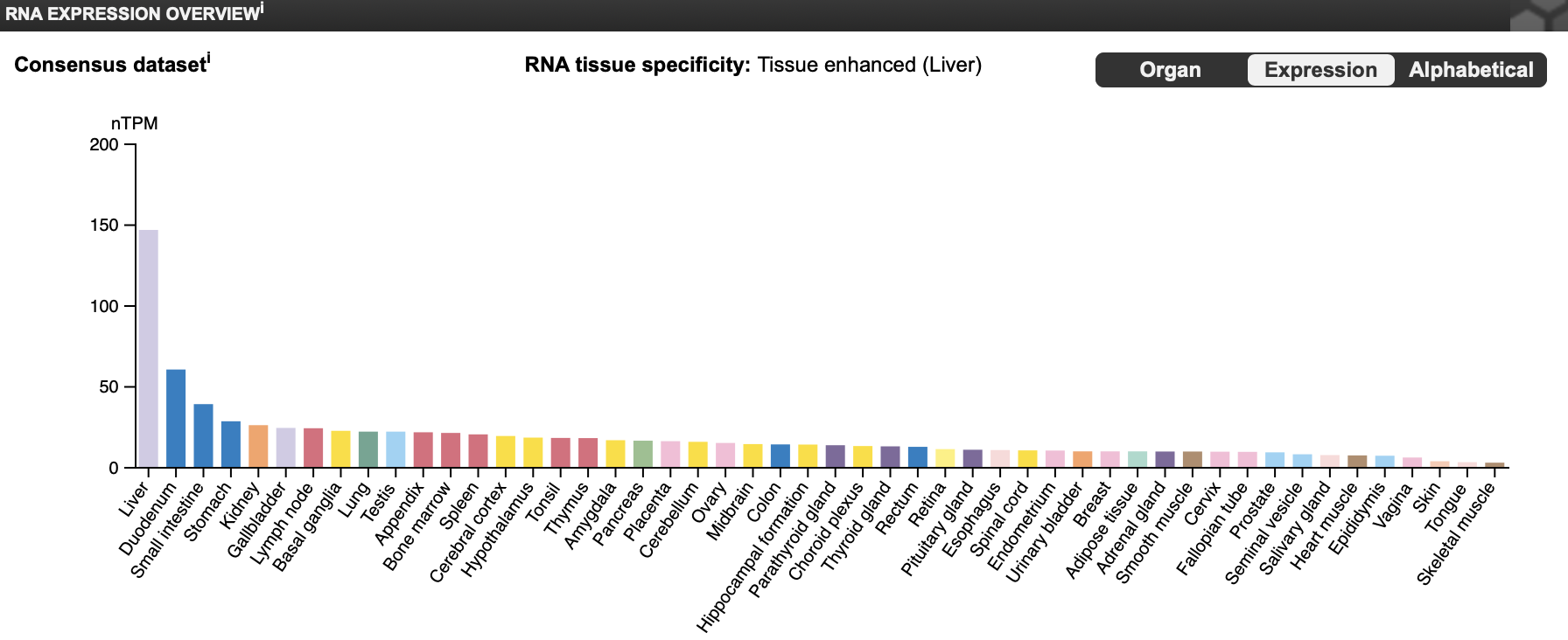 Рисунок 3. Уровни экспрессии РНК в различных тканях. По горизонтали отложены различные ткани (органы). По вертикали уровень экспрессии (мера - nTPM (transcripts per million)).
Рисунок 3. Уровни экспрессии РНК в различных тканях. По горизонтали отложены различные ткани (органы). По вертикали уровень экспрессии (мера - nTPM (transcripts per million)).
Также из выдачи можно узнать, что LPIN2 является маркером для прогнозирования так называемых опухолей головы и шеи (к ним, например, относятся опухоли языка, глотки, слюнных желез и т.д.) и светлоклеточного рака почки.
Для краткого обобщения экспрессии РНК и белка используется анатограмма (рисунок 3)
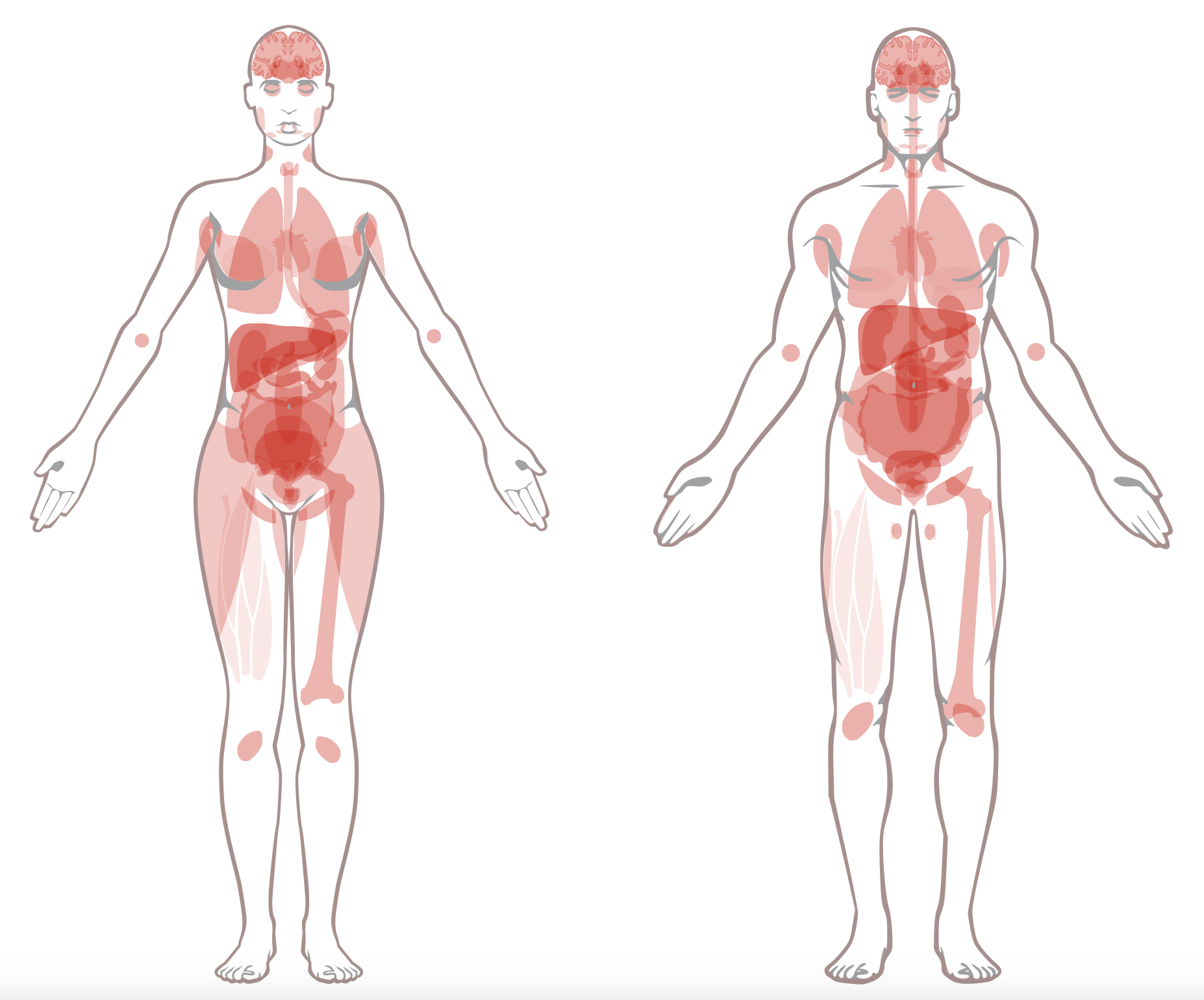 Рисунок 3. Уровни экспрессии РНК и белка в различных тканях в виде анатограммы - "карты" человеческого организма (слева – женщина, справа – мужчина), где красным цветом отмечены области (ткани), где наблюдается экспрессия. При этом уровень экспрессии отображается насыщенностью цвета.
Рисунок 3. Уровни экспрессии РНК и белка в различных тканях в виде анатограммы - "карты" человеческого организма (слева – женщина, справа – мужчина), где красным цветом отмечены области (ткани), где наблюдается экспрессия. При этом уровень экспрессии отображается насыщенностью цвета.
Более того, в данной базе данных можно найти большое количество очень красивых изображений. Например, при упоминании различных тканей часто рядом прикрепляется изображение её гистологического среза. Ещё можно встретить изображения окрашенных клеток под микроскопом - в общем, очень визуально приятно.
Мне очень понравилось работать с этой базой данных. Веб-интерфейс очень удобный, все понятно (а даже если непонятно – везде есть выплывающие подробные объяснения). Информация структурированная. Но что самое главное, на мой взгляд, информация подкреплена визуально в виде различных графиков, гистограмм, таблиц, изображений, что значительно упрощает восприятие новой информации.
Общие выводы
Я проанализировала выданный мне список ID генов при помощи GO. В результате стало понятно, что список генов неслучаен: все они связаны с метаболизмом и биосинтезом фосфолипидов. То есть данная база данных дала общее представление о взяимосвязи этих генов.
Далее они из генов был проанализировал в The Human Protein Atlas. В итоге я узнала локализацию экспресии РНК и белка, обнаружила некоторые интересные факты, получила эстетическое удовольствие.
В целом, обе базы очень информативны и удобны в использовании.