Практикум 8
Сигналы в геноме
Описание сигнала
Целью данного практикума является исследовать один из известных сигналов генома.
В качестве такого сигнала я выбрала G-квадруплексы, располагающиеся в промоторах генов и регулирующие транскрипцию у прокариот и эукариот.
G-квадруплексы (G4) относятся к неканоническим вторичным структурам ДНК. Они формируются в последовательностях, богатых гуанином [1].
G-квадруплексы могут иметь различную топологию в зависимости от ориентации цепей ДНК (рисунок 1).
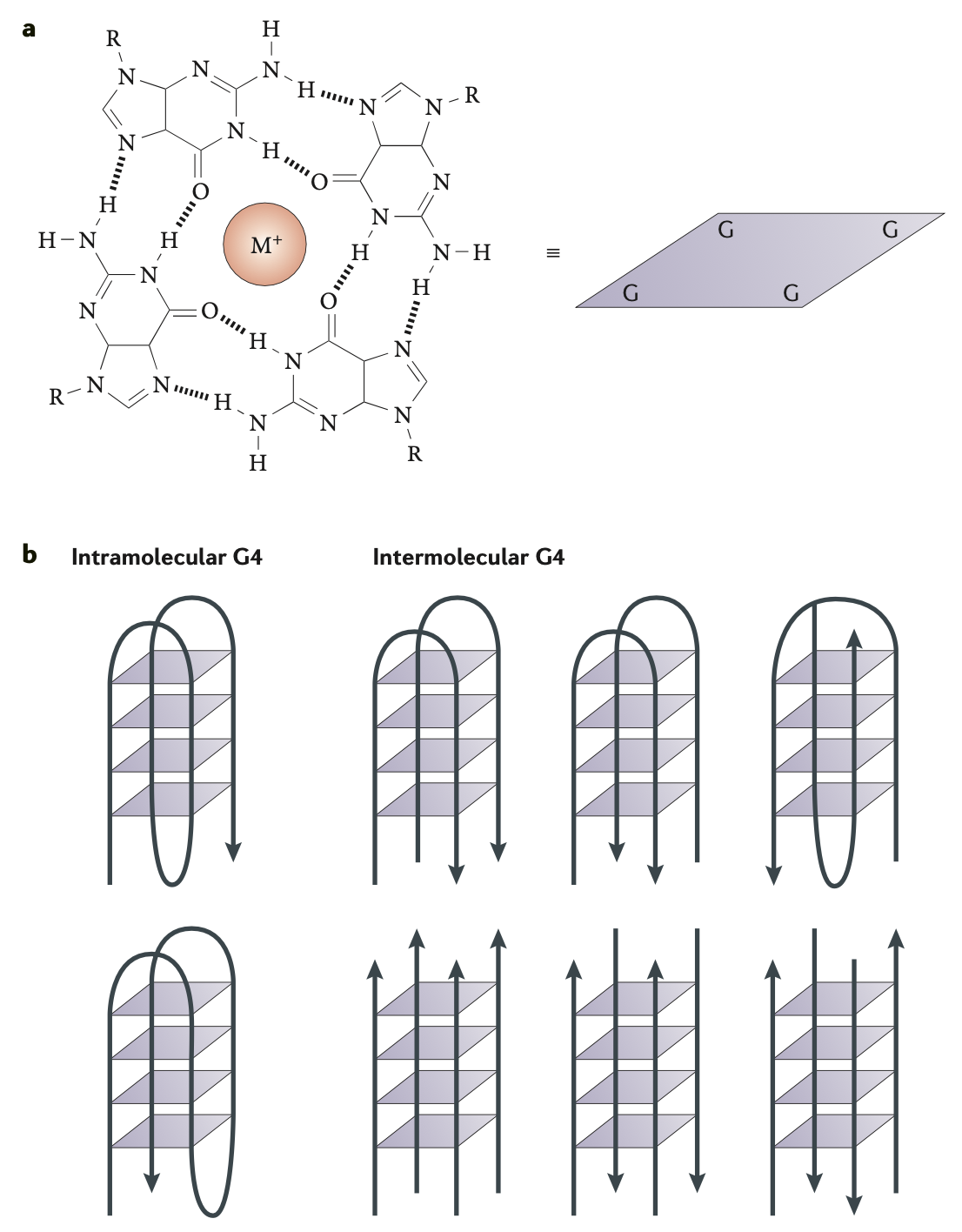 Рисунок 1. a. Взаимодействия в G-квартете; b. Различные топологии G-квадруплекса [1].
Рисунок 1. a. Взаимодействия в G-квартете; b. Различные топологии G-квадруплекса [1].
Ранее предполагалось, что G-квадруплексы могут в равной степени влиять на транскрипцию как положительно, так и отрицательно. Например, это могло бы зависеть от того, на какой цепи располагается G4-мотив [1]. Однако по более свежим данным, полученным при помощи метода ChIP-Seq, было показано, что G-квадруплексы в основном располагаются в промоторных областях активно транскрибируемых генов и что они, скорее, положительно влияют на экспрессию генов [2]. Таким образом, G-квадруплексы даже начали рассматривать как потенциальные терапевтические мишени.
G-квадруплексы могут связываться с транскрипционными факторами и другими белками, принимающими участие в транскрипции.
Сила сигнала может зависеть от стабильности G-квадруплексов, которая в свою очередь зависит от многих факторов, например, от общей длины G4-мотива, размера петель между гуанинами и связанных катионов [1].
Например, регуляцию транскрипции путем G-квадруплекса в промоторной области можно встретить у млекопитающих в промоторе онкогена MYC [1].
Сервисы для поиска G-квадруплексов
Примером сервиса для поиска G-квадруплексов является QGRS Mapper.
Ссылка на сервис
Метод, который используется для работы данного сервиса, основан на следующих принципах: более короткие петли встречаются чаще, чем более длинные; G-квадруплексы, как правило, имеют петли одинакового размера; чем больше количество G-квартетов, тем стабильнее квадруплекс [3].
Сервис принимает на вход нуклеотидную последовательность (можно ввести в специальное окно на сайте или загрузить в формате fasta). Также нуклеотидные последовательности можно получать из баз данных по идентификатору.
В результате работы сервиса мы получаем некоторую информацию о нашей последовательности, в том числе количество G-квадруплексов.
Также полученные данные визуализированы, цветом указаны участки, для которых предсказано наличие G-квадруплексов.
Я решила опробовать работу этого сервиса и ввела туда последовательность c-MYC промотора. В результате был обнаружен G-квадруплекс (рисунок 2).
 Рисунок 2. Результат работы сервиса QGRS Mapper. Цветом покрашена последовательность предполагаемого G-квадруплекса.
Рисунок 2. Результат работы сервиса QGRS Mapper. Цветом покрашена последовательность предполагаемого G-квадруплекса.
Используемые источники:
1) Bochman M.L., Paeschke K., Zakian V.A., 2012. DNA secondary structures: stability and function of G-quadruplex structures // Nature Reviews Genetics. V. 13. № 11. P. 770-780.
2) Robinson J., Raguseo F., Nuccio S.P., Liano D., Di Antonio M., 2021. DNA G-quadruplex structures: more than simple roadblocks to transcription? // Nucleic Acids Research. V. 49. № 15. P. 8419-8431.
3) Kikin O., D'Antonio L., Bagga P.S., 2006. QGRS Mapper: a web-based server for predicting G-quadruplexes in nucleotide sequences. // Nucleic Acids Research. V. 34. P. 676-682.