Практикум 9
Поиск de novo сигналов в ДНК
В первом семестре я писала мини-обзор генома бактерии Sulfurimonas aquatica.
Целью данного практикума является найти основные сигналы, связанные с инициацией транскрипции/трансляции, в геноме данной бактерии.
Для выполнения данного задания я с разрешения воспользовалась скриптом моего однокурсника Масленникова Вячеслава.
Скрипт
Данный скрипт принимает на вход два файла: непосредственно геном бактерии и файл с аннотацией генома.
Далее скрипт ищет участки, в состав которых предположительно входит последовательность Шайна-Дальгарно. Эта последовательность является сигналом старта трансляции у прокариот, являясь сайтом посадки рибосомы на мРНК.
В результате работы скрипта получается 3 файла с различными наборами последовательностей.
Группа положительного контроля: скрипт вырезает из всех генов белков участки размером в 25 нуклеотидов, располагающиеся до старт-кодона. В данных последовательностях мы хотим найти последовательность Шайна-Дальгарно (она располагается примерно за 10 нуклеотидов до старт-кодона).
Группа отрицательного контроля: в эту группу попадают участки размером в 25 нуклеотидов, располагающиеся после старт-кодона.
Тренировочная группа: в эту группу входят промоторы генов, в которых вероятно встретится последовательность Шайна-Дальгарно (наиболее консервативные гены). На этом файле впоследствии обучается MEME.
Также хочу отметить, что этот скрипт хорош тем, что можно с легкостью изменить координаты вырезаемых участков. Таким образом, этот скрипт можно модифицировать для поиска каких-то других интересующих последовательностей.
Итак, я запустила этот скрипт для генома Sulfurimonas aquatica и получила соответствующие три файла:
Группа положительного контроля
Группа отрицательного контроля
Тренировочная группа
MEME
Далее я запустила следующую команду:
meme TRAIN.fasta -dna -nmotifs 1 -minw 5 -maxw 10,
где -dna обозначает использование буквенного обозначения, характерного для ДНК, -nmotifs число мотивов, которое необходимо выводить в результате работы программы, -minw и -maxw задают минимальную и максимальную длину мотива соответственно.
Полная выдача
p-value находки достаточно маленький, то есть она является статистически значимой. Ее LOGO представлено на рисунке 1.
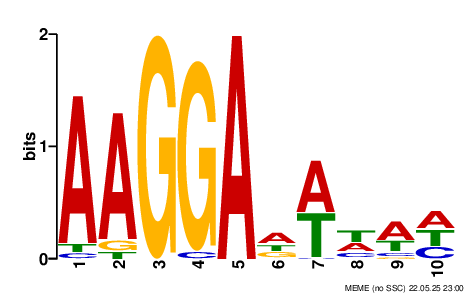 Рисунок 1. LOGO находки.
Рисунок 1. LOGO находки.
Консенсусом для последовательности Шайна-Дальгарно является AGGAGG. Однако существуют разные вариации последовательности Шайна-Дальгарно, одной из которых как раз является AAGGA. Таким образом, можно предположить, что мы нашли последовательность Шайна-Дальгарно для последовательностей тренировочной группы. Теперь можно поискать полученный мотив в последовательностях двух других групп (положительного и отрицательного контроля).
FIMO
Я запустила следующую команду:
fimo --oc fimo_pos -thresh 0.001 meme_out/meme.txt POSITIVE.fasta,
где --thresh задает порог p-value для отображения результатов.
Выдача
Аналогичную команду я запустила для группы отрицательного контроля:
fimo --oc fimo_neg -thresh 0.001 meme_out/meme.txt NEGATIVE.fasta
Выдача
В первом случае при пороге p-value 0,001 нашлось 726 находок, во втором случае – 127. То есть в первой группе находок значительно больше, что позволяет предположить, что найденный мотив является последовательностью Шайна-Дальгарно для Sulfurimonas aquatica.