Трансмембранные белки
База данных OPM
Для работы был выбран белок Rhodopsin I (5ax0). Толщина гидрофобного слоя составляет 32.4 ± 2.2 Å, согласно базе данных OPM, похожие значения были получены с помощью JMol: 33.5 Å. Как и все другие родопсины, рассматриваемый белок имеет 7 гидрофобных спиралей, проходящих через мембрану. Номера остатков, формирующих эти трансмембранные спирали имеют следующие номера: 12-36, 42-64, 84-103, 109-131, 136-157, 174-197, 204-227. Среднее количество аминокислотных остатков, входящих в трансмембранную спираль, составляет 22. Белок находится в в плазматической мембране эукариотической клетки (организм – Acetabularia acetabulum). В Таблице 1 системно собраны вышеприведенные параметры (для 5ax0), а также данные для ещё одного белка 4ctd, образующего бета-бочонок.
| 5ax0 (All alpha) | 4ctd (Beta-barrel) | |
|---|---|---|
| Толщина гидрофобного слоя (OPM) | 32.4 ± 2.2 Å | 24.6 ± 1.3 Å |
| Толщина гидрофобного слоя (Jmol) | 33.5 Å | 25.5 Å |
| Координаты трансмембранных спиралей | 12-36, 42-64, 84-103, 109-131, 136-157, 174-197, 204-227 | 6- 16, 31- 39, 43- 50, 68- 78, 85- 92, 111- 120, 128- 136, 151- 160, 167- 174, 192- 201, 204- 214, 223- 230, 237- 244, 259- 268 |
| Среднее количество остатков в трансмембранном участке | 22 | 8,4 |
| В какой мембране находится белок | Периплазматическая мембрана эукариотической клетки | Внешняя мембрана грам-отрицательной бактерии |
| Рисунок | 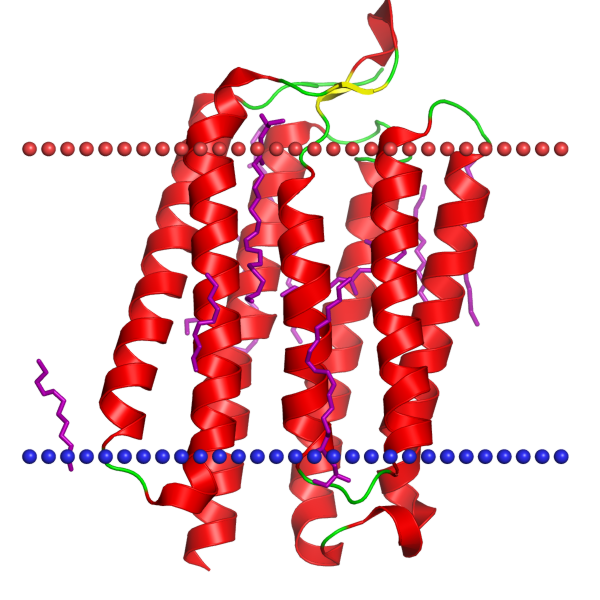 | 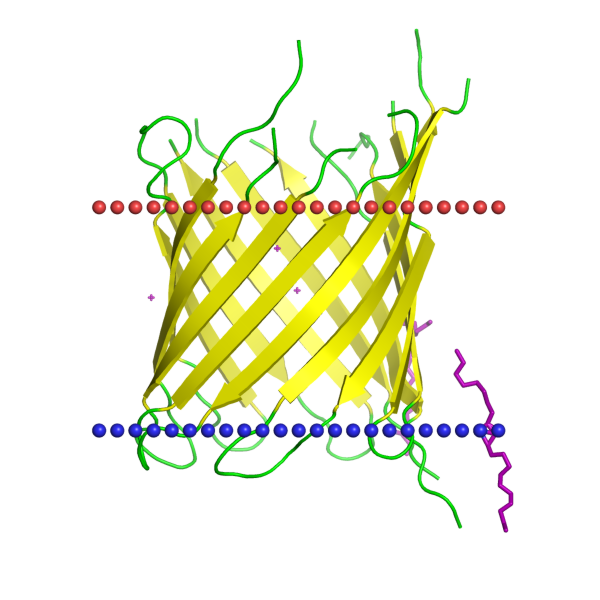 |
Таблица 1. Данные о белках 5ax0 и 4ctd.
Анализ предсказания трансмембранных спиралей
Даже беглого взгляда понятно (Рис. 1 и 2), чтобы понять, что выдачи TMHMM и Phobius практически полностью совпадают между собой (если посмотреть текстовый отчет, заметны различия в несколько аминокислотных остатков для предсказанных участков). Скорее всего, это связано с тем, что родопсин не является "хитрым" трансмембранным белком, и представляет собой 7 трансмембранных альфа-спиралей. Относительно настоящих данных, ни TMHMM, ни Phobius не "придумали" и не "потеряли" никаких структурных элементов белка. Ближе к реальной оказалась выдача Phobius.
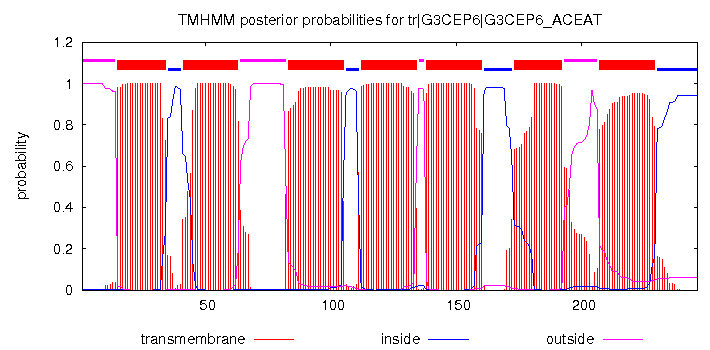
# tr|G3CEP6|G3CEP6_ACEAT Length: 246 # tr|G3CEP6|G3CEP6_ACEAT Number of predicted TMHs: 7 # tr|G3CEP6|G3CEP6_ACEAT Exp number of AAs in TMHs: 152.87846 # tr|G3CEP6|G3CEP6_ACEAT Exp number, first 60 AAs: 37.68017 # tr|G3CEP6|G3CEP6_ACEAT Total prob of N-in: 0.00165 # tr|G3CEP6|G3CEP6_ACEAT POSSIBLE N-term signal sequence tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 outside 1 14 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 15 34 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 inside 35 40 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 41 63 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 outside 64 82 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 83 105 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 inside 106 111 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 112 134 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 outside 135 137 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 138 160 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 inside 161 172 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 173 192 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 outside 193 206 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 TMhelix 207 229 tr|G3CEP6|G3CEP6_ACEAT TMHMM2.0 inside 230 246
Текстовая выдача TMHMM.
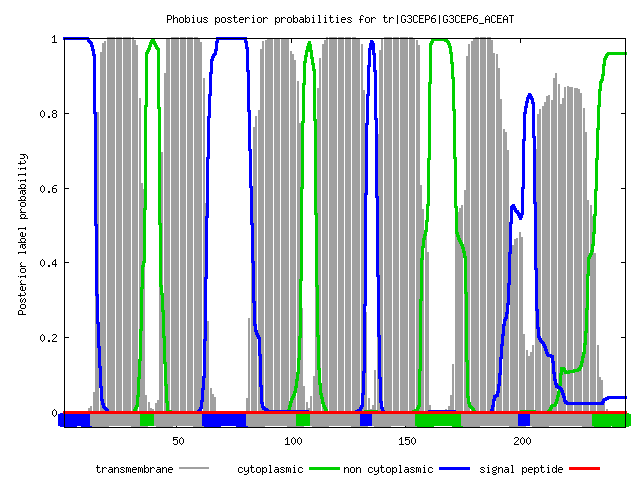
ID tr|G3CEP6|G3CEP6_ACEAT FT TOPO_DOM 1 14 NON CYTOPLASMIC. FT TRANSMEM 15 36 FT TOPO_DOM 37 42 CYTOPLASMIC. FT TRANSMEM 43 63 FT TOPO_DOM 64 82 NON CYTOPLASMIC. FT TRANSMEM 83 104 FT TOPO_DOM 105 110 CYTOPLASMIC. FT TRANSMEM 111 132 FT TOPO_DOM 133 137 NON CYTOPLASMIC. FT TRANSMEM 138 156 FT TOPO_DOM 157 176 CYTOPLASMIC. FT TRANSMEM 177 201 FT TOPO_DOM 202 206 NON CYTOPLASMIC. FT TRANSMEM 207 233 FT TOPO_DOM 234 246 CYTOPLASMIC. //
Текстовая выдача Phobius.
База данных TCDB
В базе TCDB был обнаружен белок 4ctd. Структура белка, как уже было описано в Таблице 1 представляет из себя бета-бочонок, сформированный 14ю бета-тяжами. Ген, кодирующий данный белок располагается в генном кластере, содержащем гены ABC транспортера глюкозы, гликозил гидролазы и двух оксидоредуктаз. На основании этого можно предположить, что рассматриваемый белок связан с поглощением клеткой глюкозы. При высоких или нейтральных значениях pH 4ctd открыт и является проницаемым для частиц размером до 900 Da. При низких значениях pH канал закрывается. Предположительно, блокировка происходит в следствие протонирования пары гистидинов на соседных бета-тяжах и последующего их отталкивания, что и вызывает конформационные изменения (закрытие поры).
TC# исследуемого белка 1.B.21.1.1 означает следующее: 1 – принадлежность белка к каналам, 1.B – бета-бочонковые каналы, 1.В.21 – OmpG каналы E.coli, зависимые от pH.