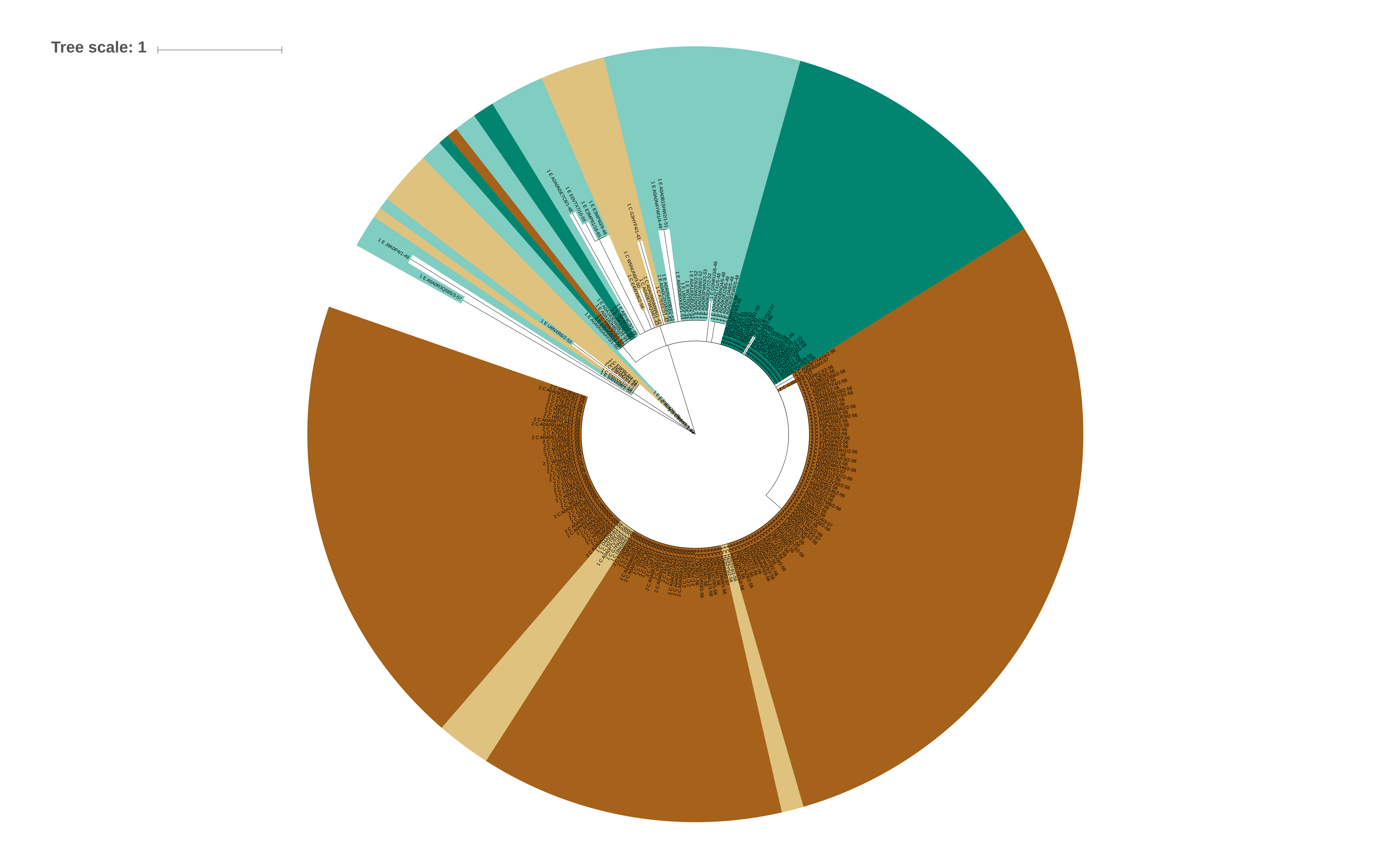Реконструкция эволюции доменной архитектуры
Выбор семейства доменов
Для работы был выбран домен Tudor-knot (PF11717), участвующий в связывании РНК.
Посмотреть список архитектур можно ЗДЕСЬ. Для дальнейшей работы были выбраны 2 архитектуры:
U6NXR6 (230 последовательностей) и W5K820(195 последовательностей). Архитектура U6NXR6 имеет в своём составе только Tudor-knot домен. Вторая архитектура W5K820
несёт в своём составе помимо Tudor-knot домена, домены ARID и RBB1NT. ARID связывается с ДНК, а функции RBB1NT неизвестны, но он часто обнаруживается в ДНК-связывающих белках, содержащих ARID домен.
Выравнивание доменов этого семейства было скачено с Pfam и визуализировано с помощью программы JalView. Выравнивание раскрашено ClustalX, порог консервативности - 11.
[Скачать исходный jvp проект]
Выбор семейства доменов, таксонов и представителей архитектур
C помощью python скриптов и средств Excel для архитектур U6NXR6 и W5K820 были выбраны представители таксона Metazoa и подтаксонов Chordata и Ecdysozoa. Список выбранных последовательностей представлен на листах Chosen1 и Chosen2 excel файла.
[Скачать xlsx файл]
[Скачать отфильтрованное выравнивание]
Филогенетическое дерево выбранных последовательностей.
Для большей наглядности к именам последовательностей были добавлены цифры и буквы, обозначающие их принадлежность к определенному таксону и архитектуре. Буква E в названии обозначает Ecdysozoa, а C – Chordata. Цифра 1 – первая доменная архитектура U6NXR6, 2 – вторая, W5K820. Дерево было построено методом максимального правдоподобия в MEGA7 и визуализировано с помощью ITOL (Рис. 1). Судя по укоренению дерева, можно предположить, что изначально возникла архитектура 1, причем в общем предке С и E (так как и в той, и в той архитектуре есть "пришельцы" из других подтаксонов).
[Скачать скобочную форму дерева]