HMM профили
Получение профиля
Для работы было выбрано семейство Enolase_C (PF00113) из 6 практикума, при этом отбирались белки, принадлежащие протеобактериям. Запрос в Uniprot: database:(type:pfam id:PF00113) taxonomy:"Proteobacteria [1224]" AND reviewed:yes. Выдача Uniprot была сохранена в excel файл для последующей работы. Далее с помощью Jalview из pfam было скачано выравнивание всех белков, содержащих рассматриваемый домен, затем скачанное выравнивание было отфильтровано с помощью скриптов из практикума 7. Полученное выравнивание использовалось для дальнейшего построения профиля домена.
[скачать excel файл]
[скачать выравнивание, использованное для получения профиля]
[скачать профиль]
Проверка профиля
Поиск проводился по SwissProt, с E-value 1000 и порогом веса -50, что было необходимо для построения roc кривой и подбора порога score. Результаты поиска сохранены на отдельном листе excel файла. На рисунке 1 приведена гистограмма весов находок. Далее, используя описание из презентаций, была построена roc кривая (Рис. 2). По полученным данным был подобран порог score 710 (было выбрано максимальное значение Sensitivity + Specificity - 1). В целом, построенный профиль при выбранном пороге показывает неплохие результаты (Таблица 1).
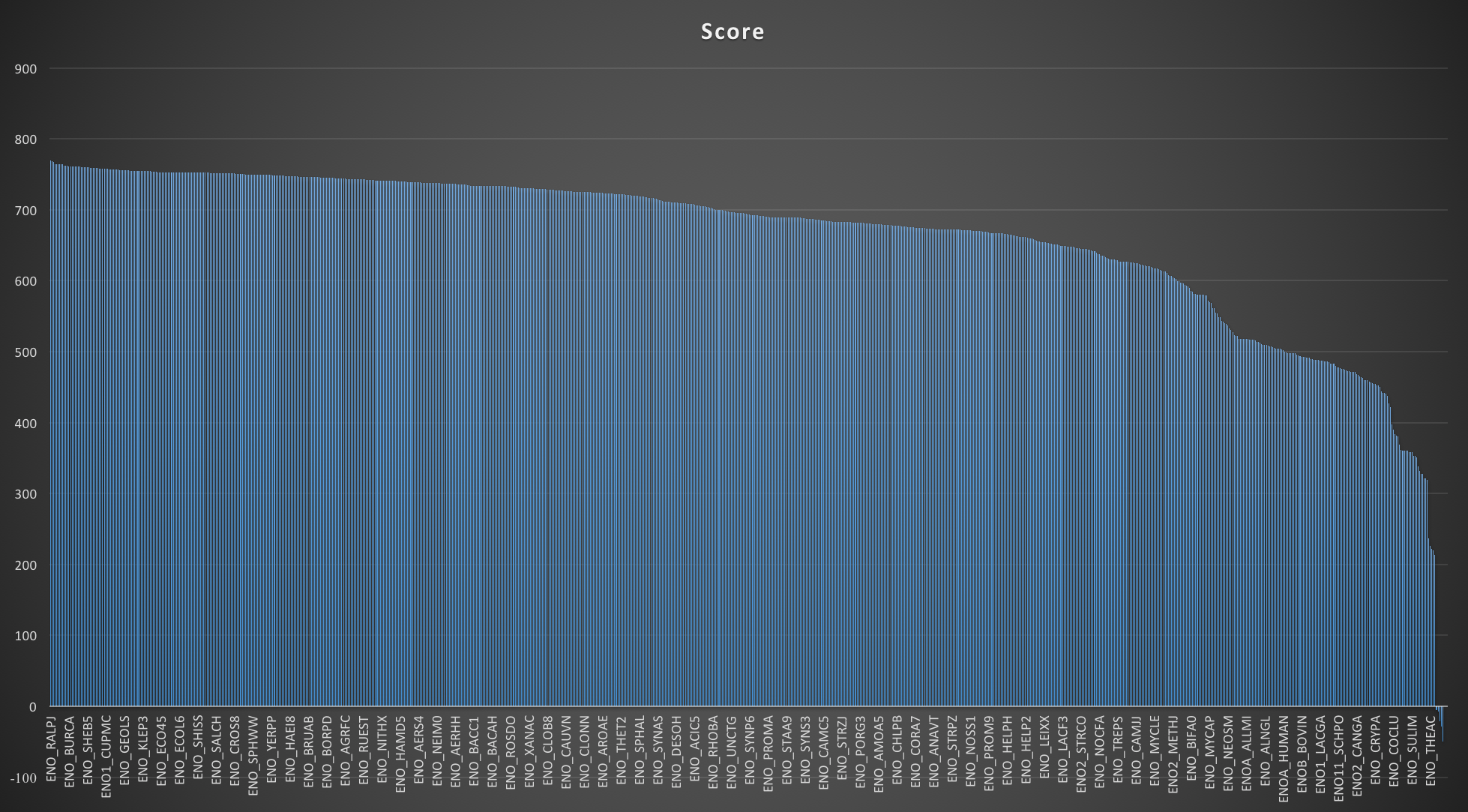
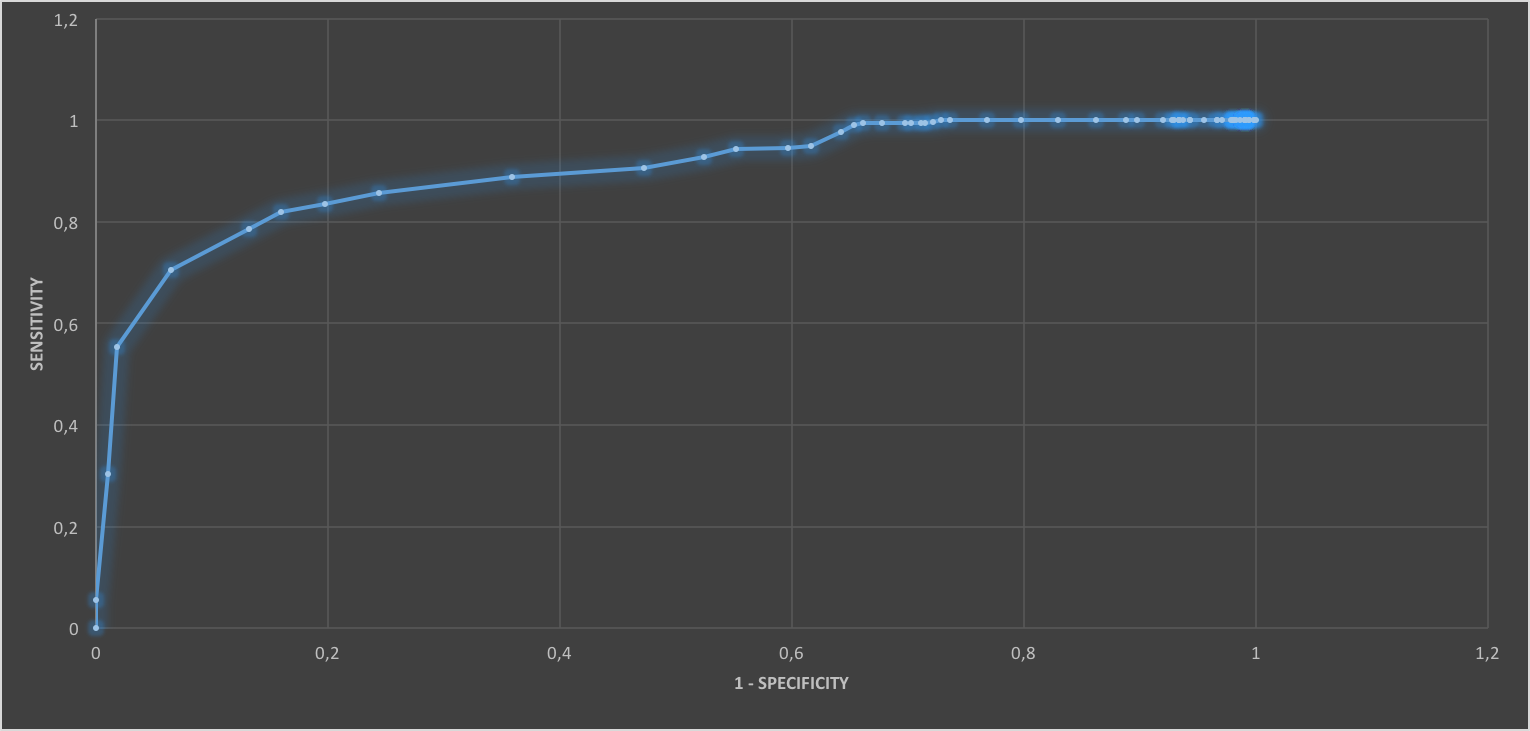
| TP | 329 |
|---|---|
| TN | 428 |
| FP | 81 |
| FN | 72 |
| Sensitivity | 0,82 |
| Specificity | 0,84 |
| Positive predictive value | 0,8 |
| Negative predictive value | 0,86 |
| Accuracy | 0,83 |
Таблица 1. Результаты предсказания при выбранном пороге.