cat *.fa>adapt.fastaАдаптеры были удалены с помощью команды:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240380.fastq trimm.fastq ILLUMINACLIP:adapt.fasta:2:7:7
| До чистки | После чистки | |
|---|---|---|
| Размер файла | 525M | 515M |
| Число чтений | 5217318 | 5119139 |
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 trimm.fastq trimm_cut.fastq TRAILING:20 MINLEN:30
| До чистки | После чистки | |
|---|---|---|
| Размер файла | 515 M | 490 M |
| Число чтений | 5119139 | 4879707 |
velveth out 29 -fastq -short trimm_cut.fastqКонтиги были собраны командой:
velvetg out/
| K-mers 29 | K-mers 25 | |
|---|---|---|
| N50 | 18128 | 5601 |
| Длина | 669685 | 708017 |
| Число контигов | 884 | 3676 |
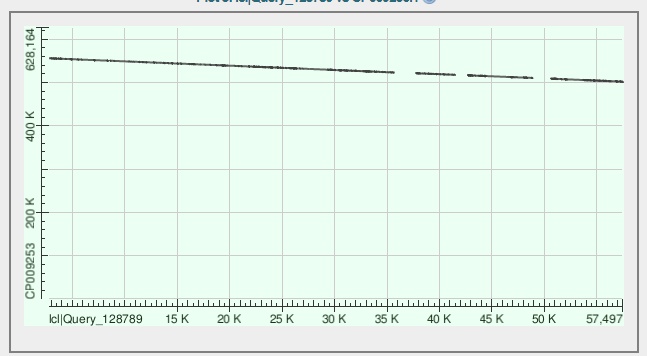
| Самые длинные контиги | 1)ID 9, lgth 57469, cov 35.8205822)ID 3, lgth 43960, cov 36.2742273)ID 8, lgth 33034, cov 35.383968 | 1)ID 11,lgth 23320, cov 51.5190392)ID 69,lgth 19422, cov 53.8030073)ID 41,lgth 15503, cov 47.728569 |

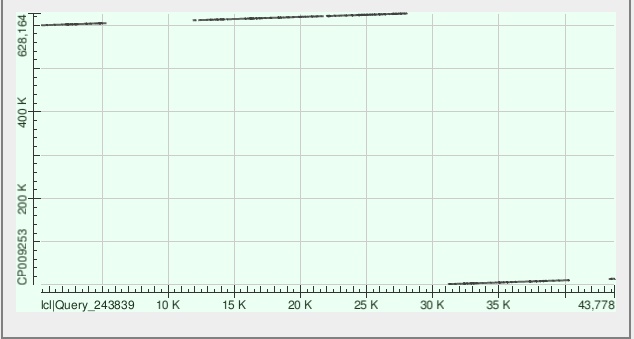
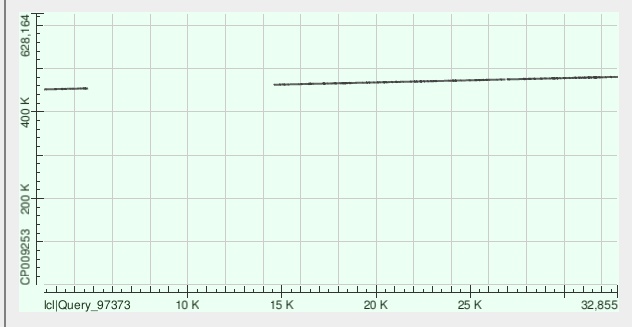
| Контиг | mismatches | gaps | chromosome | query coverage | identity |
|---|---|---|---|---|---|
| 9 | 4026 | 545 | 528794-550219 | 86% | 81% |
| 3 | 1992 | 252 | 2063-11103 | 68% | 78% |
| 8 | 1697 | 208 | 467471-474667 | 61% | 77% |
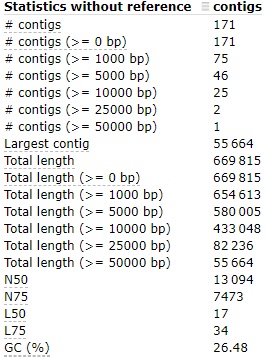
spades.py -s trimm_cut.fastq -k 29 -o spaДля анализа выдачи была использована программа quast: