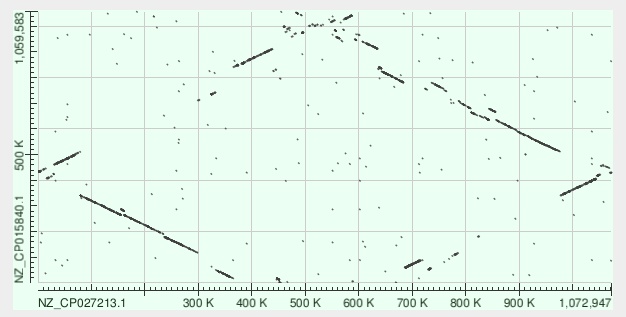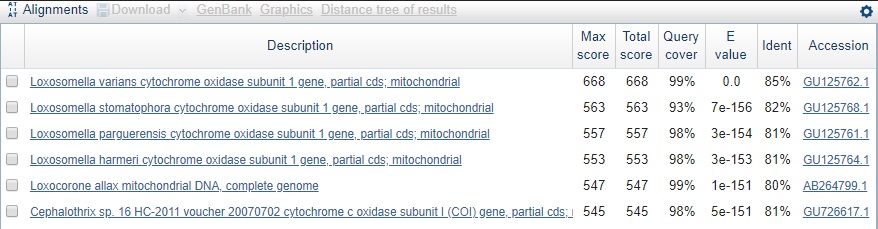
>EMBOSS_001
gnacngctttaagtatattaattcgaattgaattaagacaaccaggagctttgttaggag
atgatcaactatatnacgtaattgtaacggctcanGCTTTTGTTATAATTTTTTTTTTAG
TTATACCTGTTTTAATTGGAGGATTTGGTAATTGattATTAGTACCTTTAATATTAGGGG
CTCCCGATATAGCTTTCCCCCGAATAAATAACATAAGTTTTTGGTTATTACCACCTTCTT
TAACTCTTCTGTTAARAAKTTCTTTAGTGGAAATAGGGGCAGGAACKGGGTGAACAGTAT
ACCCTCCACTATCTAGTAATTTAGCCCATTCAGGAGGGTCTGTAGATTTAGCTATTTTTT
CATTACATTTAGCTGGKGTTTCTTCTATTTTAGGKGCTATTAATTTTATTACTACTACAA
TTAATATGCGGTGATATGGATATCAATTTGAANATATCCCATTATTTGNGNGGNCNGTAA
AATTAACAGCTANTTTATTATTGCTATCTCTACCTGTTTTAGCTGGGGCTATTACAATAT
TATTAACARATCKTAATTTTAAWACGTCATTTTTTGATccttg
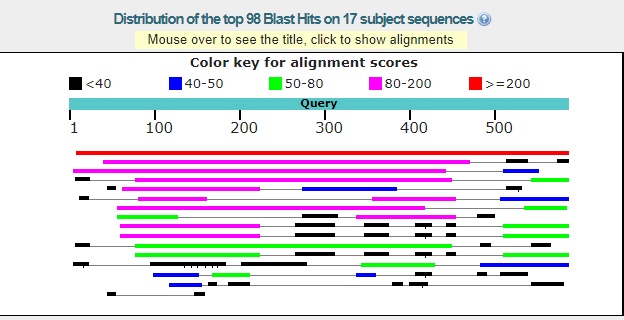
Ниже приводится 6 лучших находок blastn

| Алгоритм | Параметры алгоритма | Число находок |
|---|---|---|
| megablast | Стандартные | 1 |
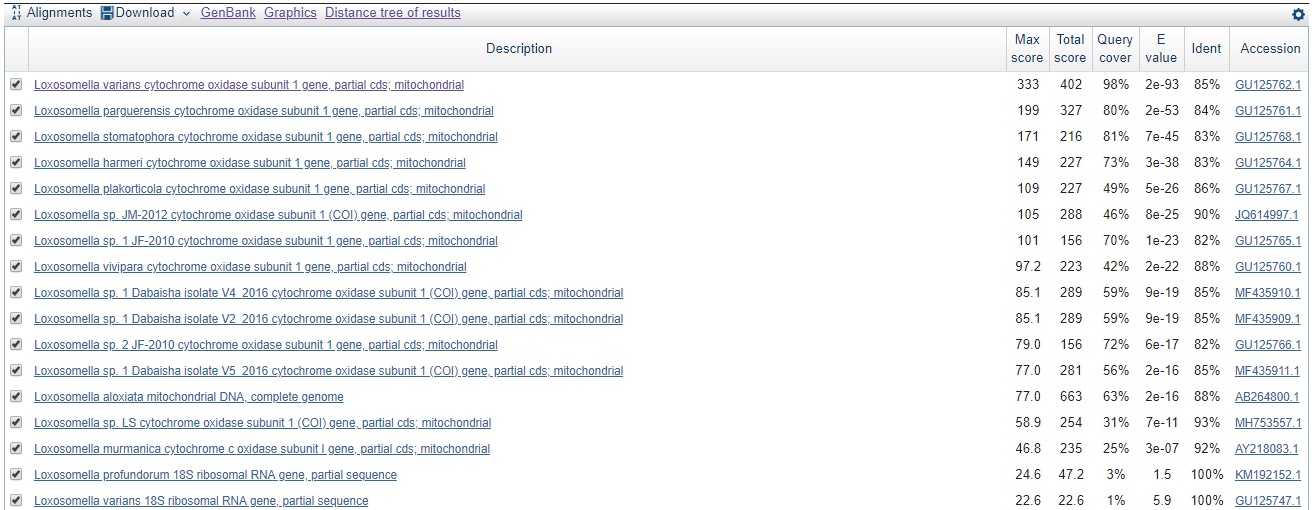
| blastn | Стандартные | 16 |
| balstn | Длина слова =7; M/M 1;-4 | 17 |


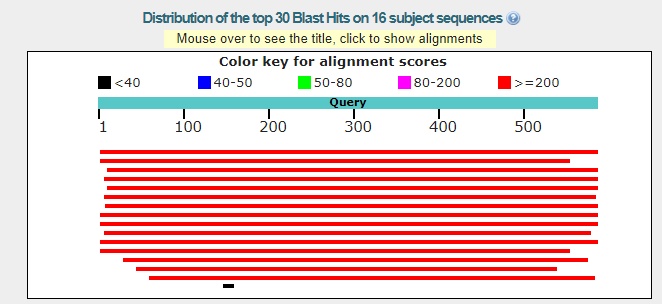
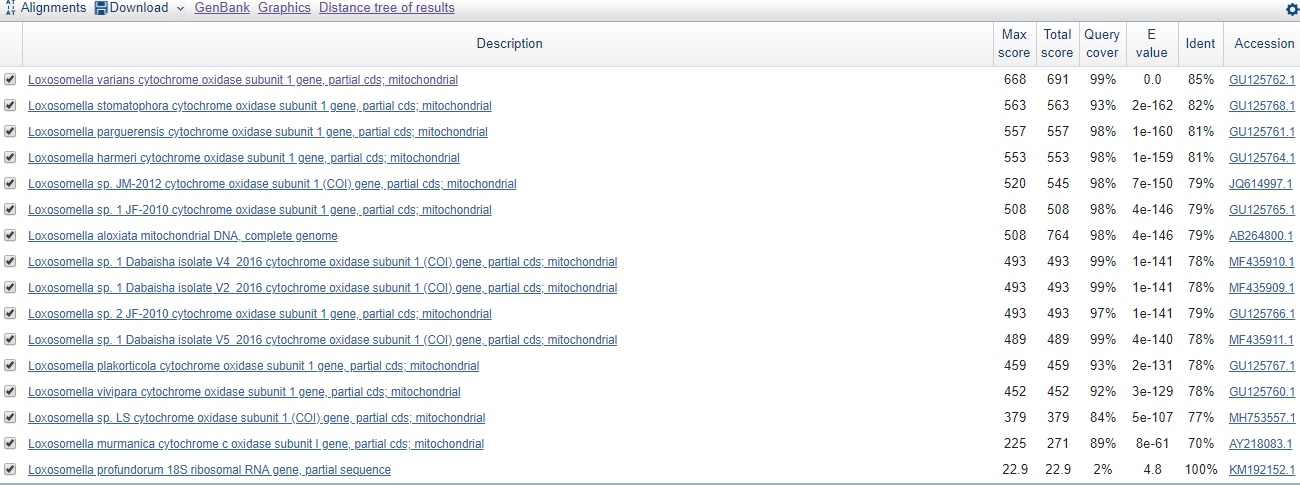
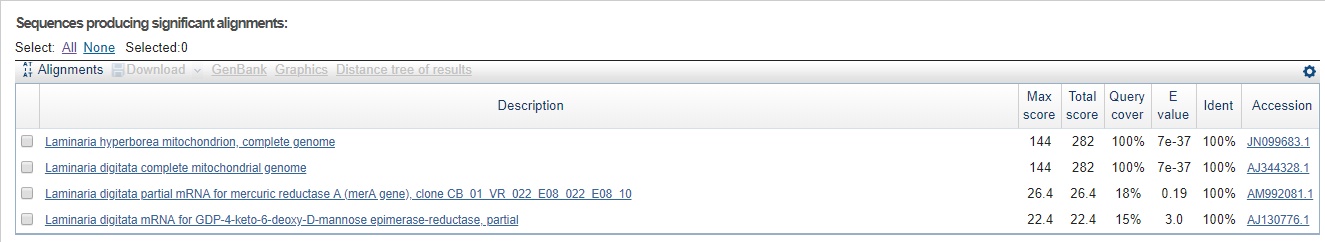
>AJ344328.1 AJ344328 Laminaria digitata complete mitochondrial genome GTTTGGGTAGCTCAGTTGGTAGAGTGAAGGACTGAAAATCCTTAGGTCGTCGGTTCAAGC CCGATCCCAAAC
| Алгоритм | Параметры алгоритма | Число находок |
|---|---|---|
| megablast | Стандартные | 1 |
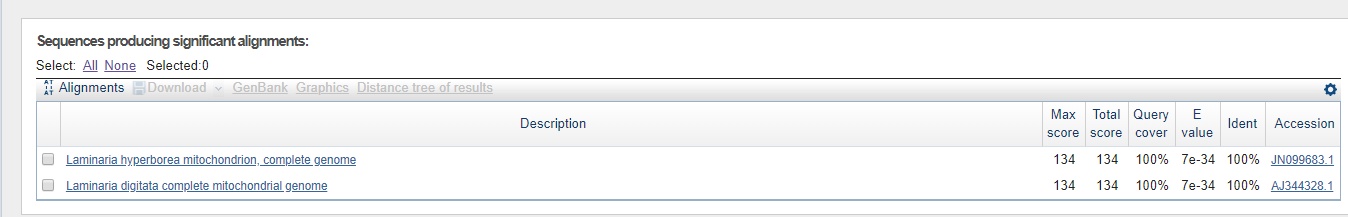
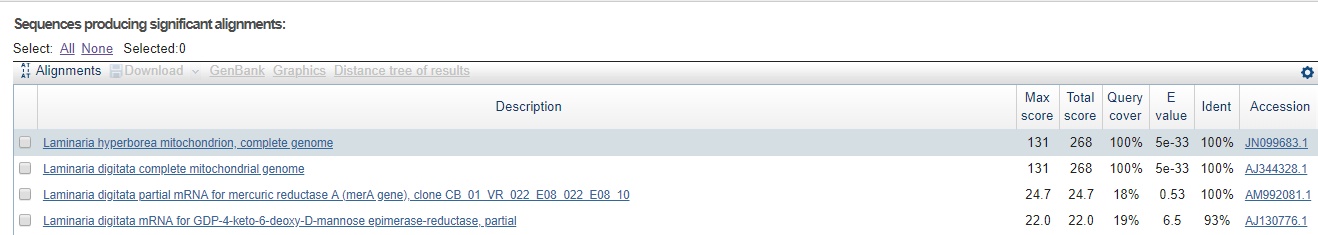
| blastn | Стандартные | 4 |
| balstn | Длина слова =7;M/M 1;-4 | 4 |



makeblastdb -in X5.fasta -dbtype nuclДалее в нем с помощью BLAST+ производился поиск гомологов белоков HSP71_YEAST; TERT_SCHPO; PRPC_EMENI Пример команды запуска:
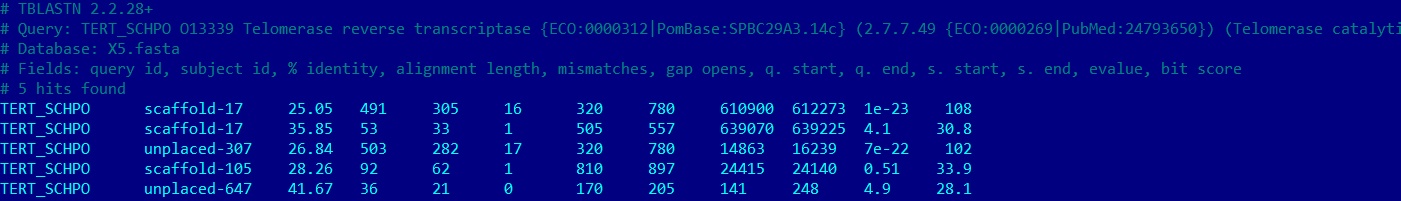
tblastn -query tert_schpo.fasta -db X5.fasta -outfmt 7 > tert_schpo.outПример выдачи:
| HSP71_YEAST | TERT_SCHPO | PRPC_EMENI | |
|---|---|---|---|
| Лучшее совпадение | "scaffold-199 Score:920, E-value 0.0 | "scaffold-17 Score;108, E-value 1e-23 | "scaffold-693 Score:393 E-value: 6e-121 |
| Вывод о гомологии | Много участков(22) с высокими процентами идентичности и E-value. Находка положительная | Есть один скэффолд и один контиг с невысоким уровнем идентичности, но достаточно хороший E-value позволяет предположить гомологичность отдельных доменов белка. | Найдено всего 6 совпадений, причем только в двух из них довольно хороший E-value. В данном случае можно говорить о том, что находка условно положительная и можно предположить родственность данных белков |