Выравнавание белковых последовательностей и создание матрицы аминокислотных замен
Работа с программами FAR maneger, Excel, GeneDoc
1) Создание выравниваний
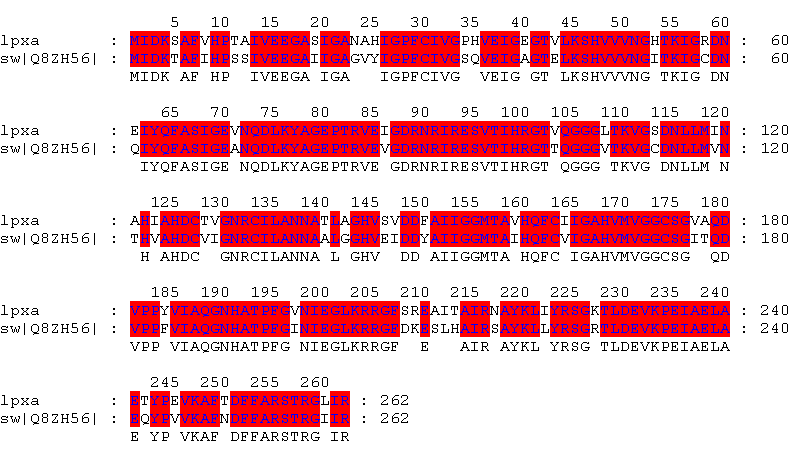
В чисто педагогических целях наши преподаватели предложили нам создать в ручную выравнивания предложенных аминокислотных последовательностей. Выравнивание производилось в FARE (по этому и смотрится через Explorer столь криво).>Seq1 _______ATRYSSLIILDFRFFGLAWQELLPYHGGDFFWNVCAWILKNPAQDW-PAA
>Seq3 METQQATRYSSVAILDFR-------------LLPYHGGDFFWQVCAWILKNPAQEYEP
Далее производился подсчёт весового коэффициента выравнивания. Указательным пальцем правой руки были подсчитаны совпадающие аминокислотные остатки и гепы. После чего был произведёно мощнейшее вычисление 33-9=24.
Далее дело пошло веселей.
Для поднытия рабочего духа было предложено также в ручную произвести в GeneDoc выравнивания нескольких белков из выборки описанной ранее, а именно P10440,Q8ZH56,Q9KPW4 .

Создание матриц
На последнем этапе наших интереснейших и информативнейших занятий нам предстояло составить матрицы аминокислотных замен. Весь процесс состоял из следующих этапов:
1)Производится подсчёт вышеупамянутым пальцем аминокислотных замен и совпадений для обоих выравниваний(сильно развивает внимание на досуге). При таком способе подсчёта практически не может возникнуть никаких ошибок, по этому результаты приведённые ниже крайне надёжны.
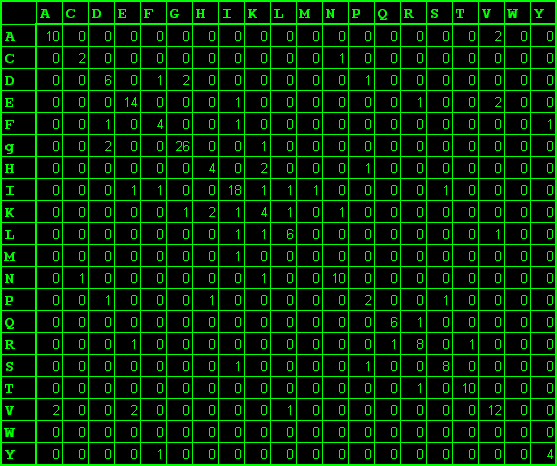

2)Далее в Excele полученые матрицы были сложены и нормированы.В результате чего была получена приведенная ниже МЕГАМАТРИЦА.

Задание было столь сложным и трудоёмким, что по нему получили зачёт только 6 человек из группы, но в то же время очень интересным и увлекательным, учащим нас аналитически мыслить и мыслически анализировать. Я убеждён, что этот опыт несомненно пригодится мне в дальнейшей жизне.